מיפוי אתר הקישור של Aptamer על ATP שימוש microscale Thermophoresis
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
אינטראקציה בין המולקולות היא הבסיס של הטבע. לפיכך, מדענים בתחומים רבים של מחקר בסיסי ויישומים לנסות להבין את עקרונות היסוד של אינטראקציות מולקולריות מסוגים שונים. Microscale Thermophoresis (MST) מאפשרת למדענים לבצע את מהירה מדויקת, יעילה וחסכונית, ואיכות שבשליטת אפיון של אינטראקציות מולקולריות בתמיסה, עם בחירה חופשית של מאגרים. יש כבר יותר מ -1,000 פרסומים באמצעות MST, משנת 2016 בלבד, המתאר סוגים שונים של ניתוחים, כוללים סריקות ספרייה, מחייב אימותי אירוע, מבחני תחרות, וניסויים עם שותפים מחייבים מספר 1-8. באופן כללי, MST מתיר חקר הפרמטרים המחייבים הקלסיים, כגון זיקה מחייבת (PM כדי מ"מ), ורכב, ותרמודינאמיקה, מכל סוג של אינטראקציה מולקולרית. יתרון גדול של MST הוא היכולת ללמוד אירועים מחייבים תלות בגודל של שותפי האינטראקציה. אפילו chalאינטראקציות lenging בין aptamers חומצות גרעין קטן (15-30 NT) ויעדים כגון מולקולות קטנות, תרופות, אנטיביוטיקה, או מטבוליטים ניתן לכמת.
נוכח המדינה- of-the-art טכנולוגיות לאפיין אינטראקציות aptamer-היעד הם או מעבדה קשות ומורכבות מאוד או להיכשל לכמת aptamer-מולקולה קטנה קשרי גומלין 9,10. Surface Plasmon תהודה (SPR) מבוססי מבחני 11,12 וגישות קלוריות ללא תווית באמת, כגון Calorimetry טיטרציה איזו תרמים (ITC) 13-15, elution isocratic 16, ltration fi שיווי משקל 17,18, ב-קו חיטוט 19, ג'ל משמרת מבחנים, והשתתק fl זרימת uorescence ספקטרוסקופיה 20,21, אנאיזוטרופיה קרינה (FA) 22,23, uorescence ההדמיה fl מולקולה בודדת 24,25, ו interferometry ביו השכבתי (BLI) 26 גם אם לא מדויקים או לא תואמים עם מולקולת aptamer-קטן יחסי גומלין. principa אחרסוגיות l של שיטות אלו רגישות נמוכה, צריכת דגימה גבוהה, חוסר תנועה, מגבלות להסעת המונים על משטחים, ו / או הגבלות חיץ. רק מעטי טכנולוגיות אלה מספקים בקרות משולבות אפקטי צבירת ספיחה.
MST מהווה כלי רב עוצמה עבור המדענים להתגבר על מגבלה זו כדי לחקור את יחסי הגומלין בין aptamers ומולקולות קטנות 27-29, כמו גם מטרות אחרות כגון חלבונים 30-33. הטכנולוגיה מתבססת על תנועת מולקולות באמצעות הדרגתיים טמפרטורה. התנועה מכוונת זו, המכונה "thermophoresis," תלוי בגודל, פריצה, ופצצת הידרציה של המולקולה 34,35. עקידת ליגנד למולקולה תשנה במישרין לפחות אחד הפרמטרים האלה, וכתוצאה מכך ניידות thermophoretic השתנה. הליגנדים עם מידות קטנות לא שיכולים להשפיע בצורה מהותית מבחינת שינוי הגודל מתוך המאוגד אל המדינה הנכנסת, אבל הם יכולים להיות dr תופעות amatic על הקליפה הידרציה ו / או תשלום. השינויים בתנועה thermophoretic של מולקולות לאחר אינטראקציות עם שותף מחייב מאפשרת כימות של פרמטרים מחייבים בסיסיים 2,7,34,36,37.
כפי שמתואר באיור 1A, מכשיר MST מורכב ליזר אינפרא אדום הממוקד על המדגם בתוך נימי הזכוכית באמצעות אותו אופטיקה ובאשר גילוי קרינה. התנועה thermophoretic של חלבונים באמצעות uorescence fl הפנימי של tryptophans 6 או של 3,8 שותף אינטראקציה שכותרתו fluorescently ניתן לנטר בזמן הלייזר קובע שיפוע הטמפרטורה (ΔT של 2-6 מעלות צלזיוס). הבדל הטמפרטורה והתוצאה הוא יותר שטח, ΔT, מוביל הדלדול או ההצטברות של מולקולות בתחום הטמפרטורה גבוהה, אשר ניתן לכמת ידי Soret COEF fi יעיל (S T):
ז "/>
ג חם מייצג את הריכוז באזור המחומם, וקרת c היא הריכוז באזור הקר הראשוני.
כפי שניתן לראות בתרשים 1B, תוצאות ניסוי MST טיפוסיות פרופיל תנועת MST (זכר זמן), מורכב שלבים שונים, אשר יכול להיות מופרד על ידי לוחות הזמנים שלהם. הקרינה הראשונית נמדדה ב -5 s הראשונה בהיעדרו של שיפוע הטמפרטורה כדי להגדיר את הקרינה החל מדויקת כדי לבדוק photobleaching או photoenhancement. קפיצת הטמפרטורה (T-Jump) מייצגת את השלב שבו שינויי הקרינה לפני תנועת thermophoretic. ירידה ראשונית זו קרינה תלויה בשינויים חומים תלויה של fl uorophore תשואת קוונטים. שלב thermophoresis כדלקמן, שבו ירידות הקרינה (או עליות) עקב תנועת thermophoretic של המולקולות עד החלוקה היציבה הוא הגיע.TJump הפוכה דיפוזיה בחזרה קשורה של מולקולות uorescent fl ניתן לצפות כמצוין באיור 1B לאחר הלייזר מכובה. כדי לגשת פרמטרים מחייבים בסיסיים, יחסי טוחנת שונים של השותפים באינטראקציה מנותחים ומשווים. בדרך כלל, 16 יחסים שונים נלמדים בניסוי אחד MST, ואילו מולקולת הגלוי האופטית נשמרה קבועה מסופקת עם כמות הולכת וגדלה של ליגנד ללא תווית. האינטראקציה בין שני השותפים מחייבים גורמת לשינויי thermophoresis, ומכאן גם uorescence fl המנורמל, נורמת F, אשר מחושבת כדלקמן:
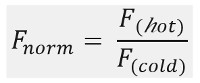
F חם וקר F מייצג ממוצעי עוצמות fl uorescence ב דה fi מועד לפי שעון נד של עקבות MST. זיקות מחייבות (ד K או EC 50 ערכים) יכולות להיות מחושבות על ידי הכמתמ"כהולם דואר (תרשים 1C).
בסך הכל, MST הוא כלי רב עוצמה כדי לחקור אינטראקציות מולקולריות מכל סוג שהוא. כתב יד זה מציע פרוטוקול לאפיין את האינטראקציה המאתגרת בין אדנוזין אדנוזין המולקולה הקטן (ATP; 0.5 KDA) ואת 25-NT הקצר ssDNA aptamer DH25.42 (7.9 KDA). במהלך של כתב היד, אתר הקישור של aptamer על מולקולת ATP ממופה אל קבוצת אדנין של ATP.
Protocol
Representative Results
Discussion
בקרות איכות:
דבק נוקבים / ספיחה של חומר מדגם למשטחים, וכן תופעות צבירה, יש השפעה דרמטית על איכות נתוני הזיקה. עם זאת, רק המדינה- of-the-art כמה טכנולוגיות מציעות אפשרויות מדויקות ומהירות לפקח למנוע את ההשפעות הללו. MST מציעה בקרת איכות משול…
Disclosures
The authors have nothing to disclose.
Acknowledgements
יש המחברים לא בתודות.
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. 생화학. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. 생화학. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. 생화학. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. 생화학. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

