ממשק משתמש גרפי למעקב בסיוע תוכנה של ריכוז חלבון בהפרעות ניידות דינמיות
Summary
אנו מציגים פתרון תוכנה למעקב חצי אוטומטי של ריכוז חלבון יחסי לאורך של בליטות תאיות דינמיות.
Abstract
פילופודיה הן בליטות תאיות דינמיות, דמויי אצבע, הקשורות בהעברה ובתקשורת תאית. על מנת להבין טוב יותר את מנגנוני האיתות המורכבים העומדים בבסיס החניכה הגופנית, הארכה וייצוב או נסיגה שלאחר מכן, חשוב לקבוע את פעילות החלבון הזמני-הטמפורלי במבנים דינמיים אלה. כדי לנתח את תפקוד החלבון ב filopodia, פיתחנו לאחרונה אלגוריתם חצי אוטומטי מעקב שמתאים לשינויים צורה filopodial, ובכך מאפשר ניתוח מקבילי של הדינמיקה בליטה וריכוז חלבון יחסית לאורך כל אורך filopodial. כאן, אנו מציגים מפורט שלב אחר שלב פרוטוקול עבור טיפול התא אופטימיזציה, רכישת התמונה וניתוח תוכנה. בנוסף, אנו מספקים הנחיות לשימוש בתכונות אופציונליות במהלך ניתוח תמונות וייצוג נתונים, וכן הנחיות לפתרון בעיות עבור כל השלבים הקריטיים לאורך הדרך. לבסוף, אנו כוללים גם השוואה של דEscribed תוכנת ניתוח תמונה עם תוכניות אחרות הזמינות לכימות filopodia. יחד, הפרוטוקול המוצג מספק מסגרת לניתוח מדויק של הדינמיקה חלבון ב בליטות filopodial באמצעות תוכנת ניתוח התמונה.
Introduction
שליטה ספציו-טמפורלית של חלבונים רגולטוריים אקטין קשורה דינמיקה פילופודיום 1 , 2 . מעקב ריכוז חלבונים בריכוז מרחבי לאורך כל אורך filopodial דרך הזמן ולכן חיוני כדי לקדם את הבנתנו את המנגנונים ביסוד היזום, הארכה, ייצוב או קריסה של מבנים דינמיים אלה 3 , 4 . שלא כמו ניתוח חלבון cytosol, שבו שינויים רבים צורה התא מתרחשים בקנה מידה גדול יותר, filopodia הם מבנים דינמיים מיקרו אשר כל הזמן אבזם 5 וכופף, ובכך מונע ניתוח באמצעות גישה פשוטה כגון קו סריקה.
פתרונות תוכנה שונים למעקב אחר הצורה filopodial זמינים 6 , 7 , 8 , 9 . כמו כןIse, תוכנה למעקב ratiometric של הדינמיקה חלבון בתוך הגוף התא פותחה 10 , 11 . כדי לשלב מעקב אוטומטי של הצורה filopodial וניתוח חלבון זמן ספטיו, פיתחנו לאחרונה ניתוח תמונה תוכנה המבוססת על קמור גוף האלגוריתם 12 . שיטת ניתוח חדשנית זו, המופעלת באמצעות ממשק משתמש גרפי (GUI), משלבת לראשונה את ריכוז החלבון היחסי לאורך אורך הגדילה ומהירות הגדילה, ומאפשרת מדידה מדויקת של התפלגות חלבונים מזמניים-טמפורליים ללא תלות בתנועה של אלה מבנים דינמיים 12 .
הרעיון מאחורי התוכנה (קוד המקור זמין באופן חופשי, ראה להלן) היא כי אחד הקודקודים של גוף קמור יעלה בקנה אחד עם קצה של פילופודיום ( איור 1 א ). על ידי הסתכלות על מסגרת הבאהR את הקודקוד הקרוב ביותר של גוף קמור, עצה מרגש ניתן לעקוב לאורך כל הסרט. לאחר קצה מזוהה בכל מסגרת, המיקום שלה משמש לצייר ציר על ידי הצטרפות קצה עם נקודת התייחסות בבסיס של פילופודיום ( איור 1 ב ). לבסוף, תוך שימוש בנקודות ראשיות שווה, שתפקידן נקבע על ידי הפיקסל החציוני בעוצמה מקסימלית לאורך הקו האורתוגונלי לציר, משמשות לקביעת עמוד שדרה העוקב אחר הצורה הפילופודיאלית. ניצול של עמוד שדרה זה מסתגל, kymograph מופק כדי לעקוב אחר הצמיחה filopodial ריכוז חלבון עד שלושה ערוצים לאורך אורך filopodial ( איור 1C ).
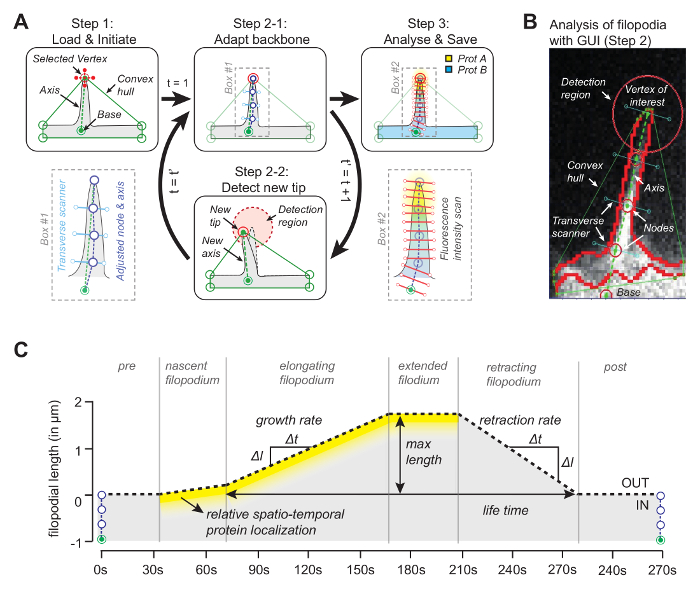
איור 1: עקרון העבודה של תוכנת ניתוח תמונה. ( א ) האלגוריתם שמאחוריהתוכנה. ב Step1 המשתמש מציין את הפניה (בסיס) ואת קודקוד (קצה) של פילופודיום. בשלב 2-1 את עמוד השדרה של פילופודיום מתקבל באמצעות פיקסל חציון עם ערך אינטנסיביות מקסימלית. בשלב 3 את עמוד השדרה משמש פרופיל חלבון מרחבי פרופיל. בשלב 2-2 התוכנה עוקבת באופן אוטומטי את קצה במסגרת הבאה. כל התהליך חוזר. ( ב ) תצלום של אלגוריתם עם פילופודיום אמיתי מציגה אלמנטים חשובים כגון גוף קמורה כי הוא משמש למעקב. ( ג ) סקירה של פרמטרים שניתן למדוד עם האלגוריתם. נתון זה שונה מהתייחסות 12 . אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
תוכנת ניתוח התמונה מופעלת ב Matlab (המכונה תוכנה תכנות) באמצעות שימוש גרפיממשק. כדי למקסם את הגמישות והחוסן להגדרה הניסויית, המשתמש יכול להתאים סדרה של פרמטרים למעקב ( למשל זווית כיפוף מותרת ותנועה בין-מסגרתית) וכן לבצע כמה תיקונים לסרטים ( למשל חיתוך, סיבוב, הסרת אובייקטים לא רצויים) ( איור 2 א וטבלה 1) .
| GUI | לא. | חובה | תאור | שם (ב- GUI) | ||||
| # 1 | 1 א | Y | טעינת קובץ .tiff מוערמים המייצגים את גוף התא (עם תיבת מסומנת) או ליצור גוף תא superimposed מערוצים | סלבודי | ||||
| # 1 | 1b | Y | <td colspan="5"> טוען קובץ תמונה מוערם המתאים לחלבון 1חלבון 1 | |||||
| # 1 | 1 ג | Y | טעינת קובץ תמונה מוערם המתאים לחלבון 2 | חלבון 2 | ||||
| # 1 | 1 ד | Y | טעינת קובץ תמונה מוערם המתאים לחלבון 3 | חלבון 3 | ||||
| # 1 | 1e | N | איפוס הכל לטעינת קובצי תמונות מוערמים מראש | אִתחוּל | ||||
| # 1 | 2 א | Y | גלילה בר כדי לקבוע את המסגרת הראשונית לניתוח בחלון GUI # 2 | NA | ||||
| # 1 | 2 ב | Y | גלילה בר כדי לקבוע את המסגרת הסופית לניתוח בחלון GUI # 2 | NA | ||||
| # 1 | 2 ג | Y | סרגל גלילה המייצגמסגרת nt | NA | ||||
| # 1 | 2 ד | N | הערך האפור של הפיקסלים שמתחתיו כל הפיקסלים יוגדרו לאפס | NA | ||||
| # 1 | 2e | N | הערך האפור של הפיקסלים שמעליהם כל הפיקסלים יוגדרו לערכים מקסימליים | NA | ||||
| # 1 | 2 ו | N | הגדר את ערכי העוצמה של הפיקסלים שצוינו על ידי <2e> <2f> | מַעֲרֶכֶת | ||||
| # 1 | 2 גרם | N | הפעל את הסרט מותאם-עוצמה | לְשַׂחֵק | ||||
| # 1 | 2h | N | חתוך תמונה | יְבוּל | ||||
| # 1 | 2i | N | סובב תמונה | לְסוֹבֵב | ||||
| # 1 | 2 | N | מחק אזורים בכל הערימה | מחק אזורים | ||||
| # 1 | 3 א | Y | לחץ כדי לפתוח את 'חלון ניתוח' (חלון GUI # 2) | חלון מעקב | ||||
| # 1 | 3 ב | Y | הזן את הגודל של פיקסל במיקרונים | הזן גודל פיקסל | ||||
| # 2 | 4 | Y | לחץ כדי ליצור את התמונה גבול / קצה של גוף התא על גבי | גְבוּל | ||||
| # 2 | 5 | Y | לחץ כדי לבחור את הבסיס ואת קצה filopodia | הפניה | ||||
| # 2 | 6 א | Y | הזן את מספר הפלחים או הצמתים | לא של מקטעים | ||||
| # 2 | 6 ב | Y | הזן את אורך הסריקה (בניצב לציר) | רוחב סריקה | ||||
| # 2 | 6 ג | Y | הזן את אורך מעל אשר filopodia מתחיל כיפוף | דקות מדויקות לאחר | ||||
| # 2 | 6 ד | Y | הזן את הרדיוס של מעגל איתור קצה (כלומר אזור שבו קודקוד יכול להיות מקומי במסגרת הבאה) | רדיוס של איתור קצה | ||||
| # 2 | 6e | Y | הזן את הזווית המקסימלית פילופודיום יכול לכופף מן הציר האנכי | זווית סף | ||||
| # 2 | 6F | N | הוסף נקודות התייחסות לבסיס ולעצה עבור מסגרת ספציפית זו | בחר התייחסות | ||||
| # 2 | 6 גרם | N | הזן את אורך מעל אשר filopodia מתחיל כיפוף עבור מסגרת מסוימת | דקות מדויקות לאחר | ||||
| # 2 | <Td> 6hN | הזן את הרדיוס של מעגל האיתור עבור המסגרת הספציפית הזו | רדיוס של איתור קצה | |||||
| # 2 | 6i | N | לאחר הזנת כל הפרמטרים עבור המסגרת הספציפית לחץ כדי לאחסן את הערכים לזיכרון ולקובץ לעיון נוסף | לְהוֹסִיף | ||||
| # 2 | 6 | N | לחץ כדי למחוק את הפרמטרים באופן ידני עבור אותה מסגרת | לִמְחוֹק | ||||
| # 2 | 6k | N | לחץ כדי למחוק את כל הפרמטרים המאוחסנים ידנית באמצעות 'לוח תכונות אופציונלי' עבור כל המסגרות | אִתחוּל | ||||
| # 2 | 6l | N | בדוק לפני המעקב כדי לאחסן את כל תוצאות המעקב בזיכרון לצורך הפניה בעתיד | היסטוריה עקבות | ||||
| # 2 | 7 | Y | לחץ כדי להתחיל במעקב | לעקוב ולנתח | ||||
| # 2 | 8 א | N | לחץ כדי להתחיל מעקב אחר עוצמת ערוץ חלבון | ניתוח חלבון אינטנסיבות | ||||
| # 2 | 8 ב | N | בדוק כדי לעקוב אחר עוצמת ערוץ חלבון לאורך אורך filopodial | פילופודיה שלמה | ||||
| # 2 | 8 ג | N | בדוק עבור מעקב אחר חלבון התייחסות או חלבון A | פרוטאין | ||||
| # 2 | 8 ד | N | בדוק את המעקב אחר החלבון B | פרוטיין | ||||
| # 2 | 8e | N | בדוק את המעקב אחר החלבון C | ProteinC | ||||
| # 2 | 8F | N | בדוק כדי לעקוב אחר עוצמת החלבון הממוצע ב thעצה | קצה מוביל | ||||
| # 2 | 8g | N | הזן את אורך הקצה | אורך עצה | ||||
| # 2 | 8h | N | הזן את המרחק המינימלי מבסיס שמעליה מתחיל הקצה להיווצר | סף | ||||
| # 2 | 8i | N | לחץ כדי לשמור את התוצאות המוביל ניתוח קצה לקובץ | לחץ על הכפתור | ||||
| # 2 | 9 א | N | לחץ כדי להתחיל ניתוח חלבון ratiometric | לְהַשְׁווֹת | ||||
| # 2 | 9b | N | בדוק כדי להשוות בין חלבון B לגבי A | Log10 (B / A) | ||||
| # 2 | 9 ג | N | בדוק כדי להשוות חלבון C ביחס A | Log10 (C / A) | ||||
| # 2 | <Td> 9dN | בדוק כדי להשוות חלבון B ביחס A בקצה | Log10 (B / A) | |||||
| # 2 | 9e | N | בדוק כדי להשוות חלבון C ביחס A בקצה | Log10 (C / A) | ||||
| # 2 | 10 א | N | בחר מפת צבע אחרת (ברירת מחדל: Jetplot) | מפת צבע | ||||
| # 2 | 10 ב | N | ערוך את colormap | עריכת colormap | ||||
טבלה 1: סקירה כללית של כל הפונקציות המופיעות ב- GUI Windows # 1 ו- # 2.
ברגע שזה נעשה, התוכנית יוצרת גוף קמורה באופן אוטומטי מסלולים קצה לאורך הסרט. פרמטרים שחולצו מהסרט, כגון קימוגרפיה רטיומטרית, מהירות גדילה ואורך פילופודיאליRe מוצג גם מאוחסן בתיקיית העבודה כתמונות וכקובצי נתונים. פרמטרים אחרים כגון החיים filopodial, קצב הצמיחה שיעור נסיגה לאחר מכן ניתן לחלץ ונותח נוספת מקבצי הנתונים המאוחסנים ( איור 2 ב ).
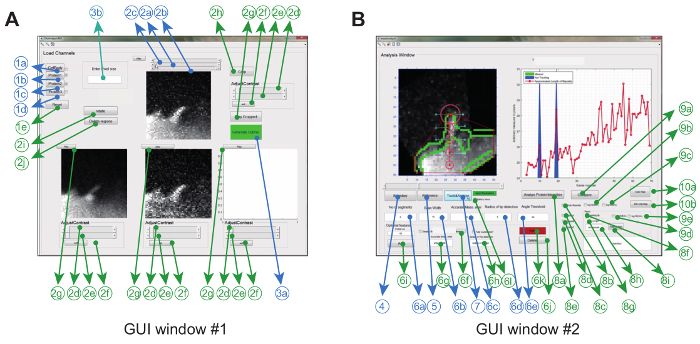
איור 2: ממשק משתמש גרפי לשימוש בתוכנת ניתוח התמונות. ( A ) GUI חלון # 1 משמש לטעינה ועיבוד של תמונות. התוכנית יכולה לטעון עד 3 ערוצי חלבון, לפיה 2 ערוצים מושווים זוג חכם. החלון מגיע עם חובה (כחול) תכונות אופציונלי (ירוק) לעיבוד מראש את התמונות לפני המעקב ( B ) GUI חלון # 2 משמש למעקב אחר פילופודיום, כמו גם ספטיו- temporal ו יחס- metric ניתוח חלבון. שוב, תכונות אופציונליות מסומנות בירוק. נתון זה היה modifiEd מתוך התייחסות 12 . אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
כאן, אנו מציגים פרוטוקול מפורט להכנת המדגם וטיפול בתוכנה. אנו מתחילים עם הוראות מפורטות על תאים culturing ו רכישת סרטים אופטימיזציה עבור ניתוח התמונה. סעיף זה על רכישת נתונים ואחריו תיאור מפורט להפעלת תוכנת ניתוח התמונה. לאורך הפרוטוקול, אנו מציגים צעדים קריטיים ותכונות אופציונליות שיש לקחת בחשבון בעת איסוף ועיבוד נתונים. לבסוף, אנו מנתחים filopodia ממערכות מודל שונים עם תוכנת ניתוח התמונה, לפני הסגירה עם השוואה של התוכנה המתוארת ניתוח התמונה עם תוכניות אחרות הזמינות לכימות filopodial ודיון על מגבלות והכיוון העתידי.
Protocol
Representative Results
Discussion
כאן אנו מציגים פרוטוקול מפורט למעקב דינמיקה הצמיחה filopodial וניתוח של ריכוזי חלבון יחסית מבנים דינמיים אלה באמצעות אלגוריתם קמור האל. באמצעות התוכנה, עד 3 ערוצים ניתן להשוות זוג חכם בריצה אחת, לפיה הריכוזים היחסיים של שני ערוצים ( כלומר חלבונים) נקבע במהלך מחזור הרח…
Disclosures
The authors have nothing to disclose.
Acknowledgements
המחברים מכירים במימון של DFG (EXC-1003 ל- MG).
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
References
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

