Gelijktijdige meting van HDAC1 en HDAC6 activiteit in HeLa cellen met behulp van UHPLC-MS
Summary
De huidige methode dient ter identificatie van isovorm-specifieke remmers van Histon deacetylases (HDAC) in HeLa cellen door de UHPLC-MS-analyse van meerdere substraten. Dit is een antilichaam-vrije methode ontwikkeld om te reflecteren van HDAC1 en HDAC6 activiteit in de levende cel omgeving, in tegenstelling tot één-isovorm cel-gratis testen.
Abstract
De zoektocht naar nieuwe histone deacetylase (HDAC) remmers wordt steeds belangstelling voor drugontdekking. Isovorm selectiviteit is in de spotlight sinds de goedkeuring van romidepsin, een klasse ik HDAC remmer voor kankertherapie, en het klinisch onderzoek van HDAC6-specifieke remmers voor multiple myeloma. De huidige methode wordt gebruikt om te bepalen van de inhiberende activiteit van de verbindingen van de test op HDAC1 en HDAC6 in cellen. De isovorm activiteit wordt gemeten met behulp van de ultra-high-performance vloeibare chromatografie-Spectrometrie van de massa (UHPLC-MS) analyse van specifieke substraten geïncubeerd met behandeld en onbehandeld HeLa cellen. De methode heeft het voordeel van de endogene activiteit van het HDAC binnen de cel-omgeving, in tegenstelling tot cel-vrije biochemische tests uitgevoerd op geïsoleerde isoforms weerspiegelt. Bovendien, omdat het is gebaseerd op het kwantificeren van synthetische substraten, de methode hoeft niet de erkenning van het antilichaam van endogene geacetyleerd eiwitten. Het is gemakkelijk aan te passen aan verschillende cellijnen en een geautomatiseerd proces. De methode is al nuttig zijn bij het vinden van HDAC6-selectieve verbindingen in neuroblasten gebleken. Representatieve resultaten worden hier weergegeven met het standaard HDAC remmers trichostatin een (niet-specifieke), MS275 (HDAC1-specifiek), en tubastatin een (HDAC6-specifiek) met behulp van HeLa cellen.
Introduction
HDACs behoren tot een familie van enzymen kunnen deacetylate van histones binnen de structuur van de chromatine. Ook zij hebben andere substraten eiwit in het cytosol en bevinden zich in verschillende compartimenten van de cel. Een totaal van 18 HDAC isoforms tot nu toe hebben geïdentificeerd en verband met verschillende cel mechanismen, met inbegrip van de verordening van transcriptiefactoren genexpressie, alsmede cel signalering en vervoer van1,2,3,4,5,6,7. HDAC katalytische remmers opgedoken als potentiële therapeutische geneesmiddelen voor kankertherapie. Meeste HDAC remmers momenteel goedgekeurd door de FDA zijn voor de behandeling van T-cel lymfoom en multiple myeloma en zijn niet-specifieke HDAC remmers, zoals vorinostat (SAHA), belinostat en panobinostat8,9. Echter een aantal bijwerkingen zijn geassocieerd met pan-remmers, en het zoeken naar isovorm-specifieke kleine moleculen is een hot topic in medicinale chemie en drug discovery. Dienovereenkomstig, de klasse ik (HDAC1-3 en HDAC8) selectieve remmer romidepsin is een reeds goedgekeurde drug10, terwijl HDAC6-specifieke remmers momenteel onder klinische proeven, met verhoogde therapeutisch potentieel in multiple myeloma11,12,13,14,15 zijn.
Screening tests karakteriseren HDAC zijn remmers gebaseerd op de incubatie van een HDAC substraat met een enzymatische bron (één isovorm, nucleaire extract of cel lysate). Het substraat is meestal een kleine peptide-reeks met een acetyl lysine residu gekoppeld aan een cleavable fluorophore (bijvoorbeeld coumarine), zoals N-(4-methyl-7-aminocoumarinyl)-Nα-(t-butoxycarbonyl)-Nω-acetyllysineamide (MAL)16. Om deze te onderscheiden isovorm-specifieke activiteiten, afzonderlijke cel-gratis testen waarbij elke isovorm zijn noodzakelijk en mogelijk niet overeen met de echte isovorm activiteit in levende cellen. Isovorm-specifieke substraten zijn commercieel verkrijgbaar, zoals benzyl (S)-[1-(4-methyl-2-oxo-2H-chromen-7-ylcarbamoyl)-5-propionylaminopentyl]carbamate (MOCPAC, HDAC1 specifieke substraat) en (S)-[5-acetylamino-1-(2-oxo-4-trifluoromethyl-2H-chromen-7-ylcarbamoyl)pentyl]carbamic zuur tert-butyl ester (BATCP, HDAC6 specifieke substraat) (figuur 1B). Echter toestaat een meer-substraat-mengsel dat MAL bevat, MOCPAC en BATCP gegeven aan levende cellen niet de detectie van de afzonderlijke deacetylated producten door fluorimetrische meting, gezien het feit dat zij de zelfde cleavable fluorophore dragen.
De hier beschreven methode zorgt voor de detectie en kwantificering van de relatieve van elk substraat en haar deacetylated product in HeLa cellen met behulp van een meer-substraat assay gevolgd door UHPLC-ESI-MS/MS analyse17. Een HDAC-test is uitgevoerd op HeLa cellen om de directe identificatie van HDAC inhiberende activiteit en van de specificiteit van de test verbindingen op endogene HDACs. Er is een focus op HDAC1 en HDAC6, die gelijktijdig worden geëvalueerd. Om te bereiken deze enzymatische metingen in een enkele incubatie assay, wordt een mengsel van niet-specifieke en specifieke HDAC substraten toegevoegd aan behandeld en onbehandeld HeLa cellen verguld op een 96-wells-plaat. Na een incubatie stap, zijn cellen lysed de substraten en hun respectieve reactieproducten, die worden gescheiden en ontdekt een UHPLC-MS methode (Figuur 1 c) vrij te laten. De deacetylated producten van de MAL, MOCPAC en BATCP substraten zijn de deacetylated MAL (dMAL), deacetylated MOCPAC (dMOCPAC) en deacetylated BATCP (dBATCP), respectievelijk. Dosis / respons-curven kunnen worden gebouwd met actieve verbindingen.
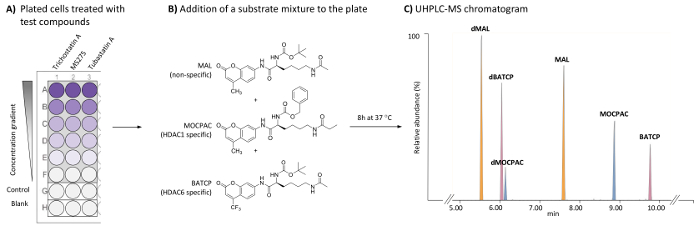
Figuur 1: algemene regeling voor de cel-gebaseerde HDAC assay te identificeren van HDAC1 – en HDAC6-specifieke remmers door de UHPLC-MS-analyse van meerdere substraten. (A) schema van een typische 96-wells-plaat met behandeld (test verbindingen) onbehandeld (control) HeLa cellen, alsmede cel-vrij leeg. (B) chemische structuur van substraten toegevoegd als een mengsel (elke 21 µM) door endogene HDACs worden deacetylated. (C) UHPLC-MS (normaal) chromatografische tonen de pieken van de toegevoegde substraten (MAL, MOCPAC en BATCP) en hun deacetylated producten (dMAL, dMOCPAC, en dBACTP, respectievelijk). Klik hier voor een grotere versie van dit cijfer.
Protocol
Representative Results
Discussion
HDAC inhibition is een hot topic in de Geneesmiddelenontwikkeling, met een huidige focus op HDAC6-selectieve remmers voor kanker therapie21. HDAC selectiviteit wordt meestal beoordeeld door een reeks van high-throughput, cel-gratis tests, die voornemens zijn te bepalen van de remmende potentie naar individuele HDAC isoforms21. Echter de selectiviteit van een remmer moet bovendien bevestigd worden in levende cellen door de beoordeling van de status van de acetylation van end…
Disclosures
The authors have nothing to disclose.
Acknowledgements
De auteurs erkennen kosten actie CM1406 (epigenetische chemische biologie). Onderzoek gemeld in deze publicatie werd gesteund door de Stichting Pierre Mercier.
Materials
| BATCP | Sigma-Aldrich | B4061 | |
| MAL | Sigma-Aldrich | SCP0168 | Synonym: BOC-Ac-Lys-AMC |
| MOCPAC | Sigma-Aldrich | M2195 | |
| MS275 | Sigma-Aldrich | EPS002 | |
| Trichostatin A | Sigma-Aldrich | T8552 | |
| Tubastatin A | Sigma-Aldrich | SML0044 | |
| Acetonitrile, HPLC grade | Fisher Scientific | 10660131 | |
| Formic acid, LC/MS grade | Fisher Scientific | 10596814 | |
| H2O, HPLC grade | distilled H2O filtered through a Milli-Q purification system | ||
| HeLa cells | ATCC | ATCC CRM-CCL-2 | |
| Cell dissociation reagent TrypLE Express | ThermoFisher Scientific | 12604013 | |
| DMSO, cell culture grade | Applichem | 146463 | |
| DPBS | ThermoFisher Scientific | 14190144 | |
| Fetal bovine serum | Biowest | S1810 | |
| MEM | ThermoFisher Scientific | 22561021 | |
| Penicillin-Streptomycin | Bioconcept | 4-01F00-H | |
| 10X RIPA buffer | Abcam | ab156034 | |
| SigmaFast protease inhibitor tablets | Sigma-Aldrich | S8820 | |
| 96-well plates, sterile, flat-bottom, tissue culture treated Corning | VWR | 29442-058 | |
| 96-well plates, non-sterile, V-bottom Corning | VWR | 29442-404 | used in the centrifugation step |
| 96-well plate, conical bottom, Nunc | ThermoFisher Scientific | 249944 | compatible with the Acquity UHPLC system |
| T75 cell culture flasks Corning | Sigma-Aldrich | CLS430641 | |
| Peelable heat sealing foil | Waters | 186002789 | |
| Acquity UPLC system | Waters | ||
| Eppendorf Centrifuge 5810 R | Fisher Scientific | 05-413-323 | |
| Integration software: MassLynx V4.1 | Waters | Catalog number not available | |
| Combi thermo-sealer SP-0669/240 | Waters | Catalog number not available | |
| Quattro micro API Tandem Quadrupole System | Waters | Catalog number not available |
References
- Arrowsmith, C. H., Bountra, C., Fish, P. V., Lee, K., Schapira, M. Epigenetic protein families: a new frontier for drug discovery. Nat Rev Drug Discov. 11 (5), 384-400 (2012).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory and gene regulation. Curr Biol. 26 (14), R644-R648 (2016).
- Kim, C., et al. HDAC6 inhibitor blocks amyloid beta-induced impairment of mitochondrial transport in hippocampal neurons. PLoS One. 7 (8), (2012).
- Kawaguchi, Y., et al. The deacetylase HDAC6 regulates aggresome formation and cell viability in response to misfolded protein stress. Cell. 115 (6), 727-738 (2003).
- Zhao, Y., et al. Acetylation of p53 at lysine 373/382 by the histone deacetylase inhibitor depsipeptide induces expression of p21(Waf1/Cip1). Mol Cell Biol. 26 (7), 2782-2790 (2006).
- Berger, S. L. Histone modifications in transcriptional regulation. Curr Opin Genet Dev. 12 (2), 142-148 (2002).
- Simões-Pires, C., et al. HDAC6 as a target for neurodegenerative diseases: what makes it different from the other HDACs?. Mol Neurodegener. 8 (7), 1-16 (2013).
- Mottamal, M., Zheng, S., Huang, T. L., Wang, G. Histone deacetylase inhibitors in clinical studies as templates for new anticancer agents. Molecules. 20 (3), 3898-3941 (2015).
- Manal, M., Chandrasekar, M. J., Gomathi Priya, J., Nanjan, M. J. Inhibitors of histone deacetylase as antitumor agents: A critical review. Bioorg Chem. 67, 18-42 (2016).
- Mack, G. S. To selectivity and beyond. Nat Biotech. 28 (12), 1259-1266 (2010).
- . Study of ACY-1215 alone and in combination with bortezomib and dexamethasone in multiple myeloma (ACY-1215) Available from: https://clinicaltrials.gov/ct2/show/NCT01323751 (1211)
- . Study of ACY-1215 in combination with lenalidomide, and dexamethasone in multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT01583283 (2012)
- . 13ACY-1215 (ricolinostat) in combination with pomalidomide and low-dose dex in relapsed-and-refractory multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT01997840 (2013)
- . Study of ACY-241 alone and in combination with pomalidomide and dexamethasone in multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT02400242 (2015)
- Yee, A. J., et al. Ricolinostat plus lenalidomide, and dexamethasone in relapsed or refractory multiple myeloma: a multicentre phase 1b trial. Lancet Oncol. 17 (11), 1569-1578 (2016).
- Jung, M. Homogenous non-isotopic assays for histone deacetylase activity. Expert Opin Ther Pat. 13 (6), 935 (2003).
- Zwick, V., Simões-Pires, C., Cuendet, M. Cell-based multi-substrate assay coupled to UHPLC-ESI-MS/MS for a quick identification of class-specific HDAC inhibitors. J Enzyme Inhibi Med Chem. 31 (1), 209-214 (2016).
- Khan, N., et al. Determination of the class and isoform selectivity of small-molecule histone deacetylase inhibitors. Biochem J. 409 (2), 581-589 (2008).
- Glaser, K. B., et al. Differential protein acetylation induced by novel histone deacetylase inhibitors. Biochem Biophys Res Commun. 325 (3), 683-690 (2004).
- Butler, K. V., et al. Rational design and simple chemistry yield a superior, neuroprotective HDAC6 inhibitor, tubastatin A. J Am Chem Soc. 132 (31), 10842-10846 (2010).
- Jung, M., Yong, K. -. J., Velena, A., Lee, S., Sippl, W., Jung, M. Cell-Based Assays for HDAC Inhibitor Hit Validation. Epigenetic Targets in Drug Discovery. , (2010).
- Milli, A., et al. Proteomic analysis of cellular response to novel proapoptotic agents related to atypical retinoids in human IGROV-1 ovarian carcinoma cells. J Proteome Res. 10 (3), 1191-1207 (2011).
- Zwick, V., et al. Synthesis of a selective HDAC6 inhibitor active in neuroblasts. Bioorg Med Chem Lett. 26 (20), 4955-4959 (2016).
- Ciossek, T., Julius, H., Wieland, H., Maier, T., Beckers, T. A homogeneous cellular histone deacetylase assay suitable for compound profiling and robotic screening. Anal Biochem. 372 (1), 72-81 (2008).
- Heltweg, B., Dequiedt, F., Marshall, B. L., Brauch, C., Yoshida, M., et al. Subtype selective substrates for histone deacetylases. J Med Chem. 47 (21), 5235-5243 (2004).
- Copeland, R. A. . Evaluation of enzyme inhibitors in drug discovery. , (2005).

