लाइव प्राथमिक पपनी में उच्च गति सुपर संकल्प गति माइक्रोस्कोपी के आवेदन
Summary
हाल ही में हम तीन आयामी (3 डी) विभिन्न प्रोटीन के लिए परिवहन मार्गों के स्थानिक स्थानों में translocating प्राथमिक cilia अंदर रहते कोशिकाओं में मैप. यहां इस पत्र का विवरण प्रयोगात्मक सेटअप, जैविक नमूनों की प्रक्रिया और डाटा विश्लेषण के लिए 3 डी सुपर संकल्प प्रतिदीप्ति इमेजिंग दृष्टिकोण नव लाइव प्राथमिक cilia में लागू किया ।
Abstract
प्राथमिक पपनी कई युकेरियोटिक कोशिकाओं की सतह पर एक microtubule आधारित दखल है और सेल गतिशीलता और संकेतन में महत्वपूर्ण कार्य है कि प्रोटीन का एक अनूठा पूरक शामिल हैं. चूंकि cilia अपने स्वयं के प्रोटीन synthesizing में असमर्थ हैं, लगभग २०० अद्वितीय सिलिअरी प्रोटीन की जरूरत है cytosol और प्राथमिक cilia के बीच की तस्करी । हालांकि, यह अभी भी एक तकनीकी चुनौती वर्तमान मौजूदा तकनीकों की सीमाओं के कारण लाइव प्राथमिक cilia में इन प्रोटीन के लिए परिवहन रास्ते के तीन आयामी (3d) स्थानों का नक्शा है । चुनौती को जीत के लिए, हाल ही में हम विकसित किया है और एक उच्च गति आभासी 3 डी सुपर संकल्प माइक्रोस्कोपी, के लिए परिवहन रास्ते के 3 डी स्थानिक स्थान निर्धारित करने के लिए एक बिंदु एज-उत्तेजना उप विवर्तन (गति) माइक्रोस्कोपी कार्यरत है जीवित कोशिकाओं के प्राथमिक cilia में cytosolic और झिल्ली प्रोटीन दोनों । इस अनुच्छेद में, हम गति माइक्रोस्कोपी के विस्तृत सेटअप, प्रतिदीप्ति व्यक्त कोशिकाओं की तैयारी प्रदर्शित करेगा-प्रोटीन लेबल सिलिअरी प्रोटीन, लाइव पपनी में व्यक्तिगत प्रोटीन के वास्तविक समय एकल अणु ट्रैकिंग और उपलब्धि सिलिअरी प्रोटीन के लिए परिवहन मार्गों के 3 डी स्थानिक संभावना घनत्व नक्शे के ।
Introduction
के बाद से १८७३ में अर्नस्ट अब्बे द्वारा कहा गया है, पारंपरिक प्रकाश माइक्रोस्कोपी के संकल्प को लगभग २०० उद्देश्य1,2से प्रकाश विवर्तन के कारण एनएम तक ही सीमित माना गया है । वर्तमान में, सुपर संकल्प प्रकाश माइक्रोस्कोपी तकनीक इस सीमा को तोड़ने और उप-विवर्तन (< २०० एनएम) संकल्प के साथ गतिशील छवियों का कब्जा करने की अनुमति । तकनीक आम तौर पर दो व्यापक श्रेणियों में गिर: प्रेरित उत्सर्जन घट (STED) सूक्ष्म आधारित दृष्टिकोण है, जो उप विवर्तन रोशनी की वजह से नमूनों में fluorophores के रैखिक ऑप्टिकल प्रतिक्रिया के कारण मात्रा उत्पंन3; और photoactivated प्रकाश माइक्रोस्कोपी (पाम) और stochastic ऑप्टिकल पुनर्निर्माण माइक्रोस्कोपी (तूफान) आधारित सुपर संकल्प तकनीक है, जो गणितीय कार्यों का उपयोग करने के लिए fluorophores के centroids स्थानीयकृत और फिर पुनर्गठन इन centroids सुपर संकल्प छवियों4,5बनाने के लिए । वर्तमान में, अपेक्षाकृत सीधी ऑप्टिकल सेटअप, पाम और तूफान के कारण बड़े पैमाने पर केवल एक जैविक तैयारी की एक लंबी वीडियो के प्रत्येक फ्रेम में fluorophores के एक छोटे सबसेट को सक्रिय द्वारा नियोजित कर रहे हैं । इस फ्लोरोसेंट जगह के 2 डी गाऊसी फिटिंग द्वारा और अधिक सटीक स्थानीयकरण के लिए अनुमति देता है, वीडियो के प्रत्येक फ्रेम में फ्लोरोसेंट-लेबल प्रोटीन की बात फैल समारोह (पीएसएफ), कहा । प्रत्येक फ्लोरोसेंट लेबल अणु के 2d स्थान तो एक इमेजिंग विमान पर आरोपित हो सकता है जैविक तैयारी1,2के एक सुपर संकल्प छवि का उत्पादन । हालांकि इन एकल अणु स्थानीयकरण, सूक्ष्मता के लिए सुपर संकल्प दृष्टिकोण निश्चित रूप से क्रांति कैसे जैविक नमूनों की इमेजिंग किया गया था, वहां अभी भी चुनौतियों से उबरने के लिए कर रहे हैं । उदाहरण के लिए, तूफान और हथेली जैविक नमूनों के निर्धारण के बाद अपने सबसे अच्छे स्थानिक संकल्प को प्राप्त कर सकते हैं और इस प्रकार इलेक्ट्रॉन माइक्रोस्कोपी की एक ऐसी ही सीमा है जो फ्लोरोसेंट लेबल प्रोटीन, का एक स्थिर प्रतिनिधित्व पेश करते हैं । इसके अतिरिक्त, लाइव कोशिकाओं में प्रत्येक फ्लोरोसेंट लेबल प्रोटीन के लिए उच्च स्थानिक संकल्प प्राप्त करने के लिए, नमूनों बहुत लंबे समय framerates जो प्रोटीन गतिशीलता पर कब्जा करने में असमर्थ है पर imaged किया जाना चाहिए । इसलिए इन मुख्य तकनीकी बाधाओं को दूर करना आवश्यक है ।
एक उच्च spatiotemporal संकल्प है कि तेजी से चलती प्रोटीन या जीवित कोशिकाओं में RNAs का पता लगाने के लिए अच्छी तरह से अनुकूल है प्राप्त करने के लिए, हम अपनी प्रयोगशाला में सुपर संकल्प गति माइक्रोस्कोपी विकसित की है (चित्रा 1)6,7, 8. गति माइक्रोस्कोपी में कई प्रमुख तकनीकी अग्रिमों पहले हमें सफलतापूर्वक देशी परमाणु ताकना परिसरों के माध्यम से छोटे अणुओं, प्रोटीन, mRNA और वायरस के nucleocytoplasmic परिवहन को ट्रैक करने के लिए सक्षम है (NPCs)6, 7 , 8. संक्षेप में, गति माइक्रोस्कोपी की निम्नलिखित विशेषताएं लाइव कोशिकाओं में उप माइक्रोमीटर घूर्णन रूप से सममित संरचनाओं के माध्यम से तेजी से चलती अणुओं ट्रैक करने के लिए इस्तेमाल किया जाएगा, ऐसे NPCs और प्राथमिक cilia के रूप में: (1) एक झुका या एक कार्यक्षेत्र प्रदीप्ति पीएसएफ फोकल विमान में एक छोटे से विवर्तन-सीमा मात्रा के भीतर एकल अणुओं के उत्तेजना सक्षम बनाता है (चित्रा 1); (2) झुका पीएसएफ बहुत बाहर के ध्यान प्रतिदीप्ति से बचने और इस तरह संकेत करने वाली शोर अनुपात में सुधार कर सकते हैं । (3) रोशनी पीएसएफ में 100-500 किलोवाट/सेमी2 के ऑप्टिकल घनत्व फोटॉनों के हजारों की अनुमति देता है तेजी से पता लगाने की गति के साथ एकल fluorophores से एकत्र (> ५०० हर्ट्ज) । (4) तेजी से पता लगाने की गति भी बहुत ही जीवित कोशिकाओं में फ्लोरोसेंट अणुओं हिल के स्थानिक पथ का निर्धारण करने में एकल अणु स्थानिक स्थानीयकरण त्रुटि (< 10 एनएम) को कम कर देता है, क्योंकि आणविक प्रसार प्रमुख कारकों में से एक है अणु बढ़ने के लिए एकल अणु स्थानीयकरण की खामियों के कारण. (5) अच्छी तरह से 3 डी रूपांतरण एल्गोरिदम के लिए 2 डी की स्थापना की हम एनपीसी या प्राथमिक पपनी में अणुओं के लिए परिवहन मार्गों के 3d स्थानिक संभाव्यता घनत्व नक्शे प्रदान करने के लिए सक्षम । यह उल्लेखनीय है कि काटीज़ियनवादी और बेलनाकार समंवय प्रणाली के बीच हमारी रूपांतरण प्रक्रिया 3 डी एकल अणु ट्रैकिंग (चित्रा 2) के बजाय एक 3 डी स्थानिक संभावना घनत्व नक्शा उत्पंन करने के लिए प्रयोग किया जाता है । पहले, इलेक्ट्रॉन माइक्रोस्कोपी डेटा से पता चला है कि एनपीसी ने9,10 और प्राथमिक पपनी11 दोनों एक घूर्णन सममित संरचना है । सिद्धांत रूप में, बेतरतीब ढंग से बढ़ते अणुओं को एनपीसी या प्राथमिक पपनी के माध्यम से भी घूर्णन रूप से सममित वितरण होना चाहिए । के रूप में चित्रा 2में दिखाया गया है, सिलेंडर के अंदर बेतरतीब ढंग से फैलाना अणुओं की एक उच्च संख्या है कि एनपीसी में के रूप में पार अनुभाग देखने में रोटेशन सममित वितरण उत्पंन होगा, आगे एक लगभग एक समान स्थानिक में जिसके परिणामस्वरूप दो पड़ोसी के छल्ले के बीच प्रत्येक बहुत छोटे उप क्षेत्र के भीतर वितरण (चित्रा 2ई) । इस वर्दी वितरण की ओर जाता है कि बेलनाकार प्रणाली में θ आयाम के साथ स्थानिक वितरण स्थिर है । फिर 3d निर्देशांक (r, x, θ) को 2d निर्देशांक (r, x, स्थिरांक) होना सरल हो सकता है. दरअसल, काटीज़ियनवादी और बेलनाकार सिस्टम के बीच हमारी रूपांतरण प्रक्रिया 2d (x, Y) से 2d (R, x, स्थिरांक) तक है । लगातार θ,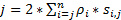 चित्रा 2ईमें स्थानिक घनत्व पी को संदर्भित करता है, समीकरण एकका उपयोग करके की गणना की है ।
चित्रा 2ईमें स्थानिक घनत्व पी को संदर्भित करता है, समीकरण एकका उपयोग करके की गणना की है ।
अंततः, एकल अणु ट्रैकिंग जैविक अनुसंधान में व्यापक आवेदन किया है, इस प्रकार, यह स्वाभाविक है कि तकनीक के ढेर सारे विशिष्ट जैविक niches12,13,14भरने के लिए विकसित की जाएगी । इस तरह की गति माइक्रोस्कोपी के साथ मामला है । पहले, जब एक 3 डी परिवर्तन एल्गोरिथ्म के साथ युग्मित, इस तकनीक को NPCs, एक उप विवर्तन के आकार और रोटेशन सममित जैविक संरचना6के माध्यम से पारगमन अणुओं के 3 डी परिवहन मार्गों को हल करने के लिए विकसित किया गया था । इस पत्र में प्राथमिक cilia को उत्कृष्ट मॉडल organelles के रूप में भी दिखाया गया है. प्राथमिक cilia बेलनाकार हैं, एंटीना की तरह organelles (~ १२५ एनएम त्रिज्या) कि परियोजना के सबसे स्तनधारी कोशिकाओं की सतह से15,16,17। वे बाहरी संकेतों को प्राप्त करने और एक intracellular आम तौर पर विकास और चयापचय15,16के साथ जुड़े प्रतिक्रिया संचारित करने के लिए जिंमेदार हैं । इसलिए, संरचनात्मक प्रोटीन का प्रवाह, transmembrane रिसेप्टर्स के पुनर्चक्रण, और intracellular दूतों के संचरण प्राथमिक cilia के महत्वपूर्ण जिम्मेदारियां हैं. प्राथमिक cilia और कोशिका शरीर के बीच के मोड़ पर एक महत्वपूर्ण selectivity बाधा है, संक्रमण क्षेत्र या TZ कहा जाता है, जिसके माध्यम से यह सभी प्रोटीन परिवहन होना चाहिए11,18,19, 20. TZ के गेटिंग फंक्शन के अलावा, कम से दो परिवहन प्रक्रियाओं, intraflagellar परिवहन और निष्क्रिय प्रसार, इस क्षेत्र के माध्यम से प्रोटीन की आवाजाही के लिए जिंमेदार माना जाता है16,21, 22. एक मानव स्वास्थ्य के दृष्टिकोण से, प्राथमिक cilia और बहाव संकेतन के बाद के विनियमन के नुकसान कई कैंसर की विशेषता है । इसके अलावा, कई आनुवंशिक रोगों, जैसे Bardet-Biedl सिंड्रोम और पॉलीसिस्टिक गुर्दे की बीमारी, दोषपूर्ण प्रोटीन परिवहन के साथ जुड़े रहे हैं23। दोनों उप विवर्तन सीमा आकार और TZ के माध्यम से चयनात्मक प्रोटीन परिवहन की जटिल प्रक्रिया प्राथमिक cilia इस तकनीक के लिए एक प्रमुख लक्ष्य बनाते हैं । इस तरीके कागज में, हम एक सिलिअरी transmembrane प्रोटीन, सोमेटोस्टेटिन रिसेप्टर 3 (SSTR3) के ट्रैकिंग का प्रदर्शन करेंगे24, Alexa Fluor ६४७ और IFT, IFT2025के एक घटक, एक फ्यूज GFP अणु के साथ लेबल के साथ बाह्य लेबल ।
Protocol
Representative Results
Discussion
इस प्रोटोकॉल प्राथमिक पपनी, एक सेलुलर संकेत organelle है कि कुशल प्रोटीन परिवहन पर अत्यधिक निर्भर है की गति माइक्रोस्कोपी के आवेदन का वर्णन । गति माइक्रोस्कोपी फ्लोरोसेंट-लेबल अणुओं के लिए उच्च संकल्प (< 10 ए?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
हम डॉ िईद्भस्टेन Verhey (मिशिगन विश्वविद्यालय, एन आर्बर) और डॉ ग्रेगरी Pazour (मैसाचुसेट्स मेडिकल स्कूल के विश्वविद्यालय) कुछ plasmids प्रदान करने के लिए धंयवाद । इस परियोजना को राष्ट्रीय स्वास्थ्य संस्थानों (NIH GM097037, GM116204 और GM122552 W.Y.) से अनुदान द्वारा समर्थित किया गया था ।
Materials
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25×36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
References
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell’s antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. 신경과학. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).

