凝胶序列: 一种同时测序库的方法用水凝胶基质制备 DNA 和 RNA
Summary
凝胶序列使研究人员能够同时为 DNA 和 RNA 的存储提供从 100-1000 细胞开始使用简单的水凝胶装置的可忽略的额外成本。本文给出了该装置的制作方法, 以及生成配对库的生物协议。
Abstract
在过去的五年里, 从小的起始样本中放大和序列 DNA 或 RNA 的能力才得以实现。不幸的是, 生成基因组或 transcriptomic 库的标准协议是不相容的, 研究人员必须选择是否为特定的样本序列 DNA 或 RNA。凝胶序列解决了这个问题, 使研究人员可以同时准备的 DNA 和 RNA 的图书馆从 100-1000 细胞使用一个简单的水凝胶装置。本文给出了该装置的制作方法, 以及生成配对库的生物协议。我们设计的凝胶序列, 使它可以很容易地执行其他研究员;许多遗传学实验室已经有必要的设备, 以重现凝胶序列的设备制造。我们的协议使用常用的工具包进行全文本放大 (transcriptomic) 和库准备, 这对于已经精通生成基因组和库的研究人员也很有可能熟悉。我们的方法使研究人员能够在不分裂和微不足道的附加成本的情况下, 在单个样品上携带 DNA 和 RNA 序列的能量。
Introduction
下一代测序技术对遗传学研究的实施方式产生了深远的影响。研究人员曾经专注于对整个物种的基因组进行排序, 现在可以在一个实验中序列化单个肿瘤的基因组, 甚至单个细胞。1转录还使在单元格中找到的 RNA 记录集 (称为 “数据收集”) 的序列化具有成本效益。在过去的五年里, 从小的起始样本中放大和序列 DNA 或 RNA 的能力才得以实现。2,3,4不幸的是, 标准协议是不兼容的, 研究人员必须选择是否为给定的样本序列 DNA 或 RNA。当一个起始样本足够大时, 它可以分成两半。然而, 在较小的尺度上, 由于分裂样本造成的材料损失会影响到图书馆的质量, 而样品的汇集可以平均出细胞之间的有趣变化。5此外, 研究人员越来越热衷于检查不能分割的样本, 如单细胞或小的异质肿瘤活检。6
为了解决这一问题, 最近开发了三项协议, 用于从同一起始样本中序列化 DNA 和 RNA: 凝胶顺序7、G & t-序列8和 DR-序列9。本文给出了凝胶序列的详细协议, 可用于同时生成少量100细胞的 DNA 和 RNA 库, 其成本可以忽略不计。凝胶序列的新方面是将 DNA 和 RNA 完全用低成本水凝胶基质分离出来的能力。凝胶序列协议的核心创新是 DNA 与 RNA 的物理分离。这种分离是实现电泳使用聚丙烯酰胺膜的组合, 利用这些分子之间的大小差异。要将这些大小差异放在上下文中, 请考虑 dna 和 RNA 是如何成像的: 虽然 dna 存在于微米尺度上, 可以用传统显微镜观察, 但 RNA 存在于纳米尺度上, 必须使用诸如低温电子等复杂技术进行成像。显微镜.10
在这个协议中分离 DNA 和 RNA 的方法如图 1所示。左面板显示 DNA 和 RNA 游离漂浮在溶液附近的膜。当电场被应用时, 如右面板所示, DNA 和 RNA 经历了一种电泳力, 通过细胞膜诱导迁移。通过调整膜的性能, 我们创造了一种半渗透膜, 将 DNA 与 RNA 分离。DNA 分子被推到膜上, 但由于它们的体积大而被边缘纠缠。小 RNA 分子, 另一方面, 可以重新配置和编织他们的方式通过膜。这个过程, 被称为蠕动, 类似于蛇在草丛中穿行的方式。最终, 这些 RNA 分子被第二个高密度膜所阻挡, 对于更小的聚合物 (> 200 基对) 来说, 这是非常困难的。一旦物理分离, DNA 和 RNA 可以被恢复和处理, 以产生关于基因组和转录的信息。虽然我们可以分离 DNA 和 rna, 我们发现更好的结果, 如果 RNA 反转转录为 cDNA 在分离之前。cDNA/rna 杂交种比 rna 更稳定, 还能通过低密度膜。
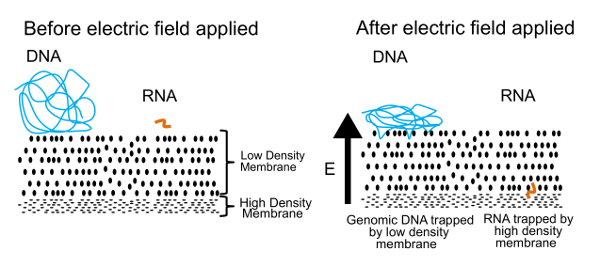
图 1.凝胶的操作原理.用于物理分离 DNA 和 RNA 的基本原则。在应用电场中, 小 RNA 分子通过低密度膜迁移, 但大量的 DNA 分子被困在表面。这个数字是从参考文献7中转载的, 得到了皇家化学学会的许可。请单击此处查看此图的较大版本.
本文详细介绍了凝胶序列器件的制备和生成配对 DNA 和 RNA 库的生物协议。这两者的概览都显示在图 2中。该装置是通过分层三种不同密度的聚丙烯酰胺凝胶在一个类似于创建标准的堆叠凝胶的过程中制造的。11生物协议从 PBS 中悬浮的 100-1000 细胞开始。细胞被裂解, RNA 被转化为 cdna, 然后用该装置将基因组 DNA 与 cdna/RNA 杂交体分离。在分离和恢复后, 基因组和 transcriptomic 库是使用一个密切遵循标准全基因组库准备套件协议的过程编写的。关于凝胶序列的发展和验证的进一步细节可以在实验室中读到一个芯片出版物“凝胶序列: 全基因组和转录测序通过同时低输入 DNA 和 RNA 库准备使用半透水水凝胶屏障.”7
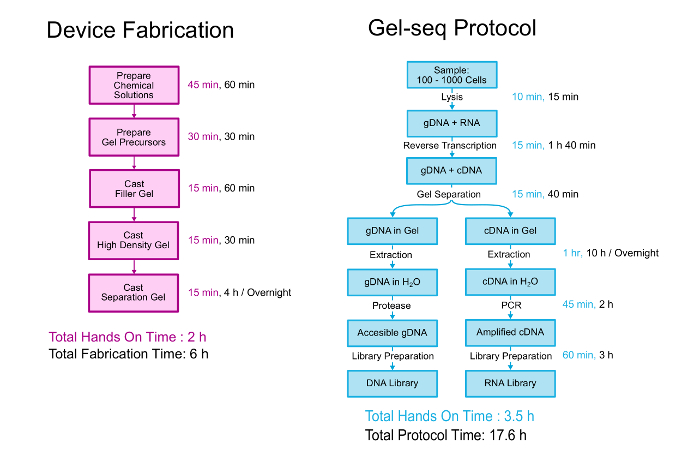
图 2.凝胶序列协议.概述了制造凝胶序列装置的步骤和生成配对 DNA 和 RNA 库的协议。这个数字的部分从参考文献7被复制了从皇家化学学会的允许。请单击此处查看此图的较大版本.
为了从单细胞中生成 DNA 和 rna 库, 研究人员应该考虑使用 g & t 序列或 DR-下克 & t 序列, 如凝胶序列, 依赖于从基因组 DNA 中提取 rna 的物理分离。这种方法依靠信使 RNA 的 (mRNA) 3′ polyadenylated 尾巴作为下拉目标。该 mRNA 使用生物素化寡核苷酸底漆在磁珠上捕获。一旦基因被捕获, 珠子就会被放置在一个磁铁上, 含有基因组 DNA 的上清液可以被移除并转移到另一管。在这种物理分离完成后, 可以从 mRNA 和 DNA 中生成独立的库。8如果感兴趣的 RNA 是 polyadenylated, 则此方法很有效, 但是它不能用于研究非 polyadenylated 转录, 如核糖体 rna、tRNA 或原核生物中的 rna。
DR 序列依赖于一个预放大的步骤, 从 RNA 中提取的 DNA 和 cDNA 在同一管中被放大。然后将该样本分成两个, 并并行处理以准备 DNA 和 RNA 序列库。为了区分基因组 DNA 和 RNA 衍生的 cDNA, DR 序列采用了计算方法。只有外显子存在的序列在基因组 DNA 数据中被计算抑制, 因为它们可能来源于 DNA 或 RNA。9这种方法的优点是, DNA 和 cDNA/RNA 不需要物理分离, 如在凝胶序列和 G & T。然而, 缺点是, 转录博士需要先验知识的基因组和 (即, 外显子与内含子), 并可能不理想的应用, 如核测序, 其中许多成绩单尚未完全拼接并仍包含内含子。12
凝胶序列的新的方面是能够分离 DNA 和 RNA 在数以百计的细胞完全基于大小。此方法不需要先验知识的基因组或转录, 是强大的反对不完全剪接, 并不仅限于多 adenylated 成绩单。对于研究人员可以从至少100个细胞开始的应用, 凝胶序列提供了一个简单的方法使用廉价和广泛可用的材料。
Protocol
Representative Results
Discussion
有几个关键步骤相关的凝胶序列设备的制造以及协议本身。在制作过程中, 我们建议从规定的层厚度开始, 为凝胶的各个区域。我们花了大量的时间测试不同的制造选项, 此处描述的协议为材料和试剂表中列出的磁带制作最佳设备。如果研究人员使用另一种卡带系统, 他们可能会发现有必要调整在创建设备时使用的卷。制造中的主要挑战是, 如果高密度凝胶区域太大, 它可以从卡带的边?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
这项工作的经费由圣地亚哥大学、国家科学基金会研究生研究金方案、NIH 赠款 R01-HG007836 以及韩国科学、信息和通信技术和未来规划部提供。
早期版本的几个数字首次发表在 “Hoople, 动力局等。凝胶序列: 用半透水水凝胶屏障同时进行低输入 DNA 和 RNA 文库制备的全基因组和转录测序。实验室在芯片 17, 2619-2630, 对: 10.1039/c7lc00430c (2017)。芯片上的实验室已经批准了这个出版物中的数字重用。
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

