Gel-seq : PROCEDE de Preparation de bibliothèque simultanée de séquençage de l’ADN et l’ARN à l’aide de Matrices d’Hydrogel
Summary
Gel-seq permet aux chercheurs de préparer simultanément les bibliothèques pour les deux ADN – et RNA-seq au surcoût négligeable à partir de 100-1000 cellules à l’aide d’un dispositif simple hydrogel. Cet article présente une approche détaillée pour la fabrication de l’appareil ainsi que le protocole biologiques pour générer des bibliothèques appariés.
Abstract
La capacité d’amplifier et de séquences d’ADN ou ARN de petits échantillons de départ est parvenue seulement dans les cinq dernières années. Malheureusement, les protocoles standard pour la production génomique ou bibliothèques de transcriptomique sont incompatibles et chercheurs doivent choisir de séquence d’ADN ou ARN pour un échantillon donné. Gel-seq résout ce problème en permettant aux chercheurs préparer simultanément des bibliothèques pour les ADN et d’ARN à partir de 100-1000 cellules à l’aide d’un dispositif simple hydrogel. Cet article présente une approche détaillée pour la fabrication de l’appareil ainsi que le protocole biologiques pour générer des bibliothèques appariés. Nous avons conçu le Gel-seq afin qu’il pourrait être facilement mis en œuvre par d’autres chercheurs ; de nombreux laboratoires de génétique ont déjà l’équipement nécessaire pour reproduire la fabrication de dispositifs de Gel-seq. Notre protocole emploie couramment utilisé des kits pour les deux ensemble-transcription amplification (WTA) et préparation de bibliothèque, qui sont également susceptibles d’être familière aux chercheurs déjà versé dans la génération génomique et transcriptomique bibliothèques. Notre approche permet aux chercheurs de mettre à profit la puissance du séquençage de l’ADN et l’ARN sur un seul échantillon sans le découper et avec surcoût négligeable.
Introduction
Prochaine génération séquençage (NGS) a eu un impact profond sur le déroulement de la recherche en génétique. Là où autrefois, chercheurs se sont penchés sur le séquençage du génome d’une espèce entière, il est maintenant possible de séquencer le génome d’une tumeur unique ou même une seule cellule dans une expérience. 1 NGS a également rendu rentable pour séquencer les transcriptions d’ARN présentes dans une cellule, une collection de données appelées le transcriptome. La capacité d’amplifier et de séquences d’ADN ou ARN de petits échantillons de départ est parvenue seulement dans les cinq dernières années. 2 , 3 , 4 malheureusement, protocoles standard sont incompatibles et chercheurs doivent choisir de séquence d’ADN ou ARN pour un échantillon donné. Lorsqu’un échantillon de départ est assez grand, il peut être divisé en deux. Toutefois, à des échelles plus petites, perte de matériel en raison de la séparation des échantillons peut affecter la qualité de la bibliothèque et mise en commun des échantillons peut moyenne des variations intéressantes entre les cellules. 5 par ailleurs, les chercheurs sont plus en plus intéressés à l’examen d’échantillons qui ne peut pas être divisés, comme des cellules individuelles ou des biopsies de petite tumeur hétérogène. 6
Pour résoudre ce problème, trois protocoles ont récemment été mis au point pour séquencer l’ADN et l’ARN de l’échantillon de départ même : Gel-seq7, G & T-seq8et9de la DR-seq. Cet article présente un protocole détaillé pour Gel-seq, qui peut être utilisé pour générer simultanément bibliothèques d’ADN et d’ARN à partir d’aussi peu que 100 cellules au surcoût négligeable. La nouveauté de Gel-seq est la capacité de séparer l’ADN et l’ARN fondée exclusivement sur la taille à l’aide de matrices de faible coût hydrogel. L’innovation de base du protocole Gel-Seq est la séparation physique de l’ADN de l’ARN. Cette séparation se fait par électrophorèse en utilisant une combinaison des membranes de polyacrylamide qui tirent parti de la différence de taille entre ces molécules. Pour mettre ces différences de taille en contexte, examiner comment l’ADN et l’ARN sont imagés : alors que l’ADN existe sur l’échelle de micron et peut être consulté à l’aide de microscopes traditionnels, ARN existe à l’échelle du nanomètre et doit être copié à l’aide de techniques complexes comme cryo-electron microscopie. 10
L’approche à la séparation de l’ADN et l’ARN dans le présent protocole est illustré à la Figure 1. Le panneau de gauche montre l’ADN et l’ARN libre flottant dans la solution près d’une membrane. Lorsqu’un champ électrique est appliqué, comme indiqué dans le panneau de droite, ADN et l’ARN l’expérience une force électrophorétique qui induit la migration à travers la membrane. En ajustant les propriétés de la membrane, nous avons créé une membrane semi-perméable qui sépare l’ADN de l’ARN. Les molécules d’ADN sont poussés contre la membrane, mais sont accrocher au bord à cause de leur grande taille. En revanche, les petites molécules d’ARN, peuvent reconfigurer et tissent leur chemin à travers la membrane. Ce processus, connu comme la reptation, est semblable à la manière de qu’un serpent se déplace dans l’herbe. Finalement, ces molécules d’ARN sont arrêtés par une deuxième membrane haute densitée qui est trop difficile pour les polymères encore plus petits (> 200 paires de bases) de se soustraire à travers. Une fois physiquement séparés, l’ADN et l’ARN peuvent être récupérées et traitées pour générer des informations sur le génome et le transcriptome. Alors que nous pouvons séparer les ADN et ARN, nous avons identifié les meilleurs résultats sont obtenus si l’ARN est inverse, transcrit d’ADNc avant la séparation. Les hybrides de cDNA/ARN sont plus stables que RNA seule et peuvent toujours passer à travers la membrane de faible densitée.
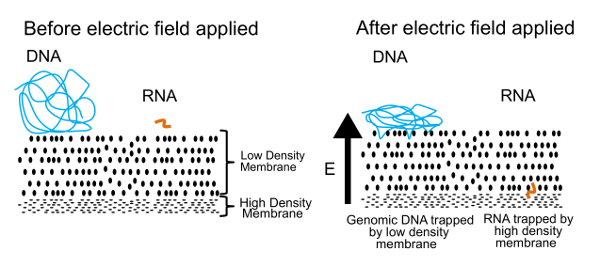
Figure 1 . Principe de fonctionnement de gel-seq. Le principe sous-jacent utilisé pour séparer physiquement les ADN et ARN. Dans un champ électrique appliqué, petites molécules d’ARN migrent à travers la membrane de faible densitée, mais grosses molécules d’ADN sont pris au piège à la surface. Ce chiffre a été reproduit de Réf. 7 avec la permission de la Royal Society of Chemistry. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Cet article décrit en détail les deux la fabrication de l’appareil de Gel-seq et le protocole biologiques pour générer jumelé bibliothèques d’ADN et d’ARN. Un aperçu des deux est illustré à la Figure 2. Le dispositif est fabriqué par marcottage trois densité différente des gels de polyacrylamide uns sur les autres dans un processus similaire à la création des gels empilables standard. 11 le protocole biologique commence par 100-1000 cellules suspendues dans du PBS. Les cellules sont lysées et l’ARN est convertie en ADNc avant que l’appareil est utilisé pour séparer l’ADN génomique des hybrides cDNA/ARN. Après la séparation et de récupération, génomique et transcriptomique bibliothèques sont préparés à l’aide d’un processus qui suit de près le protocole de kit de préparation de bibliothèque standard du génome entier. Plus de détails sur le développement et la validation de Gel-seq peut être lu dans son laboratoire sur une publication de la puce « Gel-seq : génome entier et le séquençage de transcriptome de faibles intrants ADN et l’ARN bibliothèque préparation simultanée à l’aide de barrières semi-perméables hydrogel .” 7
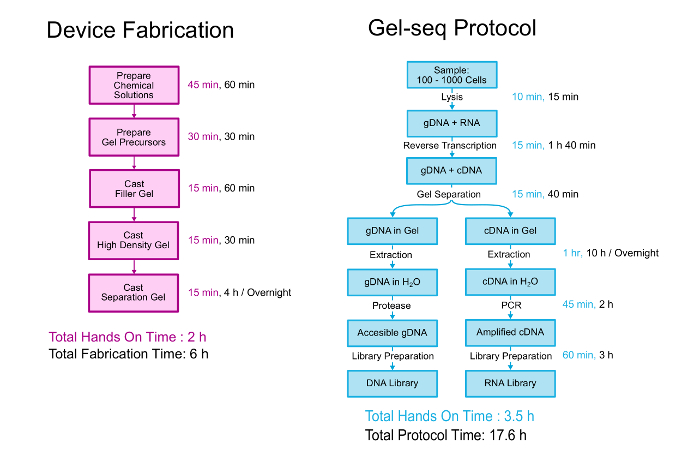
Figure 2 . Protocole gel-seq. Une vue d’ensemble des étapes pour fabriquer le dispositif Gel-seq et le protocole à généré jumelé bibliothèques d’ADN et d’ARN. Certaines parties de cette figure ont été reproduits à partir Réf. 7 avec la permission de la Royal Society of Chemistry. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Pour générer des bibliothèques de l’ADN et l’ARN de cellules individuelles, les chercheurs doivent envisager d’utiliser soit G & T-seq ou DR-suiv. G & T-Seq, comme Gel-seq, repose sur une séparation physique de l’ARN de l’ADN génomique. Cette approche s’appuie sur l’ARN messager (ARNm) 3′ polyadénylé queue comme une menu déroulant cible. L’ARNm est capturée sur une pastille magnétique utilisant une amorce d’oligo-dT biotinylé. Une fois l’ARNm a été capturée, les perles sont maintenus en place avec un aimant et le surnageant contenant l’ADN genomic peut être enlevé et transféré dans un autre tube. Après que cette séparation physique est terminée, bibliothèques distinctes peuvent être générés de l’ARNm et l’ADN. 8 cette approche fonctionne bien si l’ARN d’intérêt est polyadénylé, cependant il ne peut pas servir à étudier non polyadénylé transcriptions, tels que les ARN ribosomiques, ARNt ou l’ARN des procaryotes.
DR-seq s’appuie sur une étape de pré amplification où l’ADN et ADNc dérivé de l’ARN sont amplifiés dans le même tube. L’échantillon est alors divisé en deux et traitée en parallèle de préparer des bibliothèques d’ADN – et RNA-seq. Pour distinguer l’ADN génomique et l’ADNc dérivé de RNA, DR-seq adopte une approche computationnelle. Séquences où se trouvent seulement les exons sont supprimés par le calcul dans les données d’ADN génomiques, comme ceux qui pourraient provenir de l’ADN ou ARN. 9 un avantage de cette approche est que l’ADN et l’ARNC/ARN ne doivent pas être physiquement séparés comme cela se fait en Gel-seq et G & T-suiv. L’inconvénient, cependant, est que DR-seq nécessite une connaissance a priori du génome et du transcriptome (c.-à-d., les exons et introns) et qu’il n’est peut-être pas idéal pour des applications telles que le séquençage de noyaux, dans lequel plusieurs transcriptions ne sont pas encore pleinement épissé et contiennent encore des introns. 12
La nouveauté de Gel-seq est la capacité de séparer l’ADN et l’ARN dans des centaines de cellules basées exclusivement sur la taille. Cette méthode nécessite aucune connaissance a priori du génome ou du transcriptome, est robuste contre l’épissage incomplète et n’est pas limitée aux transcriptions de poly-adenylated. Pour les applications où un chercheur peut commencer avec au moins 100 cellules, Gel-seq offre une approche simple à l’aide de matériaux bon marché et largement disponible.
Protocol
Representative Results
Discussion
Il y a plusieurs étapes critiques associées à la fabrication de dispositifs de Gel-seq ainsi que le protocole lui-même. Pendant la fabrication, nous vous recommandons de commencer avec les épaisseurs de couche prescrite pour les différentes régions du gel. Nous avons passé beaucoup de temps test options de fabrication différente et le protocole décrit ici produit les meilleurs dispositifs pour les cassettes répertoriés dans Table des matières et réactifs. Si les chercheurs utilisent un syst…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Financement de ce travail a été fourni par l’Université de San Diego, la National Science Foundation recherche Fellowship programme d’études supérieures, accorder des NIH R01-HG007836 et par le ministère coréen des sciences, de TIC et de la planification Future.
Les versions antérieures d’une plusieurs personnalités ont été tout d’abord publiées dans « Hoople, G. D. et al. Gel-seq : génome entier et séquençage de transcriptome de faibles intrants ADN et l’ARN bibliothèque préparation simultanée à l’aide de barrières semi-perméables hydrogel. Laboratoire sur une puce 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). » Laboratoire sur une puce a sanctionné la réutilisation des figures dans cette publication.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

