Gel-seq: एक साथ अनुक्रमण पुस्तकालय के लिए एक विधि डीएनए और आरएनए का उपयोग Hydrogel मैट्रिक्स की तैयारी
Summary
जेल-seq एक सरल hydrogel डिवाइस का उपयोग कर 100-1000 कोशिकाओं से शुरू नगण्य जोड़ा लागत पर दोनों डीएनए और आरएनए-seq के लिए एक साथ पुस्तकालयों को तैयार करने के लिए शोधकर्ताओं को सक्षम बनाता है. यह कागज डिवाइस के निर्माण के लिए एक विस्तृत दृष्टिकोण के रूप में के रूप में अच्छी तरह से जैविक प्रोटोकॉल युग्मित पुस्तकालयों उत्पंन करने के लिए प्रस्तुत करता है ।
Abstract
छोटे से प्रारंभिक नमूनों से या तो डीएनए या आरएनए को बढ़ाना और अनुक्रम की क्षमता केवल पिछले पांच वर्षों में प्राप्त की गई है. दुर्भाग्य से, जीनोमिक या transcriptomic पुस्तकालयों पैदा करने के लिए मानक प्रोटोकॉल असंगत हैं और शोधकर्ताओं को एक विशेष नमूने के लिए डीएनए या आरएनए अनुक्रम करने के लिए कि क्या चुनना होगा. जेल-seq एक सरल hydrogel डिवाइस का उपयोग कर 100-1000 कोशिकाओं के साथ शुरू डीएनए और आरएनए दोनों के लिए पुस्तकालयों को एक साथ तैयार करने के लिए शोधकर्ताओं को सक्षम करने से इस समस्या का हल. यह कागज डिवाइस के निर्माण के लिए एक विस्तृत दृष्टिकोण के रूप में के रूप में अच्छी तरह से जैविक प्रोटोकॉल युग्मित पुस्तकालयों उत्पंन करने के लिए प्रस्तुत करता है । हम जेल-seq बनाया है ताकि यह आसानी से अंय शोधकर्ताओं द्वारा कार्यांवित किया जा सकता है; कई आनुवंशिकी प्रयोगशालाओं पहले से ही आवश्यक उपकरण जेल-seq डिवाइस निर्माण पुन: पेश करने के लिए है । हमारे प्रोटोकॉल आमतौर पर दोनों पूरी-प्रतिलिपि प्रवर्धन (डब्ल्यूटीए) और पुस्तकालय की तैयारी है, जो भी पहले से ही जीनोमिक और transcriptomic पुस्तकालयों पैदा करने में निपुण शोधकर्ताओं को परिचित होने की संभावना है के लिए इस्तेमाल किया किट कार्यरत हैं । हमारे दृष्टिकोण शोधकर्ताओं ने बंटवारे के बिना और नगण्य जोड़ा लागत के साथ एक एकल नमूना पर दोनों डीएनए और आरएनए अनुक्रमण की शक्ति को सहन करने के लिए लाने के लिए अनुमति देता है.
Introduction
अगली पीढ़ी के अनुक्रमण (NGS) जिस तरह से आनुवंशिकी अनुसंधान आयोजित किया जाता है पर एक गहरा प्रभाव पड़ा है । शोधकर्ताओं ने एक बार एक पूरी प्रजाति के जीनोम sequencing पर ध्यान केंद्रित किया, जहां यह अब एक प्रयोग में एक ही ट्यूमर या यहां तक कि एक सेल के जीनोम अनुक्रम करने के लिए संभव है । 1 NGS भी एक सेल, transcriptome के रूप में जाना जाता डेटा का एक संग्रह के भीतर पाया आरएनए टेप अनुक्रम के लिए यह लागत प्रभावी बना दिया है । छोटे से प्रारंभिक नमूनों से या तो डीएनए या आरएनए को बढ़ाना और अनुक्रम की क्षमता केवल पिछले पांच वर्षों में प्राप्त की गई है. 2 , 3 , 4 दुर्भाग्यवश, मानक प्रोटोकॉल असंगत है और शोधकर्ताओं को यह चुनना होगा कि किसी दिए गए नमूने के लिए डीएनए या आरएनए अनुक्रम करना है या नहीं । जब एक प्रारंभिक नमूना काफी बड़ा है, यह आधा में विभाजित किया जा सकता है । छोटे तराजू पर, हालांकि, बंटवारे के नमूनों के कारण सामग्री की हानि पुस्तकालय की गुणवत्ता को प्रभावित कर सकते हैं, और नमूनों की पूलिंग कोशिकाओं के बीच दिलचस्प भिन्नता बाहर औसत कर सकते हैं. 5 इसके अलावा, शोधकर्ताओं ने तेजी से ऐसे एकल कोशिकाओं या छोटे विषम ट्यूमर बायोप्सी के रूप में, विभाजित नहीं किया जा सकता है कि नमूनों की जांच में रुचि रखते हैं । ६
इस समस्या को हल करने के लिए, तीन प्रोटोकॉल हाल ही में एक ही शुरू नमूना से डीएनए और आरएनए दोनों अनुक्रम के लिए विकसित किया गया है: जेल-seq7, जी एंड टी-seq8, और डॉ-seq9. यह लेख जेल-seq के लिए एक विस्तृत प्रोटोकॉल प्रस्तुत करता है, जो एक साथ के रूप में नगण्य जोड़ा लागत पर 100 कोशिकाओं के रूप में कुछ से डीएनए और आरएनए पुस्तकालयों उत्पंन करने के लिए इस्तेमाल किया जा सकता है । जेल-seq के उपंयास पहलू कम लागत hydrogel मैट्रिक्स का उपयोग कर आकार पर विशेष रूप से आधारित डीएनए और आरएनए अलग करने की क्षमता है । जेल-Seq प्रोटोकॉल के मुख्य नवाचार आरएनए से डीएनए की शारीरिक जुदाई है । इस जुदाई polyacrylamide झिल्ली का एक संयोजन है कि इन अणुओं के बीच आकार मतभेदों का लाभ लेने का उपयोग कर electrophoretically हासिल की है । संदर्भ में इन आकार मतभेद डाल करने के लिए, कैसे डीएनए और आरएनए imaged कर रहे हैं पर विचार करें: जबकि डीएनए माइक्रोन पर मौजूद है पैमाने पर और पारंपरिक सूक्ष्मदर्शी का उपयोग करते हुए देखा जा सकता है, आरएनए नैनोमीटर पैमाने पर मौजूद है और इस तरह के क्रायो के रूप में जटिल तकनीकों का उपयोग imaged होना चाहिए-इलेक्ट्रॉन माइक्रोस्कोपी. 10
इस प्रोटोकॉल में डीएनए और आरएनए को अलग करने का दृष्टिकोण चित्रा 1में दिखाया गया है । बाएँ पैनल डीएनए और आरएनए मुक्त एक झिल्ली के पास समाधान में तैर से पता चलता है. एक बिजली के क्षेत्र में लागू किया जाता है, के रूप में सही पैनल में दिखाया गया है, डीएनए और आरएनए झिल्ली के माध्यम से पलायन लाती है कि एक electrophoretic बल का अनुभव । झिल्ली गुणों ट्यूनिंग द्वारा, हम एक अर्द्ध पारगंय झिल्ली है कि आरएनए से डीएनए अलग बनाया है । डीएनए अणु झिल्ली के खिलाफ धक्का दिया, लेकिन उनके बड़े आकार की वजह से किनारे पर उलझ जाते हैं । दूसरी ओर छोटी आरएनए अणु, पुनः विन्यस्त और झिल्ली के माध्यम से अपने तरीके से बुनाई कर सकते हैं । इस प्रक्रिया, reptation के रूप में जाना जाता है, जिस तरह से एक सांप घास के माध्यम से चलता है के समान है । अंततः इन आरएनए अणुओं के माध्यम से wriggle करने के लिए भी छोटे पॉलिमर (> 200 आधार जोड़े) के लिए भी मुश्किल है कि एक दूसरे, उच्च घनत्व झिल्ली द्वारा बंद कर रहे हैं । एक बार शारीरिक रूप से अलग, डीएनए और आरएनए बरामद किया जा सकता है और दोनों जीनोम और transcriptome के बारे में जानकारी उत्पंन करने के लिए संसाधित । जब तक हम डीएनए और शाही सेना अलग कर सकते हैं, हमने पाया है बेहतर परिणाम प्राप्त कर रहे है अगर आरएनए रिवर्स है जुदाई से पहले सीडीएनए को लिखित । सीडीएनए/आरएनए संकर अकेले आरएनए से अधिक स्थिर हैं और अभी भी कम घनत्व झिल्ली के माध्यम से पारित कर सकते हैं ।
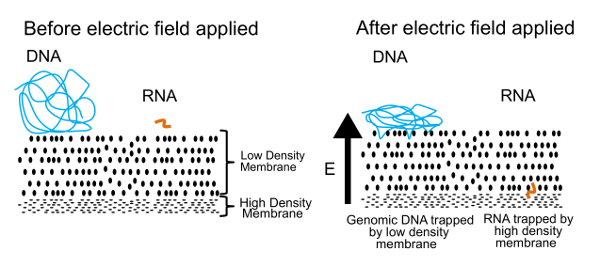
चित्र 1 . जेल-seq ऑपरेटिंग सिद्धांत । अंतर्निहित सिद्धांत शारीरिक रूप से अलग डीएनए और आरएनए के लिए इस्तेमाल किया । एक एप्लाइड इलेक्ट्रिक क्षेत्र में, छोटे आरएनए अणु कम घनत्व झिल्ली के माध्यम से स्थानांतरित लेकिन बड़े डीएनए अणु सतह पर फंस रहे हैं । यह आंकड़ा Ref. 7 से रसायन विज्ञान के रॉयल सोसायटी से अनुमति के साथ reproduced था । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।
इस पत्र में विस्तार से दोनों जेल-seq डिवाइस और जैविक प्रोटोकॉल युग्मित डीएनए और आरएनए पुस्तकालयों उत्पन्न करने के लिए का वर्णन करता है । दोनों का ओवरव्यू आरेख 2में दिखाया गया है । डिवाइस मानक स्टैकिंग जैल बनाने के लिए इसी तरह की एक प्रक्रिया में एक दूसरे के शीर्ष पर तीन अलग घनत्व polyacrylamide जैल लेयरिंग द्वारा गढ़े है । 11 जैव प्रोटोकॉल 100 के साथ शुरू होता है-1000 पंजाब में निलंबित कोशिकाओं । कोशिकाएं लीजड ड हैं और आरएनए को सीडीएनए/आरएनए संकर से जीनोमिक डीएनए को अलग करने के लिए डिवाइस का इस्तेमाल करने से पहले सीडीएनए में बदल दिया जाता है । जुदाई और वसूली के बाद, जीनोमिक और transcriptomic पुस्तकालयों एक प्रक्रिया है कि बारीकी से मानक पूरे जीनोम पुस्तकालय तैयारी किट प्रोटोकॉल इस प्रकार का उपयोग कर तैयार कर रहे हैं । विकास और जेल के सत्यापन के बारे में और अधिक विस्तार-seq एक चिप प्रकाशन “जेल-seq: अर्द्ध पारगंय hydrogel बाधाओं का उपयोग कर एक साथ कम इनपुट डीएनए और आरएनए पुस्तकालय तैयारी द्वारा पूरे जीनोम और transcriptome अनुक्रमण पर प्रयोगशाला में पढ़ा जा सकता है .” 7
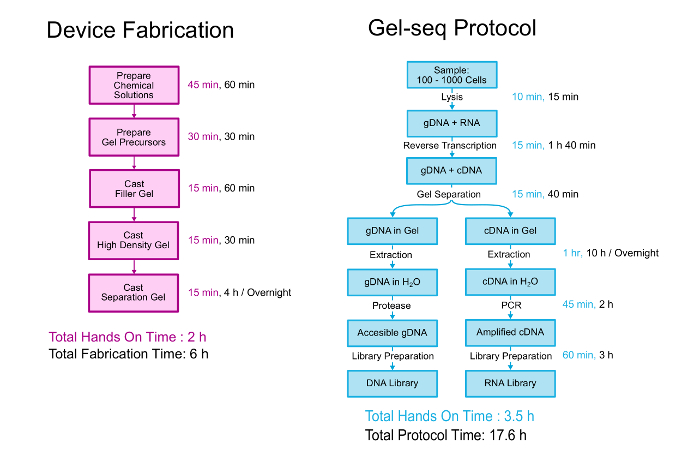
चित्र 2 . जेल-seq प्रोटोकॉल । कदम के एक सिंहावलोकन जेल-seq उपकरण और उत्पंन युग्मित डीएनए और आरएनए पुस्तकालयों के लिए प्रोटोकॉल बनाना । इस आंकड़े के अंश को Ref. 7 से रसायन विज्ञान के रॉयल सोसायटी से अनुमति के साथ reproduced थे । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।
एकल कोशिकाओं से डीएनए और आरएनए पुस्तकालयों उत्पन्न करने के लिए, शोधकर्ताओं को या तो जी एंड टी-seq या डॉ-seq. जी एंड टी-seq, जेल-seq की तरह, जीनोमिक डीएनए से आरएनए के एक भौतिक जुदाई पर निर्भर करता है का उपयोग करने पर विचार करना चाहिए. इस दृष्टिकोण दूत है आरएनए (mRNA) पर निर्भर करता है 3 ‘ एक पुल नीचे लक्ष्य के रूप में polyadenylated पूंछ । mRNA एक biotinylated oligo-डीटी प्राइमर का उपयोग कर एक चुंबकीय मनका पर कब्जा कर लिया है । एक बार mRNA पर कब्जा कर लिया गया है एक चुंबक और जीनोमिक डीएनए युक्त supernatant के साथ जगह में आयोजित कर रहे है और हटाया जा सकता है एक और ट्यूब को हस्तांतरित । इस शारीरिक जुदाई के पूरा होने के बाद, mRNA और डीएनए से अलग पुस्तकालयों उत्पन्न किया जा सकता है । 8 यह दृष्टिकोण अच्छी तरह से काम करता है अगर ब्याज की आरएनए polyadenylated है, लेकिन यह गैर polyadenylated टेप, जैसे राइबोसोमल आरएनए, tRNA, या prokaryotes से आरएनए का अध्ययन करने के लिए उपयोग नहीं किया जा सकता ।
DR-seq एक पूर्व प्रवर्धन कदम पर निर्भर करता है, जहां दोनों डीएनए और सीडीएनए आरएनए से व्युत्पंन एक ही ट्यूब में परिलक्षित कर रहे हैं । नमूना तो दो में विभाजित है और समानांतर में संसाधित करने के लिए डीएनए और आरएनए-seq पुस्तकालयों तैयार. जीनोमिक डीएनए और आरएनए से व्युत्पंन सीडीएनए के बीच अंतर करने के लिए, डॉ-seq एक गणना दृष्टिकोण लेता है. जुगाड़ जहां केवल exons है गणना जीनोमिक डीएनए डेटा में दबा रहे हैं, के रूप में उन या तो डीएनए या आरएनए से उत्पंन किया जा सकता है । 9 इस दृष्टिकोण का एक लाभ यह है कि डीएनए और सीडीएनए/आरएनए को शारीरिक रूप से अलग नहीं किया जाना चाहिए जैसा कि जेल-seq और जी एंड टी-seq में किया जाता है । दोष, तथापि, यह है कि डॉ seq जीनोम और transcriptome (यानी, exons बनाम introns) के एक प्राथमिकताओं ज्ञान की आवश्यकता है, और ऐसे नाभिक के अनुक्रमण के रूप में अनुप्रयोगों के लिए आदर्श नहीं हो सकता है, जिसमें कई टेप अभी तक पूरी तरह से नहीं कर रहे है ब्याह और अभी भी introns होते हैं । 12
जेल-seq के उपंयास पहलू आकार पर विशेष रूप से आधारित कोशिकाओं के सैकड़ों में डीएनए और आरएनए अलग करने की क्षमता है । इस विधि के जीनोम या transcriptome का कोई प्राथमिकताओं ज्ञान की आवश्यकता है, अधूरा ब्याह के खिलाफ मजबूत है, और पाली-adenylated टेप तक ही सीमित नहीं है । अनुप्रयोगों के लिए जहां एक शोधकर्ता के साथ शुरू कर सकते है कम से 100 कोशिकाओं, जेल-seq सस्ते और व्यापक रूप से उपलब्ध सामग्री का उपयोग कर एक सीधा दृष्टिकोण प्रदान करता है ।
Protocol
Representative Results
Discussion
जेल-seq डिवाइस निर्माण के साथ ही प्रोटोकॉल के साथ जुड़े कई महत्वपूर्ण कदम हैं । निर्माण के दौरान, हम जेल के विभिन्न क्षेत्रों के लिए निर्धारित परत मोटाई के साथ शुरू करने की सलाह देते हैं । हम महत्वपूर्ण स?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
इस काम के लिए धन सैन डिएगो, राष्ट्रीय विज्ञान फाउंडेशन स्नातक अनुसंधान फैलोशिप कार्यक्रम, NIH अनुदान R01-HG007836, और कोरियाई विज्ञान मंत्रालय, आईसीटी और भविष्य की योजना के द्वारा विश्वविद्यालय द्वारा प्रदान की गई थी ।
कई आंकड़ों के पहले के संस्करणों “Hoople, जी. डी. एट अल में प्रकाशित किया गया । जेल-seq: अर्द्ध पारगंय hydrogel बाधाओं का उपयोग कर एक साथ कम इनपुट डीएनए और आरएनए पुस्तकालय तैयारी द्वारा पूरे जीनोम और transcriptome अनुक्रमण । एक चिप पर लैब 17, 2619-2630, दोी: 10.1039/c7lc00430c (2017). ” एक चिप पर लैब इस प्रकाशन में आंकड़ों के पुनः प्रयोग को मंजूरी दी है ।
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

