Gel-seq: En metode for samtidige sekvensering biblioteket utarbeidelse av DNA og RNA bruker Hydrogel matriser
Summary
Gel-seq kan forskere samtidig forberede biblioteker for begge DNA – og RNA-seq ubetydelig ekstra kostnad fra 100-1000 celler ved hjelp av en enkel hydrogel enhet. Dette dokumentet presenterer en detaljert tilnærming til fabrikasjon av enheten samt biologiske protokollen for å generere sammenkoblede biblioteker.
Abstract
Muligheten til å forsterke og sekvensielt DNA eller RNA fra små start utvalg er bare oppnådd de siste fem årene. Dessverre standardprotokoller for generering genomisk eller transcriptomic biblioteker er inkompatible og forskere må velge om du vil sekvens DNA eller RNA for en spesiell prøve. Gel-seq løser dette problemet ved å aktivere forskere samtidig forberede biblioteker både DNA og RNA starter med 100-1000 celler ved hjelp av en enkel hydrogel enhet. Dette dokumentet presenterer en detaljert tilnærming til fabrikasjon av enheten samt biologiske protokollen for å generere sammenkoblede biblioteker. Vi designet Gel-seq slik at det lett kan implementeres av andre forskere; mange genetikk labs har allerede det nødvendig utstyret for å reprodusere Gel-seq apparat fabrikasjon. Våre protokollen bruker brukte Kit for både hel-transkripsjon forsterkning (WTA) og biblioteket forberedelse, som også er sannsynlig å være kjent for forskere allerede bevandret i generere genomisk og transcriptomic biblioteker. Vår tilnærming kan forskere å få til å bære kraften i både DNA og RNA sekvensering på et enkelt utvalg uten deling og med ubetydelig ekstra kostnad.
Introduction
Neste generasjons sekvensering (NGS) har hatt en betydelig innflytelse på måten genetikk forskning er utført. Der forskere når fokusert på sekvensering genomet av en hel Art, er det nå mulig å sekvens genomet av en enkelt svulst eller selv en enkeltcelle i ett eksperiment. 1 NGS har også gjort det kostnadseffektivt å sekvens RNA transkripsjoner funnet i en celle, en samling av data kjent som transcriptome. Muligheten til å forsterke og sekvensielt DNA eller RNA fra små start utvalg er bare oppnådd de siste fem årene. 2 , 3 , 4 dessverre standardprotokoller er inkompatible og forskere må velge om du vil sekvens DNA eller RNA for et gitt utvalg. Når en første prøve er stor nok, kan det deles i to. På mindre skalaer, men tap av materiale på grunn av deling prøver kan påvirke biblioteket kvalitet, og av prøver kan jevne ut interessante variasjoner mellom celler. 5 videre forskere er stadig mer interessert i å undersøke prøver som ikke kan deles, for eksempel enkeltceller eller liten heterogene svulst biopsier. 6
For å løse dette problemet, tre protokoller har nylig blitt bebygget for å sekvens både DNA og RNA fra samme Start eksempel: Gel-seq7G & T-seq8og DR-seq9. Denne artikkelen presenterer en detaljert protokoll for Gel-seq, som kan brukes samtidig generere DNA og RNA biblioteker fra så lite som 100 celler ubetydelig ekstra kostnad. Romanen aspekt av Gel-seq er evnen til å skille DNA og RNA basert på størrelse med lavpris hydrogel matriser. I kjernen av Gel-Seq-protokollen er fysisk separasjon av DNA fra RNA. Denne separasjon er oppnådd electrophoretically med en kombinasjon av polyakrylamid membraner som utnytter størrelse forskjellene mellom disse molekyler. For å sette disse størrelse forskjeller i sammenheng, vurdere hvordan DNA og RNA er avbildet: mens DNA finnes på mikro-skala og kan vises ved hjelp av tradisjonelle mikroskop, RNA finnes på nanometer skala og må avbildes bruker komplekse teknikker som cryo-elektron mikroskopi. 10
Tilnærming til skille DNA og RNA i denne protokollen er vist i figur 1. Informasjonsvinduet viser DNA og RNA fri flyt i løsning nær en membran. Når et elektrisk felt brukes, som vist i panelet til høyre, oppleve DNA og RNA en electrophoretic kraft som induserer migrasjon gjennom membranen. Ved å justere egenskapene membran, har vi opprettet et halvt gjennomtrengelig membran som skiller DNA fra RNA. DNA-molekyler er skjøvet mot membranen, men ettersittende ytterst på grunn av den store filstørrelsen. Små RNA molekyler, derimot, kan konfigurere og veve seg gjennom membranen. Denne prosessen kalles reptation, ligner måten en slange beveger seg gjennom gresset. Slutt disse RNA molekyler er stoppet av en andre, høy tetthet membran som er for vanskelig for enda mindre polymerer (> 200 base parene) å vri seg gjennom. Når fysisk skilt, kan DNA og RNA gjenopprettet og behandles for å generere informasjon om både genomet og transcriptome. Mens vi kan skille DNA og RNA, har vi funnet bedre resultater er oppnådd hvis RNA er omvendt transkribert til cDNA før separasjon. CDNA/RNA hybrider er mer stabile enn RNA alene og kan fortsatt passere gjennom lav membran.
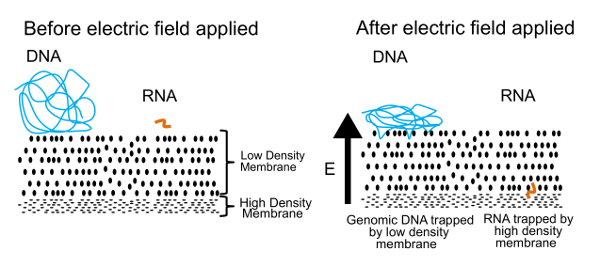
Figur 1 . Gel-seq drift prinsippet. Underliggende prinsippet fysisk mellom DNA og RNA. I en anvendt elektrisk felt, små RNA molekyler overføre gjennom lav membran men store DNA molekyler er fanget på overflaten. Dette tallet ble reprodusert Ref. 7 med tillatelse fra Royal Society of Chemistry. Klikk her for å se en større versjon av dette tallet.
Dette dokumentet beskriver i detalj både fabrikasjon av Gel-seq enheten og biologiske protokollen for å generere sammen DNA og RNA biblioteker. En oversikt over begge vises i figur 2. Enheten er fabrikkert av lagdeling tre forskjellige tetthet polyakrylamid gels oppå hverandre i en prosess som ligner til å skape standard stabling gels. 11 biologiske protokollen starter med 100-1000 celler suspendert i PBS. Cellene er lysed og RNA konverteres til cDNA før enheten brukes til å skille genomisk DNA fra cDNA/RNA hybrider. Etter separasjon og utvinning, genomisk og transcriptomic lages biblioteker av en prosess som følger tett hele-genome standardbiblioteket forberedelse kit protokollen. Ytterligere detaljer om utvikling og validering av Gel-seq kan leses i laboratoriet med en Chip publikasjon “Gel-seq: hele-genom og transcriptome sekvensering av samtidige lav-input DNA og RNA biblioteket forberedelse på halvt gjennomtrengelig hydrogel barrierer .” 7
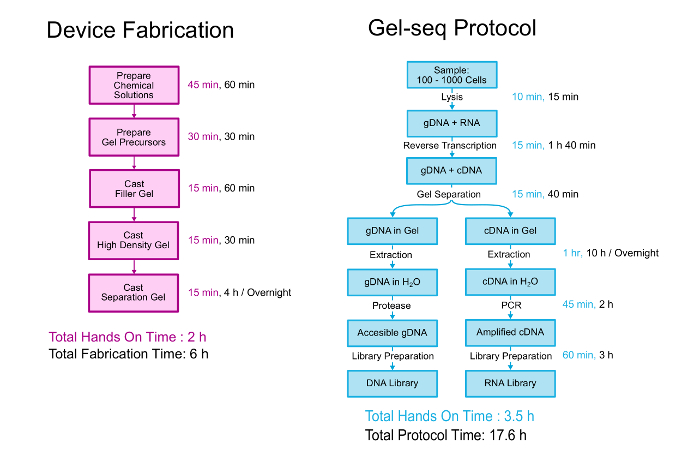
Figur 2 . Gel-seq protokollen. En oversikt over trinnene for å dikte Gel-seq enheten og protokollen generert sammen DNA og RNA biblioteker. Deler av dette tallet ble reprodusert Ref. 7 med tillatelse fra Royal Society of Chemistry. Klikk her for å se en større versjon av dette tallet.
Vil generere DNA og RNA biblioteker fra enkeltceller, forskere bør vurdere å bruke enten G & T-seq eller DR-seq. G & T-Seq, som Gel-seq, avhengig av en fysisk separasjon av fra genomisk DNA. Denne tilnærmingen er avhengig av budbringer RNA (mRNA) 3 polyadenylated halen som rullegardin mål. MRNA fanges opp på en magnetisk perle ved hjelp av en biotinylated oligo-dT primer. Når mRNA har blitt fanget perlene holdes på plass med en magnet og nedbryting inneholder genomisk DNA kan fjernet og overført til en annen tube. Når denne fysisk separasjon er fullført, kan separat biblioteker genereres fra mRNA og DNA. 8 denne tilnærmingen fungerer godt hvis RNA rundt polyadenylated, men den kan ikke brukes til å studere ikke-polyadenylated utskrifter, for eksempel ribosomal RNA, tRNA eller RNA fra prokaryoter.
DR-seq er avhengig av en før forsterkning trinn der både DNA og cDNA avledet fra RNA er forsterket i samme rør. Prøven er deretter delt i to og behandles parallelt å forberede DNA og RNA seq biblioteker. Du kan skille mellom genomisk DNA og cDNA avledet fra RNA, tar DR-seq en beregningsorientert tilnærming. Sekvenser hvor bare exons finnes undertrykt beregningsmessig i genomisk DNA data, som de kan ha sin opprinnelse fra DNA eller RNA. 9 en fordel med denne tilnærmingen er at DNA og cDNA/RNA ikke trenger være fysisk atskilt som er gjort i Gel-seq og G & T-seq. Ulempen er imidlertid at DR-seq krever en priori kunnskap om genomet og transcriptome (dvs., exons versus introns), og kan ikke være perfekt for programmer som sekvensering av kjerner, der mange transkripsjoner ikke er ennå fullt skjøtes og fremdeles inneholde introns. 12
Romanen aspekt av Gel-seq er muligheten til å skille DNA og RNA i hundrevis av cellene basert på størrelse. Denne metoden krever ikke en priori kunnskap om genomet eller transcriptome, er robuste mot ufullstendig skjøting, og er ikke begrenset til poly-adenylated utskrifter. For applikasjoner der en forsker kan starte med minst 100 celler, gir Gel-seq en enkel tilnærming med billig og lett tilgjengelig.
Protocol
Representative Results
Discussion
Det er flere viktige trinn forbundet med Gel-seq apparat fabrikasjon samt protokollen. Under fabrikasjon anbefaler vi starter med de foreskrevne Sjikttykkelse for de ulike regionene i gel. Vi brukte betydelig tid tester forskjellige fabrikasjon alternativer og protokollen beskrevet her gir de beste enhetene for kassettene oppført i tabell av materialer og reagenser. Hvis forskerne bruker en alternativ kassett-system, kan de finne det nødvendig å justere volumene brukes ved opprettelse av enhetene. Den…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Finansiering for dette arbeidet var forutsatt ved Universitetet i San Diego, National Science Foundation Graduate forskning Fellowship Program, NIH gi R01-HG007836, og av koreansk vitenskap, IKT og fremtid planlegger.
Tidligere versjoner av en flere tall ble første gang publisert i “Hoople, G. D. et al. Gel-seq: hele-genome og transcriptome sekvensering av samtidige lav-input DNA og RNA biblioteket forberedelse på halvt gjennomtrengelig hydrogel barrierer. Lab på en Chip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). ” Lab på en Chip har godkjent gjenbruk av tallene i denne publikasjonen.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

