Gel-seq: En metod för samtidig sekvensering bibliotek beredning av DNA och RNA med hjälp av Hydrogel matriser
Summary
Gel-seq gör det möjligt för forskare att samtidigt förbereda bibliotek för både DNA – och RNA-seq försumbar extra kostnad från 100-1000 celler använder en enkel hydrogel-enhet. Detta dokument presenterar en detaljerad strategi för tillverkning av enheten samt biologiska protokollet att generera Parade bibliotek.
Abstract
Förmågan att förstärka och antingen DNA eller RNA-sekvens från små start prover har bara uppnåtts under de senaste fem åren. Tyvärr standardprotokollen för att skapa genomisk eller transcriptomic bibliotek är oförenliga och forskare måste välja om att sekvensera DNA eller RNA för ett visst urval. Gel-seq löser detta problem genom att möjliggöra för forskare att samtidigt förbereda bibliotek för både DNA och RNA börjar med 100-1000 celler använder en enkel hydrogel-enhet. Detta dokument presenterar en detaljerad strategi för tillverkning av enheten samt biologiska protokollet att generera Parade bibliotek. Vi utformade Gel-seq så att det enkelt kunde genomföras av andra forskare; många genetik labs har redan nödvändig utrustning för att reproducera Gel-seq enhet tillverkning. Våra protokoll sysselsätter vanliga kit för både hela-transcript amplifiering (WTA) och bibliotek förberedelse, som sannolikt också kan vara bekant för forskare redan insatt i generera genomisk och transcriptomic bibliotek. Vår strategi tillåter forskare att utöva kraften i både DNA och RNA-sekvensering på ett enda prov utan uppdelning och med obetydlig merkostnad.
Introduction
Nästa generation sequencing (NGS) har haft en djupgående inverkan på det sättet genetik forskning bedrivs. Där forskare en gång fokuserat på sekvensering genomet hos en hela arter, är det nu möjligt att sekvensera genomet hos en enda tumör eller ens en enda cell i ett experiment. 1 NGS har också gjort det kostnadseffektivt att sekvensera RNA avskrifter finns inom en cell, en samling av data kallas transkriptom. Förmågan att förstärka och antingen DNA eller RNA-sekvens från små start prover har bara uppnåtts under de senaste fem åren. 2 , 3 , 4 tyvärr standardprotokollen är oförenliga och forskare måste välja om att sekvensera DNA eller RNA för ett givet prov. När ett start prov är tillräckligt stor, kan det delas i två halvor. På mindre skalor, men förlust av material på grund av uppdelning prover kan påverka bibliotek kvalitet och sammanslagning av prover kan i genomsnitt ut intressanta variationer mellan celler. 5 dessutom forskare är allt mer intresserade av att undersöka prover som inte kan delas, såsom enstaka celler eller små heterogena tumör biopsier. 6
För att lösa detta problem, tre protokollen har nyligen utvecklats för att både DNA och RNA-sekvens från samma start prov: Gel-seq7, G & T-seq8och DR-seq9. Denna artikel presenterar en detaljerad protokoll för Gel-seq, som kan användas samtidigt generera DNA och RNA bibliotek från så lite som 100 celler försumbar extra kostnad. Den nya aspekten av Gel-seq är förmågan att separera DNA och RNA baseras uteslutande på storlek med låg kostnad hydrogel matriser. Core innovation av Gel-Seq protokollet är den fysiska separationen av DNA från RNA. Denna separation uppnås electrophoretically med en kombination av polyakrylamid membran som drar nytta av storleksskillnaderna mellan dessa molekyler. För att sätta dessa storlek skillnader i sammanhang, överväga hur DNA och RNA är avbildade: medan DNA finns på micron-skalan och kan visas med traditionella Mikroskop, RNA finns på nanometerskalan och måste avbildas med komplexa tekniker såsom cryo-elektron mikroskopi. 10
Metoden att avskilja DNA och RNA i detta protokoll visas i figur 1. Den vänstra panelen visar DNA och RNA fri svävande i lösningen nära ett membran. När ett elektriskt fält appliceras, som visas i den högra panelen, uppleva DNA och RNA en elektrofores kraft som inducerar migration genom membranet. Genom att trimma membran egenskaper, har vi skapat ett halvgenomträngligt membran som skiljer DNA från RNA. DNA-molekylerna skjuts mot membranet, men fastnar på kanten på grund av deras storlek. Små RNA-molekyler, däremot, kan konfigurera om och väver sig igenom membranet. Denna process, som kallas reptation, är ungefär på samma sätt som en orm som rör sig genom gräset. Så småningom dessa RNA-molekyler stoppas av ett andra, hög densitet membran som är för svår för ännu mindre polymerer (> 200 baspar) slingra sig igenom. När fysiskt åtskilda, kan DNA och RNA återvinnas och behandlas för att generera information om både genomet och transkriptom. Medan vi kan separera DNA och RNA, har vi hittat bättre resultat erhålls om RNA är omvänd transkriberas till cDNA före separationen. CDNA/RNA hybrider är mer stabila än RNA ensam och kan fortfarande passera genom låg densitet membranet.
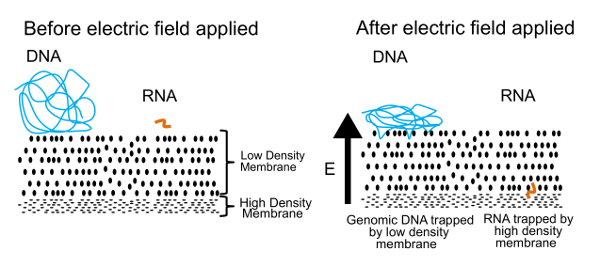
Figur 1 . Gel-seq operativa principen. Den underliggande principen används för att fysiskt skilja DNA och RNA. I en tillämpad elektriskt fält, små RNA-molekyler vandrar genom låg densitet membranet men stor DNA-molekyler är instängd på ytan. Denna siffra var återges från Ref. 7 med tillstånd från den Royal Society of Chemistry. Klicka här för att se en större version av denna siffra.
Detta dokument beskriver i detalj både tillverkning av Gel-seq enheten och biologiska protokollet att generera parat DNA och RNA bibliotek. En översikt av båda visas i figur 2. Enheten är tillverkad av skiktning tre olika densitet polyakrylamidgeler ovanpå varandra i en process som påminner om att skapa standard stapling geler. 11 biologiska protokollet börjar med 100-1000 celler upphängd i PBS. Cellerna är lyserat och RNA omvandlas till cDNA innan enheten används för att separera genomisk DNA från cDNA/RNA hybrider. Efter separation och återhämtningen, genomisk och transcriptomic bibliotek är beredda med en process som noggrant följer protokollet helgenom-standardbiblioteket förberedelser kit. Ytterligare information om utveckling och validering av Gel-seq kan läsas i labbet på en Chip publikation ”Gel-seq: helgenom- och transkriptom sekvensering av samtidiga lågintensivt DNA och RNA tillredning bibliotek via ljusgenomsläppande hydrogel hinder .” 7
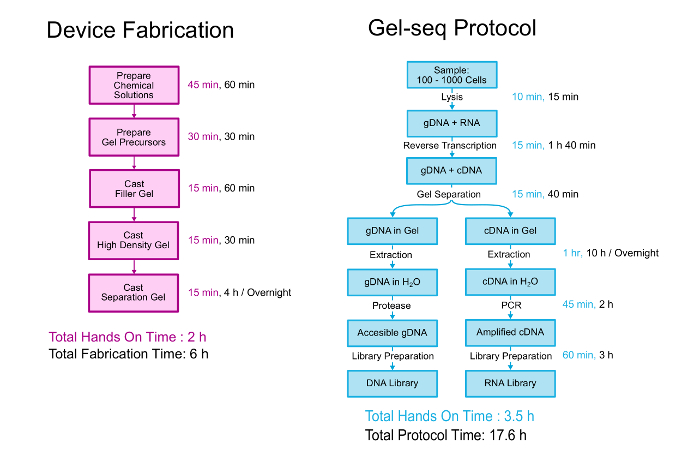
Figur 2 . Gel-seq protokollet. En översikt över stegen för att fabricera Gel-seq enheten och protokollet till genererade Parade DNA och RNA bibliotek. Delar av denna siffra reproducerades från Ref. 7 med tillstånd från den Royal Society of Chemistry. Klicka här för att se en större version av denna siffra.
För att generera DNA och RNA bibliotek från enstaka celler, forskare bör överväga att använda antingen G & T-seq eller DR-följande punkter G & T-Seq, som Gel-seq, bygger på en fysisk separation av RNA från genomiskt DNA. Denna strategi bygger på messenger RNA (mRNA) 3′ polyadenylated svans som nedrullningsbara mål. MRNA fångas på en magnetisk pärla med en biotinylerade oligo-dT primer. När mRNA har fångats pärlor hålls på plats med en magnet och supernatanten innehållande genomisk DNA kan tas bort och överföras till en annan tube. Efter denna fysiska separation är komplett, kan separata bibliotek genereras från mRNA och DNA. 8 detta tillvägagångssätt fungerar bra om RNA av intresse är polyadenylated, men det kan inte användas att studera icke-polyadenylated avskrifter, såsom ribosomalt RNA, tRNA eller RNA från prokaryoter.
DR-seq åberopat ett före förstärkning steg där både DNA och cDNA härrör från RNA förstärks i samma rör. Provet är sedan uppdelad i två och bearbetas parallellt att förbereda DNA – och RNA-seq bibliotek. För att skilja mellan genomiskt DNA och det cDNA som härrör från RNA, tar DR-seq en computational förhållningssätt. Sekvenser där det förekommer endast exoner undertrycks beräkningsmässigt i det genomiska DNA-uppgifter, som de kunde ha sitt ursprung från antingen DNA eller RNA. 9 en fördel med denna metod är att den DNA och cDNA/RNA inte behöver vara fysiskt åtskilda som sker i Gel-seq och G & T-följande punkter Nackdelen, är dock att DR-seq kräver en priori kunskap av genomet och transkriptom (dvs, exoner och introner), och kanske inte idealisk för applikationer såsom sekvensering av atomkärnor, där många avskrifter inte är ännu helt skarvas och fortfarande innehåller introner. 12
Den nya aspekten av Gel-seq är förmågan att separera DNA och RNA i hundratals celler uteslutande utifrån storlek. Denna metod kräver ingen förhand kunskap av genomet eller transkriptom, är robust mot ofullständiga skarvning och är inte begränsat till poly-adenylated avskrifter. För applikationer där forskare kan börja med minst 100 celler, ger Gel-seq en enkel metod med billigt och allmänt tillgängliga material.
Protocol
Representative Results
Discussion
I området i närheten finns det flera kritiska steg som är associerad med Gel-seq enhet tillverkning samt själva protokollet. Under tillverkning rekommenderar vi att du börjar med de föreskrivna skikttjocklekar för de olika regionerna av gelen. Vi tillbringade betydande tid testa olika fabrication alternativ och protokollet beskrivs här producerar de bästa enheterna för de kassetter som anges i tabell av material och reagens. Om forskare använder en alternativ kassettsystem, kan de finner det n…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Finansiering för detta arbete var förutsatt av universitetet i San Diego, National Science Foundation Graduate Research Fellowship Program, NIH bevilja R01-HG007836, och av koreanska ministeriet för vetenskap, IKT och framtida planering.
Tidigare versioner av en flera siffror först publicerades i ”Hoople, G. D. et al. Gel-seq: helgenom- och transkriptom sekvensering av samtidiga lågintensivt DNA och RNA tillredning bibliotek via ljusgenomsläppande hydrogel hinder. Lab på ett Chip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017) ”. Lab på ett Chip har sanktionerat återanvändning av uppgifterna i denna publikation.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

