Kalibrering-gratis In Vitro kvantifiering av Protein Homo-oligomerisering använder kommersiella instrumentering och fri, öppen källkod ljusstyrka analys programvara
Summary
Det här protokollet beskriver en kalibrering-fri metod för att kvantifiera protein homo-oligomerisering in vitro- baserat på fluorescens fluktuation spektroskopi med kommersiella ljus skanning mikroskopi. Rätt förvärvet inställningar och analysmetoder visas.
Abstract
Antal och ljusstyrka är en kalibrering-fri fluorescens fluktuation spektroskopi (FFS) teknik för att upptäcka protein homo-oligomerisering. Det kan användas med konventionella confocal Mikroskop utrustade med digitala detektorer. Ett protokoll för användning av teknik i in vitro- visas med hjälp av ett användningsfall där nummer och ljusstyrka kan ses att exakt kvantifiera Oligomera delstaten mVenus-märkt FKBP12F36V före och efter tillsats av dimerizing drogen AP20187. Vikten av att använda rätta Mikroskop förvärv parametrarna och metoderna rätt data förbehandling och analys diskuteras. Särskilt betonas vikten av valet av fotoblekning korrigering. Här billig metod kan användas för att studera protein-protein interaktioner i många biologiska sammanhang.
Introduction
Protein-protein interaktioner jag n Vitro
Traditionellt, är kristallografi och magnetiska kärnresonans-experiment kombinerat med kryo-elektronmikroskopi (kryoEM) den teknik som valt att exakt beskriva de tredimensionella arkitekturen av proteiner och att härleda deras funktion av granskar deras strukturella Detaljer för hög upplösning. Proteiner, dock är inte statiska strukturer och kan genomgå en mängd konfirmerande förändringar och vibrationer i tid och rum. Därför strukturell information från kristallografiska eller kryoEM data behöver kompletteras med andra tekniker (t.ex., molekyldynamik simuleringar och enda molekyl tekniker): funktionen av ett protein som är relaterad till dess konfirmerande förändringar och interaktioner, och denna information är inte närvarande i en statisk struktur. För att probe för transaktioner inom molekylär dynamik, är tekniker baserat på enda molekyl Forster resonans överföra energi (smFRET) mycket effektiv1. Dessa metoder ska kunna bedöma olika subpopulations av molekyler i komplexa media. Detta är mycket viktigt, eftersom dessa förändringar är snabb och uppstå under förvärv av data (d.v.s., nanosekund till andra intervallet).
Två huvudsakliga metoder är vanligen anställda att påvisa och kvantifiera dessa förändringar: proteiner i lösning och surface-immobilisering. För detektion av Inter molekylära interaktioner och i synnerhet, processen för dimerization induceras av ligander, är smFRET inte alltid det bästa verktyget. Verkligen GRÄMA beror inte bara på avståndet (≈10 nm), men också om inriktningen på de två dipolerna (givare och acceptor, χ2) och överlappningen av givare utsläpp med den acceptor Absorptionsspektra2, men kanske detta sista villkor är mindre under förutsättning att experimentalist kan valde viktigt rätt bandet paret. Särskilt missgynnar av smFRET för avsökning av homo-dimerization kommer från märkning av proteinet av intresse: för Straighta smFRET, dimerization endast kan upptäckas upp till 50% (dvs, hetero-bandet kommer endast att kunna upptäcka givare-acceptor och acceptor-givare homo-dimerer men inte givare-givare eller acceptor-acceptor, som är de andra 50% av dimerer). Användning av fluorescens korrelation spektroskopi (FCS) och derivat (S.K., etc.3) att fastställa protein diffusion konstanter och bindande konstanter i vitro är ett annat alternativ. Dessa metoder kan inte helt kvantifiera homo-dimerization antingen, som i FCS en åtgärder koncentration och diffusion, och radie och diffusion koefficient av en diffuserande partikel är mycket dåligt beroende av den molekylära vikten; till exempel innebär en 10-faldig ökning i molekylvikt endast en 2.15 faldig förändring i diffusion koefficient4. Vid två-färg FCS eller S.K., kommer endast 50% av homo-dimerer att ses av samma anledning som ovan. Den mest praktiska och kvantitativa metoder för att detektera homo-dimerization in vitro- och in-vivo är homo-bandet5 och nummer och ljusstyrka (N & B)6. Med tanke på faktumen att homo-bandet kräver särskild instrumentation återställning av anisotropi värdet (dvs. optiska element/analysatorer till återvinna den parallella och vinkelräta polariseringen), N & B presenteras här som en god teknik för att upptäcka protein homo-dimerization och aggregering. Det kan vara sysselsatt både in vitro- och in-vivo med en kommersiell set-up.
Antal och ljusstyrka
N & B har varit nyligen granskade7. Granskningen fokuserar på tillämpningen av tekniken i levande celler. Det är värt att återge matematisk formalism här som dessa ekvationer kommer att tillämpas på data som samlats in i vitro. Det första är det nödvändigt att definiera vissa termer och matematiska kvantiteter:
- En entitet är en uppsättning av molekyler som är sammanfogade.
- Den ljusstyrka ε för en entitet är antalet fotoner som den avger per tidsenhet (per bildruta).
- n är antalet enheter närvarande.
- För en viss pixel under loppet av en bild serie är dess genomsnittliga intensitet och σ2 är variansen i dess intensitet.
Sedan, med fotonräknande detektorer och förutsatt att mobila enheter och ingen bakgrund,
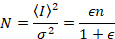
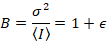
där N är den uppenbara nummer och B är skenbar ljusstyrka. Detta resulterar i
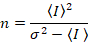
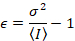
Dalal o.a. 8 visade att med analog utrustning, man behöver tre korrektionsuttryck: S factor, bakgrunden offset, och utslaget buller σ02. Sedan igen förutsatt att mobila enheter,
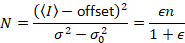
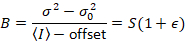
att ge
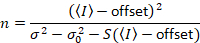
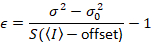
Observera att ovanstående ekvation för skiljer som ges i Dalal o.a. 8 och en efterföljande omprövning. 7 i Dalal et al. S i nämnaren utelämnades på grund av ett stavfel och felet reproducerades i översynen. Ekvationen ovan är rätta. Instruktioner för mätning av S, offset och σ02 – tillsammans med en förklaring av deras innebörd – ges av Dalal o.a. 8
Den ljusstyrka ε är proportionell mot Oligomera staten sprida enheter: ε kommer vara dubbelt så stor för dimerer som för monomerer, tre gånger så stor för trimerer som för monomerer, dubbelt så stor för hexamers som för trimerer och så vidare. På detta sätt mäta den ljusstyrka ε, kan man kvantifiera någon typ av multimerization.
Om det finns en blandning av Oligomera stater närvarande, antal och ljusstyrka är inte klarar av att återställa enskilda Oligomera stater närvarande. Detta är en begränsning för tekniken.
Detrend algoritm och nandb programvara
Vikten av att korrigera för fotoblekning har betonat tidigare9. Fotoblekning oundvikligen inträffar under ljusmikroskop experiment i time-lapse läget. både i levande celler och in vitro. Många metoder har beskrivits i litteraturen att korrigera för blekning7. Den exponentiella filtrering tekniken med automatisk val av detrending parametern T är den nuvarande bäst. Det är integrerat i fri, öppen källkod programvara nandb9. Faktiskt, programvara som kräver att användaren manuellt välja deras detrending parametern kan leda till felaktiga resultat eftersom denna parameter val kommer sannolikt att vara godtycklig och felaktig. Den automatiska algoritmen inspekterar data och bestämmer lämpliga parametern för det, utan att användaren ingripande9. Även med det bästa valet av utjämnande parameter, detrending har sina begränsningar och fungerar väl bara med fotoblekning procentandelar lägre än 25%, som visas med simuleringar9. Intressant, när du använder den automatiska detrending rutinen, dess riktighet är sådan att man kan arbeta med låg ljusstyrkevärden (även B < 1,01), och därmed låg intensitet, och fortfarande vara tillräckligt exakta för att kvantifiera homo-dimerization.
Fotoblekning orsakar också ett annat problem: förekomst av photobleached fluorophores i en komplex multimer. Detta gör exempelvis en trimer visas som en dimer när en av de tre enheterna i trimer är icke-fluorescerande. Hur och Mueller10 visade hur att rätta till detta och denna korrigering betonades också i en senare översyn7. Nandb programvaran innehåller denna korrigering9.
FKBP12 F36V systemet
FKBP12F36V är ett protein som inte naturligt oligomerize men det är känt att dimerize vid tillsats av AP20187 drogen (colloquially kallas BB dimerizing ligand)11,12. Detta gör det en perfekt testfall för nummer och ljusstyrka: med märkta FKBP12F36V, en fördubbling av Oligomera staten bör observeras vid tillägg av BB.
Protocol
Representative Results
Discussion
N & B är en teknik för att upptäcka multimerization med kommersiella ljus skanning confocal Mikroskop utrustade med digitala detektorer. Detta tillvägagångssätt är ganska attraktivt jämfört enda punkt FCS, S.K. och smFRET eftersom det är kalibrering gratis och ljusstyrka beräkningen är okomplicerad och koncentration oberoende6. Det är av stor vikt, dock att korrigera för blekning och långsiktiga intensitet fluktuationer innan du utför ljusstyrka beräkningar9</sup…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Detta arbete har stötts av Wellcome Trust bevilja R.N. 105278/Z/14/2 Wellcome Trust Centre for Human Genetics finansieras av Wellcome Trust Core Award 203852/Z/16/2. Arbetet i gruppen C.S. stöds av Cancer Research UK (C20724/A14414) och Europeiska forskningsrådet under EU: s Horizon 2020 forskning och Innovation-programmet Grant 647278.
Materials
| RosettaTM (DE3) pLysS cells | Novagen | 70956-3 | |
| Ampicillin | Sigma Aldrich | PubChem Substance ID 329824407 | |
| Chloramphenicol | Sigma Aldrich | PubChem Substance ID: 24892250 | |
| LB starter culture | QIAGEN | ||
| LB medium | QIAGEN | https://www.sigmaaldrich.com/content/dam/sigma-aldrich/head/search/external-link-icon.gif | |
| IPTG | Sigma Aldrich | PubChem Substance ID 329815691 | |
| IMAC buffer | Medicago | 09-1010-10 | |
| EDTA-free protease inhibitors | Sigma Aldrich | 11873580001 | |
| TALON resin | Clonetech | ||
| Nickel sepharose | GE Healthcare | ||
| S200 16/60 column | GE Healthcare | ||
| Glass bottom 8 well observation dish | Ibidi | 80827 |
References
- Voith von Voithenberg, L., Lamb, D. C. Single pair forster resonance energy transfer: A versatile tool to investigate protein conformational dynamics. BioEssays. 40, (2018).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Dual-color fluorescence lifetime correlation spectroscopy to quantify protein-protein interactions in live cell. Microscopy Research and Technique. 74, 788-793 (2011).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Tramier, M., Coppey-Moisan, M. Fluorescence anisotropy imaging microscopy for homo-FRET in living cells. Methods in Cell Biology. 85, 395-414 (2008).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94, 2320-2332 (2008).
- Nolan, R., Iliopoulou, M., Alvarez, L., Padilla-Parra, S. Detecting protein aggregation and interaction in live cells: A guide to number and brightness. Methods. , (2017).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71, 69-81 (2008).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. , (2017).
- Hur, K. H., et al. Quantitative Measurement of Brightness from Living Cells in the Presence of Photodepletion. PLoS One. 9, (2014).
- Amara, J. F., et al. A versatile synthetic dimerizer for the regulation of protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 94, 10618-10623 (1997).
- Clackson, T., et al. Redesigning an FKBP-ligand interface to generate chemical dimerizers with novel specificity. Proceedings of the National Academy of Sciences of the United States of America. 95, 10437-10442 (1998).
- Rollins, C. T., et al. A ligand-reversible dimerization system for controlling protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 97, 7096-7101 (2000).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82, 518-529 (2015).
- . R: A Language and Environment for Statistical Computing. R Core. , (2017).
- . RStudio: Integrated Development Environment for R. R Team. , (2016).
- . nandb R package Available from: https://CRAN.R-project.org/package=nandb (2017)
- Jung, G., Wiehler, J., Zumbusch, A. The photophysics of green fluorescent protein: influence of the key amino acids at positions 65, 203, and 222. Biophysical Journal. 88, 1932-1947 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2, 905-909 (2005).
- Butkevich, A. N., et al. Hydroxylated fluorescent dyes for live-cell labeling: synthesis, spectra and super-resolution STED. Chemistry: A European Journal. 23, 12114-12119 (2017).

