Live celle analyse av skjæring Stress på Pseudomonas aeruginosa bruker en automatisert høyere gjennomstrømming Microfluidic System
Summary
Her beskriver vi bruk av en høyere gjennomstrømming microfluidic bioreactor kombinert med fluorescerende mikroskop for analyse av skjæring stress effekter på Pseudomonas aeruginosa biofilm uttrykke grønne fluorescerende proteiner, inkludert instrument sette opp, bestemmelse av biofilm dekning, vekst og morfologiske egenskaper.
Abstract
En høyere gjennomstrømming microfluidic i vitro bioreactor kombinert med fluorescens mikroskopi er brukt til å studere bakteriell biofilm vekst og morfologi, inkludert Pseudomonas aeruginosa (P. aeruginosa). Her vil vi beskrive hvordan systemet kan brukes til å studere vekst kinetics og morfologiske egenskaper som overflateruhet og tekstur entropi P. aeruginosa belaste PA01 som uttrykker en forbedret grønne fluorescerende protein (PA01-EGFP ). En protokoll som detaljert beskriver hvordan du vokse og frø PA01-EGFP kulturer, hvordan du setter opp mikroskop og autorun, og gjennomføre bildeanalyse for å finne veksten og morfologiske egenskaper ved hjelp av en rekke skjær krefter som styres av den microfluidic enhet. Denne artikkelen gir en detaljert beskrivelse av en teknikk for å forbedre studiet av PA01-EGFP biofilm som til slutt kan brukes mot andre stammer av bakterier, sopp og alger biofilm ved hjelp av microfluidic-plattformen.
Introduction
Her viser vi en metode for å måle effekten av skjæring stress av fluorescerende Pseudomonas aeruginosa (P. aeruginosa) PA01 biofilm bruker en automatisert høyere gjennomstrømming microfluidic system.
Biofilm er samfunn av mikroorganismer, som bakterier, organisert av en ekstracellulære polymere stoff som er knyttet til en støtte og finnes vanligvis på grensesnittet mellom en væske og en solid overflate1. Disse biofilm samfunn kan være gunstig for miljøet, som bedre vannkvaliteten i vann- og bioremediation av gjenstridige forbindelser2,3. Men kan biofilm også være svært skadelig for helse med uønskede konsekvenser. For eksempel er medisinske enheter, for eksempel hofte og kne implantater, en type overflate der biofilm akkumulering har vært en utfordring og forårsaker alvorlige medisinske komplikasjoner4,5. Biofilm kan også angi naturlige vann systemer, som elver og innsjøer, og infiltrere vannforsyning rør fører til bakterier forurensning i drikkevannet resulterer i infeksjoner6,7,8. Biofilm dannet marint overholde skip og andre menneskeskapte underlag og presentere et stort økonomisk og miljømessig problem som økt friksjon fører til øke drivstoff forbruk9,10. Antimikrobielle belegg, som Tributyltin, har blitt utviklet for å hindre at disse problemene, men er giftig for sjøliv11.
P. aeruginosa er en Gram-negative bakterie med høy blomstrende evner i en rekke miljø-og nutrimental12. P. aeruginosa er en vanlig årsak til samfunnet – og sykehus-infeksjoner ervervet og funnet å være tett knyttet til skader, som alvorlige forbrenninger og immunsupprimerte verter, som i cystisk fibrose (CF)5,12, 13, AIDS og kreft pasienter5,13. Dannelsen av P. aeruginosa biofilm er mest alvorlig koblet til CF, der kroniske lunge infeksjoner er den ledende dødsårsaken for denne sykdommen5.
En referanse stamme av P. aeruginosa, PA01, brukes i denne rapporten og er genetisk modifisert for å uttrykke en forbedret grønne fluorescerende protein (PA-EGFP). EGFP representerer en mutant form for GFP med større fluorescens egenskaper som tillater i situ biofilm analyse med fluorescens mikroskopi14,15,16. Denne typen fluorescens analyse er fordelaktig for å studere biofilm fordi GFP ikke påvirke betydelig cellevekst og funksjonen17. For eksempel vokste Escherichia coli cellene som ble merket med GFP også og kontinuerlig uten å ha lidd noen toksiske effekter i forhold til de kontroll bakterier17. Andre rapporter underbygge denne påstanden18,19,20. Videre bruk av fluorescerende reporter som EGFP er rask og enkel, men bare lever celler vil bli målt fordi døde celler raskt slutte å fluoresce21.
Biofilm kan vokse under ulike miljøforhold, inkludert de med forskjellige strømningshastigheter. For eksempel kan filmer vokse i høy skjæring stress, som i elver, hvor den høy vann strømningsforhold føre til en større mikrobiell mangfold22. Contrarily, stående vann dammer eller muntlig biofilm oppleve en mye lavere skjær force23. I tillegg til strømningsrate, det er andre faktorer som påvirker biofilm vedheft, inkludert overflateruhet og hydrophobicity, media komposisjon, og selv bakterielle celler overflaten1,4,7, 24. forhold kan også forårsake variasjon i romlig struktur eller morfologi av en biofilm. Dette inkluderer miljøforhold som skjæring stress utøves av en bevegelse væske eller graderinger i nærings tilgjengelighet og biologiske faktorer som arter til stede i systemet og motilitet av celler spesifikke proteiner tilstede i den ekstracellulære polymere stoff25,26,27. I enkelte tilfeller biofilm blir plen-lignende (jevn og flat), stund andre vilkår i biofilm vil være grov, LUN eller selv mushroom-lignende28. Mens kvalitativ forskjellen biofilm plener og sopp strukturer kan sees tydelig i mikroskopiske bilder, krever forstå forholdet mellom filmen struktur og biologiske prosesser i filmen systematisk og kvantitativ metoder for å beskrive morfologi. Morfologiske egenskaper foreslått for studie av forskere inkluderer porøsitet, fraktal dimensjon, diffusional lengde, microcolony området på undergrunnen, microcolony kvantum, grovheten koeffisient og tekstur entropi29,30 .
Bioreaktorer brukes i studiet av biofilm for å etterligne virkelige forhold31. Drypp flyt reaktorer (DFR) representerer en lav-skjær miljø der næringsstoffer i media flyter langsomt over celler som er knytte til en overflate over tid å danne en biofilm med et høyt tetthet32. CDC reaktorene er bioreaktorer oppretter en høy skjæring stress flytende miljø ved kontroll av en gripende stang som kontinuerlig roterer i media fylt tank33. Disse typer bioreaktorer er enkelt å sette opp, men de er begrenset i omfang på grunn av relativt lav utvalgsstørrelsen, høyt inntak av media, store mengder biologisk farlige avfall produsert media drypp renner fra 125 µL/minutt for drypp flyt reaktorer til mer enn 1 mL/min for CDC reaktorer og måtte autoklav store mengder glass og avfall media34. Biofilm vokser ikke jevnt over overflaten på drypp flyt reaktoren fordi lav skjær av media forårsaker etterfølgende langs de større konglomerater P. aeruginosa bakterier derfor biofilm veksten er ikke veldig glatt og ujevn prøvene ikke lett analysert med fluorescens mikroskopi35,36 .
Noen vanlige bioreactor begrensninger er overvunnet ved hjelp av et medium gjennomstrømming microfluidic bioreactor, der bare ml av media er nødvendig, og reaksjonen platene er liten og lett disponibel etter autoklavering37. Videre, avhengig av antall brønner, mange replikeringer kan utføres i en reaktor kjører, som gir tilstrekkelig mengde data å gjennomføre meningsfull statistisk analyse. I figur 1vises de ulike komponentene av microfluidic-mikroskopi system som tillater kontrollerte forhold, inkludert temperatur og flow rate38,39,40. Bioreactor er kombinert med fluorescens mikroskopi visualisere fluorescens av EGFP koden i PA01 under brukt lavt gjennom høy skjær forhold som vil etterligne mer realistiske scenarier som er oppstått i miljøet eller i feltet biomedisinsk.
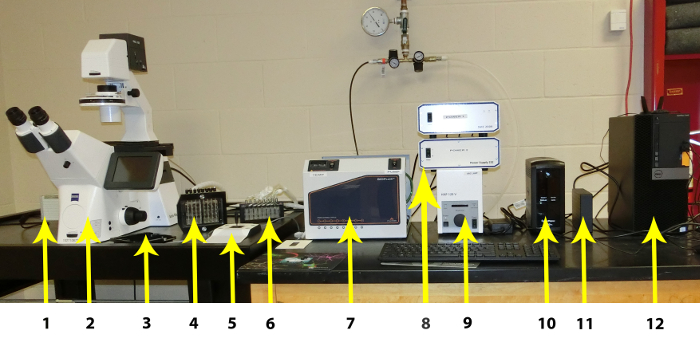
Figur 1 : Individuelle komponenter av Microfluidic. De enkelte komponentene vises fra venstre til høyre: 1. CCD kamera, 2. høy oppløsning invertert mikroskop med automatisert scenen, automatisert fluorescens modul og autofokus modulen, 3. Plate stadium 4: tenkelig system grensesnitt, 5: manuell mikroskop scenen Kontroll, 6: Damp felle, 7: tenkelig system Controller (inkludert temperatur kontroller), 8: Hardware-kontrollere, 9: fluorescens kontrolleren, 10: avbruddsfri strømforsyning, 11: ekstern harddisk for bildelagring, 12: PC-arbeidsstasjoner. Klikk her for å se en større versjon av dette tallet.
Et utdrag av microfluidic plate er vist i figur 2. Den mest brukte plater består av 48 brønner. Et eksperiment krever en vik og en stikkontakt brønner, totalt 2. Dette gir 24 samtidige eksperimenter som kan utføres med ulike eksperimentelle forhold, som bakteriell stammer, antimikrobielle og media varierte fra kanalen til kanal og kontrollert skjær flyt for hver kolonne med seks kanalene. Eksperimentell temperaturen kontrolleres også med en temperaturinnstilling hele platen. Microfluidic kanaler viser at hver kanal har en serpentin region tilstrekkelig mottrykk og kontrollert skjær.
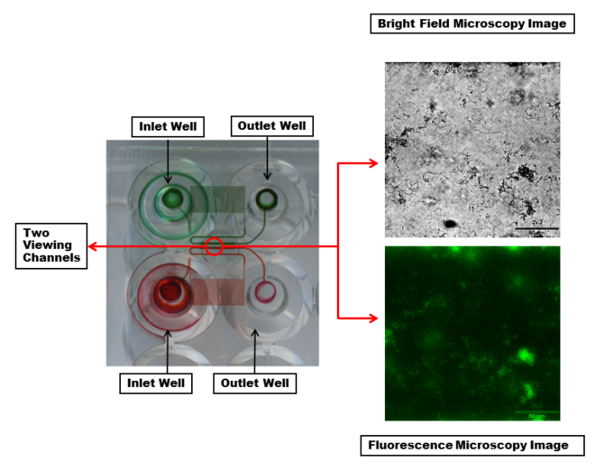
Figur 2: visualisering av microfluidic kanaler og visningsvinduet. To innløp og utløp brønner med microfluidic kanalene koble dem er markert med røde og grønne fargestoffer. Fargestoff gjør synlig en serpentin region i hver kanal som oppretter tilstrekkelig mottrykk og kontrollert skjær under væskestrøm. Hver visning kanal (i den røde sirkelen) kan avbildes med ønsket bølgelengder. Vises lysende felt (øverst) og lysrør (nederst) mikroskopi bilder av én kanal med en PA01-EGFP biofilm ved hjelp av en 20 X-målet. Skala bar = 80 µm. Klikk her for å se en større versjon av dette tallet.
En steg for steg guide gis til brukerne av microfluidic bioreaktorer som er kombinert med fluorescens mikroskopi å utføre romanen biofilm eksperimenter med ulike skjær miljøer. Denne metoden vil tillate utvidelse forsøk med andre mikroorganismer foruten bakterier, som sopp og alger, som har medisinsk og miljømessige programmer41,42,43. Den detaljerte fremgangsmåten beskriver hvordan du kultur PA01-EGFP, vaksinere en 48-vel plate og sette opp microfluidic enhet og programvare, definere fluorescerende mikroskopet og demonstrere programvare analyse for å få biofilm dekning, vekst, og morfologiske egenskaper som overflateruhet.
Protocol
Representative Results
Discussion
Microfluidic systemet og image analysis prosedyren diskutert her fokuserer for microfluidic biofilm eksperimenter for å fastsette morfologiske egenskaper som ikke krever full tredimensjonal informasjonen vanligvis finnes fra AC confocal mikroskopet studier. Disse inkluderte microcolony undergrunnen dekning (prosent dekning), overflateruhet målt grovheten koeffisient og tekstur entropi. En metode for å beregne totalt relative biofilm celle akkumulering er også presentert fase kan beregnes fra hvilke vekstrater i loggen.
Det er flere små men viktige trinn som skal markeres i denne metoden. Tørke grensesnittet med alkohol unngår forurensning av andre bakterier i går fra eksperimentet å eksperiment men også fra brønn til godt i et eksperiment. Grunning og seeding er også svært viktig fordi grunning lar brukeren bestemme hvilke kanaler tillater media å strømme gjennom kanaler uten forstyrrelser eller tilstopping. Kanaler skal ikke forstyrret (dvs. de bør være konstant full av media) etter grunning for å øke sjansene for en vellykket eksperiment med ingen luftbobler eller tilstopping. Seeding trinnet kan varieres ifølge typen bakterier og så skal være optimalisert for cellen vedlegg. For eksempel hvis cellene ikke synes å legge, overflate endringene kan ha på microfluidic platen før såing eller lengre incubations ganger kan være nødvendig. Det er også viktig å sørge for at mikroskopet er korrekt satt opp å få et i fokus bilde og bør overvåkes regelmessig gjennom å forsikre at kvalitet bilder hentes. Hvis fokus er deaktivert, mikroskopet kan og bør justeres som eksperimentet fortsetter. Imens bilde oppkjøpet, bør siste bølgelengden settes til Alle lukket for å unngå eksponering av filtre og belysning til bare én kanal under ventetid som oppstår mellom bilde oppkjøp. Også ble bildeanalyse som bestemmer den % dekningen designet i huset fordi Montage programvare manuell ikke eksplisitt beskrive prosedyren. Videre for å utvide bildeanalyse og fastsette andre egenskaper som overflateruhet, osv., åpen kilde Python-basert kode45 ble utviklet i huset og delt på gitHub oppbevaringssted. Det er også begrensninger på hvor mye data kan lagret og administrert på den lokale harddisken, så en ekstern hard kjøre eller deling av data er nødvendig som CyVerse46.
Konvensjonelle bioreaktorer, som CDC reaktoren og det dryppe flyt reaktoren34, krever mye media, gir færre utvalgene, og krever en stor mengde sterilisering av utstyr. I kontrast, fordelene ved denne høyere gjennomstrømming plattformen inkludere evnen å kontroll skjær, strømningshastigheter, og antagelsen om at i vitro eksperimenter tett ligner i vivo forhold. Ulemper av systemet inkluderer flere tilbehør og programvare som krever en omhyggelig oppsett som må utføres i riktig rekkefølge av hendelser. Videre manualen som tilbys for utstyret fullt forklarer ikke hvert trinn i eksperimenter, og kommandoene programvare, og dermed mange feil oppstår under forsøkene, inkludert tilstopping av kanaler, mangel på vekst eller vedlegg, eller mangel høykvalitets mikroskopi bilder eller filmer. Selve instrumentet og Forbruksartikler som microfluidic platene, er også relativt dyrt med en prislapp på over $200 per plate og er ikke gjenbrukes. Dermed mens teknikken gir kraftige resultater, den tekniske ekspertisen som kreves for bruken er relativt høy og krever gjentatte opplæring av eksperter på området. Denne rapporten forsøker å løse dette problemet ved å gi en guide til nye brukere av disse bioreaktorer for å studere biofilm egenskaper.
Microfluidic systemet, som er i stand til å utføre mobilnettet analyse, har fått stor oppmerksomhet for ulike vitenskapelige modaliteter, som i mikrobiologi, immunologi, hematologi, onkologi og stamcelleforskning. Mer spesifikt, har teknologien resultert i mange publikasjoner som beskriver emner som er svært relevante for medisinske anvendelser37,47, inkludert mikrobiell muntlig vedheft48, fastsettelse av virkningene av biosurfactants på Pseudomonas aeruginosa og Staphylococcus aureus49,50, vert patogen interaksjoner i E. coli51, Streptococcus tilslutning52, og behandling på cystisk fibrose53. Gitt det faktum at dette microfluidic systemet er veldig allsidig, er det forventet at flere og flere systemer vil bli distribuert over hele verden.
Noen bestemt protokoll tiltak bør vurderes nøye. Media kan bli fortynnet til 50% med dH2O for å hindre bobler og tilstopping, men var ikke nødvendig i dette tilfellet. Den bestemte verdien til OD600 brukes for seeding må bestemmes ved hjelp av prøveperioden løper av et vekst forsøk for å se hva som fungerer best for bestemt sett med betingelser som brukes. Bobler i brønnene før tetting kan føre til bobler i microfluidic kanaler og bør fjernes ved dukket eller sugd ut med Pipetter tips. Det er viktig å holde bakterier av små serpentin kanalene. Har like volum av medier i inn- og utdataene under seeding prosessen, skal flyt presset flytende volumet kontrolleres så flyt er bare følge av brukt press fra systemet. Kalibrering avstanden bør settes opp under installasjonen av selskapets representant. Disse innstillingene er spesifikke for hvert kamera.
Det finnes flere utfordringer som oppstår når finne mest representative terskelen for et bilde. Angi maksimal terskelverdier kan være vanskelig hvis gjennomsnittlig pixel intensiteten i regioner av bakgrunnen ikke er konsekvent skyldes enten velge en scenen posisjon som ikke er i midten av kanalen eller rusk på tallerkenen. Under MM Standard, falle i staver forarbeide, og velg deretter bakgrunn og skyggelegging korreksjon for korreksjon for disse inkonsekvensene. Dette verktøyet er imidlertid vanligvis bare nyttig hvis brukeren har tatt bilder av kanalene før såing som de kan bruke som referanse bilder. Hvis bakgrunnsskygger referanse bilder er utilgjengelig, brukeren må bruke deres dømmekraft til å angi en terskelverdi som dekker de celle området uten inkludert bakgrunn for hele bildet. Alternativt velge representant områder å måle som ekskluderer regioner inkonsekvens av Klikk Rektangel, Ellipse regioneneller Spor regionen velge en region og velger Aktiv Region i stedet for å hele Bilde i vinduet Vis regionen statistikk (under Analyseverktøy). Hvis en representant region benyttes for terskelverdi lyse feltet bildet, den samme regionen bør brukes for måling av tilsvarende FITC bildet. Det er nyttig å registrere bestemte romlige statistikken (venstre, toppen, bredde, høyde, området, Perimeter) tilknyttet representant regionen så samme region finner og målt på tilsvarende FITC bildet.
For å hindre en oppbygging av data på harddisken som vil føre til tregere, kan en ekstern harddisk kjøpes for datalagring. Et annet alternativ for datalagring og går lettere datadeling er CyVerse bioinformatics plattformen. Opprette en konto på CyVerse systemet ved å gå til http://www.cyverse.org/. Når logget på, starter Discovery miljøet og velg “Logg i CyVerse”. Velg “Data” og gå til du er mappen. Hvis bildestakk er på den lokale datamaskinen Velg “Last opp” og “Enkelt laste opp fra skrivebordet“. Finn stakkfilen bildet og velg for opplasting. Filen eller en mappe deles med samarbeidspartnere hvis de har en CyVerse-konto og gis tillatelse. Deling av data-mappen for allmennheten krever at metadata legges til for hver arkiv benytter CyVerse godkjente standarder. Denne prosedyren vil ikke bli diskutert her fordi dette ikke er innenfor dette arbeidet.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dette arbeidet ble gjort mulig ved tilskudd fra National Institute for generell medisinsk vitenskap (NIGMS) (5P20GM103427), en komponent av National Institutes of Health (NIH)
Materials
| Ammonium Chloride, ACS | VWR | BDH9208-500G | Part of the minimal media composition |
| BioFlux 1000 48 Well Low Shear Plate | Fluxion Biosciences | 910-0047 | |
| BioFlux 1000Z Microfluidic Imaging System | Fluxion Biosciences | BF 1000Z | |
| Calcium Chloride Dihydride, ACD Grade | VWR | 97061-904 | Part of the minimal media composition |
| Dextrose, Anhydrous, ACS | VWR | BDH9230-500G | Part of the minimal media composition |
| Magnesium Sulfate ACS Grade | VWR | EM-MX0070-1 | Part of the minimal media composition |
| Potassium Phosphate Monobasic, ACS Grade | VWR | BDH9268-500G | Part of the minimal media composition |
| Pseudomonas Aeurginosa GFP | ATCC | 15692GFP | Pseudomonas aeurinosa bacterial strain PA01 with GFP modification used for this study. |
| Sodium Chloride, ACS | VWR | BDH9286-500G | Part of the minimal media composition |
| Sodium Phosphate, Monobasic, Anhydrous, Reagent Grade | VWR | 97061-942 | Part of the minimal media composition |
References
- Donlan, R. M., et al. Model System for Growing and Quantifying Streptococcus pneumoniae Biofilms In Situ and in Real Time. Applied and Environmental Microbiology. 70 (8), 4980-4988 (2004).
- Lührig, K., et al. Bacterial Community Analysis of Drinking Water Biofilms in Southern Sweden. Microbes and Environments. 30 (1), 99-107 (2015).
- Singh, R., Paul, D., Jain, R. Biofilms: implications in bioremediation. Trends in Microbiology. 14 (9), 389-397 (2006).
- Veerachamy, S., et al. Bacterial adherence and biofilm formation on medical implants: A review. Proceedings of the Institution of Mechanical Engineers, Part H: Journal of Engineering in Medicine. 228 (10), 1083-1099 (2014).
- Lyczak, J. B., Cannon, C. L., Pier, G. B. Establishment of Pseudomonas aeruginosa infection: lessons from a versatile opportunist. Microbes and Infection. 2 (9), 1051-1060 (2000).
- Percival, S. L., Knapp, J. S., Edyvean, R., Wales, D. S. Biofilm Development On Stainless Steel In Mains Water. Water Research. 32 (1), 243-253 (1998).
- Percival, S. L., Knapp, J. S., Wales, D. S., Edyvean, R. G. J. The effect of turbulent flow and surface roughness on biofilm formation in drinking water. Journal of Industrial Microbiology and Biotechnology. 22 (3), 152-159 (1999).
- Ling, F., Whitaker, R., LeChevallier, M. W., Liu, W. -. T. Drinking water microbiome assembly induced by water stagnation. The ISME Journal. , 1 (2018).
- Moss, B. Water pollution by agriculture. Philosophical Transactions of the Royal Society B: Biological Sciences. 363 (1491), 659-666 (2008).
- Schultz, M. P., Bendick, J. A., Holm, E. R., Hertel, W. M. Economic impact of biofouling on a naval surface ship. Biofouling. 27 (1), 87-98 (2011).
- Tornero, V., Hanke, G. Chemical contaminants entering the marine environment from sea-based sources: A review with a focus on European seas. Marine Pollution Bulletin. 112 (1-2), 17-38 (2016).
- LaBauve, A. E., Wargo, M. J. Growth and Laboratory Maintenance of Pseudomonas aeruginosa. Current Protocols in Microbiology. 25 (1), 1-8 (2012).
- He, J., et al. The broad host range pathogen Pseudomonas aeruginosa strain PA14 carries two pathogenicity islands harboring plant and animal virulence genes. Proceedings of the National Academy of Sciences of the United States of America. 101 (8), 2530-2535 (2004).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2 (12), 905-909 (2005).
- Zhang, G., Gurtu, V., Kain, S. R. An Enhanced Green Fluorescent Protein Allows Sensitive Detection of Gene Transfer in Mammalian Cells. Biochemical and Biophysical Research Communications. 227 (3), 707-711 (1996).
- Patterson, G. H., Knobel, S. M., Sharif, W. D., Kain, S. R., Piston, D. W. Use of the green fluorescent protein and its mutants in quantitative fluorescence microscopy. Biophysical Journal. 73 (5), 2782-2790 (1997).
- Chalfie, M., Tu, Y., Euskirche, G., Ward, W. W., Prashert, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Heim, R., Prasher, D., Tsien, R. Wavelength mutations and posttranslational autoxidation of green fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 91, 12501-12504 (1994).
- Bakke, R., Kommedal, R., Kalvenes, S. Quantification of biofilm accumulation by an optical approach. Journal of Microbiological Methods. 44 (1), 13-26 (2001).
- Heydorn, A., et al. Statistical analysis of Pseudomonas aeruginosa biofilm development: impact of mutations in genes involved in twitching motility, cell-to-cell signaling, and stationary-phase sigma factor expression. Applied and Environmental Microbiology. 68 (4), 2008-2017 (2002).
- Wilson, E., et al. Using Fluorescence Intensity of Enhanced Green Fluorescent Protein to Quantify Pseudomonas aeruginosa. Chemosensors. 6 (21), (2018).
- Fang, H., Chen, Y., Huang, L., He, G. Analysis of biofilm bacterial communities under different shear stresses using size-fractionated sediment. Scientific Reports. 7 (1), 1299 (2017).
- Saunders, K. A., Greenman, J. The formation of mixed culture biofilms of oral species along a gradient of shear stress. Journal of Applied Microbiology. 89 (4), 564-572 (2001).
- Valquier-Flynn, H., Wilson, C. L., Holmes, A. E., Wentworth, C. D. Growth Rate of Pseudomonas aeruginosa Biofilms on Slippery Butyl Methacrylate-Co-Ethylene Dimethacrylate (BMA-EDMA), Glass and Polycarbonate Surfaces. Journal of Biotechnology & Biomaterials. 7 (4), (2017).
- Trulear, M. G., Characklis, W. G. Dynamics of biofilm processes. Journal (Water Pollution Control Federation). 54 (9), 1288-1301 (1982).
- Machineni, L., Rajapantul, A., Nandamuri, V., Pawar, P. D. Influence of Nutrient Availability and Quorum Sensing on the Formation of Metabolically Inactive Microcolonies Within Structurally Heterogeneous Bacterial Biofilms: An Individual-Based 3D Cellular Automata Model. Bulletin of Mathematical Biology. 79 (3), 594-618 (2017).
- Jiao, Y., et al. Identification of Biofilm Matrix-Associated Proteins from an Acid Mine Drainage Microbial Community. Applied and Environmental Microbiology. 77 (15), 5230-5237 (2011).
- Flemming, H. C., Wingender, J. The Biofilm Matrix. Nature Reviews Microbiology. 8 (9), 632-633 (2010).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39 (2), 109-119 (2000).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146 (10), 2395-2407 (2000).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Goeres, D. M., et al. A method for growing a biofilm under low shear at the air liquid interface using the drip flow biofilm reactor. Nature Protocols. 4 (3), 783-788 (2009).
- Goeres, D. M., et al. Statistical assessment of a laboratory method for growing biofilms. Microbiology. 151, 757-762 (2005).
- Swartz, K., Stephenson, R., Hernandez, M., Jambang, N., Boles, B. R. The use of Drip Flow and Rotating Disk Reactors for Staphylococcus aureus biofilm analysis. Journal of Visual Experiments. (46), e2470 (2010).
- Werner, E., et al. Stratified Growth in Pseudomonas aeruginosa Biofilms. Applied and Environmental Microbiology. 70 (10), 6188-6196 (2004).
- Wilson, C., et al. The Quantitative Assessment of Pseudomonas aeruginosa (PA)14 Biofilm Surface Coverage on Slippery Liquid Infused Polymer Surfaces (SLIPS). International Journal of Nanotechnology in Medicine & Engineering. 3 (3), 35-42 (2018).
- Samarian, D. S., Jakubovics, N. S., Luo, T. L., Rickard, A. H. Use of a High-throughput In Vitro Microfluidic System to Develop Oral Multi-species Biofilms. Journal of Visualized Experiments. (94), 52467 (2014).
- Rusconi, R., Garren, M., Stocker, R. Microfluidics Expanding the Frontiers of Microbial Ecology. Annual Review of Biophysics. 43, 65-91 (2014).
- Kim, J., Park, H. -. D., Chung, S. Microfluidic Approaches to Bacterial Biofilm Formation. Molecules. 17 (8), 9818-9834 (2012).
- Mosier, A. P., Cady, N. C. Analysis of bacterial surface interactions using microfluidic systems. Science Progress. 94 (4), 431-450 (2011).
- Nobile, C. J., Johnson, A. D. Candida albicans Biofilms and Human Disease. Annual Review of Microbiology. 69, 71-92 (2015).
- Chandra, J., Kuhn, D. M., Mukherjee, P. K., Hoyer, L. L., McCormick, T., Ghannoum, M. A. Biofilm Formation by the Fungal Pathogen Candida albicans: Development, Architecture, and Drug Resistance. Journal of Bacteriology. 183 (18), 5385-5394 (2001).
- Vasconcelos, M. A., et al. Effect of Algae and Plant Lectins on Planktonic Growth and Biofilm Formation in Clinically Relevant Bacteria and Yeasts. BioMed Research International. 2014, 9 (2014).
- Haralick, R. M., Shanmugam, K., Dinstein, I. Textural Features for Image Classification. IEEE Transactions on Systems, Man, and Cybernetics. 3 (6), 610-621 (1973).
- . Biofilm Morphology Suite Available from: https://github.com/cdwentworth/Biofilm-Morphology-Suite.git (2018)
- . Cyverse Available from: https://user.cyverse.org/services/mine (2018)
- Benoit, M. R., Conant, C. G., Ionescu-Zanetti, C., Schwartz, M., Matin, A. New Device for High-Throughput Viability Screening of Flow Biofilms. Applied and Environmental Microbiology. 76 (13), 4136-4142 (2010).
- Ding, A. M., Palmer, R. J., Cisar, J. O., Kolenbrander, P. E. Shear-Enhanced Oral Microbial Adhesion. Applied and Environmental Microbiology. 76 (4), 1294-1297 (2010).
- Diaz De Rienzo, M. A., Stevenson, P. S., Marchant, R., Banat, I. M. Effect of biosurfactants on Pseudomonas aeruginosa and Staphylococcus aureus biofilms in a BioFlux channel. Applied Microbiology and Biotechnology. 100 (13), 5773-5779 (2016).
- Moormeier, D. E., Bayles, K. W. Staphylococcus aureus Biofilm: A Complex Developmental Organism. Molecular Microbiology. 104 (3), 365-376 (2017).
- Tremblay, Y. D. N., Vogeleer, P., Jacques, M., Harel, J. High-Throughput Microfluidic Method To Study Biofilm Formation and Host-Pathogen Interactions in Pathogenic Escherichia coli. Applied and Environmental Microbiology. 81 (8), 2827-2840 (2015).
- Nobbs, A. H., Lamont, R. J., Jenkinson, H. F. Streptococcus Adherence and Colonization. Microbiology and Molecular Biology Reviews. 73 (3), 407-450 (2009).
- Díez-Aguilar, M., Morosini, M. I., Köksal, E., Oliver, A., Ekkelenkamp, M., Cantón, R. Use of Calgary and Microfluidic BioFlux Systems To Test the Activity of Fosfomycin and Tobramycin Alone and in Combination against Cystic Fibrosis Pseudomonas aeruginosa Biofilms. Antimicrobial Agents and Chemotherapy. 62 (1), 01650-01717 (2018).

