포유류 세포 라인 CRISPR Cas를 사용 하 여 게놈 편집
Summary
CRISPR-Cas는 식물과 동물의 복잡 한 유전자를 강력한 기술. 여기, 우리는 효율적으로 다른 Cas endonucleases를 사용 하 여 인간 게놈을 편집 하려면 프로토콜 선발. 우리는 중요 한 고려 사항 및 설계 매개 변수 편집 효율 최적화를 강조 표시 합니다.
Abstract
클러스터 된 정기적으로 interspaced 짧은 구조 반복 (CRISPR) 시스템 세균성 적응 면역에 자연스럽 게 작동 하지만 성공적으로 많은 다른 살아있는 유기 체에 있는 게놈 엔지니어링에 대 한 용도가 변경 되었습니다. 가장 일반적으로, CRISPR wildtype 관련 9 (Cas9) 또는 Cas12a endonuclease 게놈, DNA 이중 가닥 휴식 비 동종 끝 결합 (NHEJ) 통로 또는 상 동 감독 수리 (를 통해 복구 후에 특정 사이트를 쪼개 다 하는 데 사용 됩니다. HDR) 통로 기증자 서식 파일 인지에 따라 결 석 또는 각각 제시. 날짜 하려면, 다른 세균 종에서 CRISPR 시스템 게놈 포유류 세포에서 편집을 수행 할 수 표시 되었습니다. 그러나, 기술의 명백한 단순에도 불구 하 고 여러 설계 매개 변수 필요 간주, 종종 그들의 게놈 편집 실험 수행에 최고의 방법에 대 한 당황 하 게 하는 사용자를 떠나. 여기, 우리가 원하는 DNA 수정, 편집 하는 포유류 세포에 실험 하는 게놈의 성공적인 실행을 촉진의 목표를 수행 하는 세포 클론의 식별에 실험적인 디자인의 완벽 한 워크플로우를 설명 합니다. 우리는 사용자의를 위한 주요 고려 사항 선택 CRISPR 시스템, 공백 길이 그리고 단일 가닥 oligodeoxynucleotide (ssODN) 기증자 서식 파일의 디자인을 포함 하 여 강조 표시 합니다. 우리는이 워크플로 유전자 녹아웃 연구, 모델링 노력, 질병에 대 한 도움이 될 것입니다 또는 기자 생성 세포 라인을 구상.
Introduction
어떤 생명체의 게놈 엔지니어링 능력은 많은 생물 의학 및 바이오 응용 프로그램, 질병 발생의 수정 같은 돌연변이, 질병 연구에 대 한 정확한 휴대 전화 모델의 건설 또는 농업 생성 바람직한 특성을 가진 작물입니다. 세기의 차례 이후 다양 한 기술 개발 되었으며, meganucleases,12,3을 포함 한 포유류 세포에서 게놈 엔지니어링, 아연 손가락 nucleases4,5또는 전사 활성 제 같은 이펙터 nucleases (TALENs)6,7,,89. 그러나, 이러한 이전 기술을 어려운 프로그램 또는 조립, 지루한 연구 및 업계에서 광범위 하 게 채택 함으로써 방해.
최근 몇 년 동안에는 클러스터 된 정기적으로 짧은 구조 반복 (CRISPR) interspaced-CRISPR 관련 (Cas) 시스템 엔지니어링 기술10,11강력한 새로운 게놈으로 떠오르고 있다. 원래 박테리아에 적응 면역 체계, 되었습니다 성공적으로 식물과 동물, 인간을 포함 한 게놈 수정에 대 한 배포. CRISPR-Cas 같은 짧은 시간에 너무 많은 인기를 얻고 있다 왜 주된 이유 주요 Cas endonuclease, Cas9 또는 Cas12a (Cpf1 라고도 함) 등을 제공 하는 요소, 게놈에 있는 정확한 위치는 단순히 공상 단일 가이드 RN의 짧은 조각 (SgRNA), A는 디자인을 간단 하 고 저렴 한 합성. 대상 사이트에 보충 되 고, 후 Ca 효소 분자가 위 한 쌍으로 하 고 그것의 RuvC, 트, 또는 Nuc 도메인12,,1314바인딩된 DNA를 앞. 결과 두 배 좌초 휴식 (DSB)은 비 동종 끝 결합 (NHEJ) 또는 상 동 감독 수리 (HDR) 통로 통해 세포에 의해 연속적으로 복구 됩니다. 복구 서식 파일의 부재, DSB 단백질 코딩 유전자에 frameshift 돌연변이 일으키는 잠재적으로 야기할 수 있는 난수 삽입 또는 삭제 컷된 사이트에 뉴클레오티드 (indels), 오류가 NHEJ 통로 의해 수리는. 그러나, 원하는 DNA 변화를 포함 하는 기증자 템플릿을, 존재 DSB 고화질 HDR 통로 의해 수리는. 기증자 서식 파일의 일반적인 종류 단일 가닥 oligonucleotides (ssODNs) 및 플라스 미드 포함 됩니다. 전 반면 후자는 일반적으로 사용 하나 비교적 긴 시퀀스를 삽입 하고자 하는 경우 원하는 DNA 변경 (예: 단일 기본적인 쌍의) 작은 경우에 일반적으로 사용 됩니다 (예를 들어 녹색 형광 단백질의 코딩 순서 또는 GFP) 대상 장소에.
Cas 단백질의 endonuclease 활동 대상 사이트15에 protospacer 인접 한 모티프 (PAM)의 존재를 요구 한다. Cas9의 팸은 protospacer의 3′ 끝에는 PAM의 Cas12a (Cpf1 라고도 함)는 5′ 끝에 대신 하는 동안16. Cas-가이드 복잡 한 RNA는 PAM은17에 결 석 하는 경우는 DSB 소개 하 수 없습니다. 따라서, PAM 특정 Ca nuclease 쪼개 다 수가 게놈 위치에 제약 조건을 배치 합니다. 다행히, 다른 세균 종에서 Cas nucleases 일반적으로 다른 팸 요구를 전시 한다. 따라서, 우리의 엔지니어링 도구 상자에 다양 한 CRISPR Cas 시스템을 통합, 우리는 게놈에 타겟이 될 수 있습니다 사이트의 범위를 확장할 수 있습니다. 또한, 자연 Ca 효소 설계 수 있습니다 또는 대체 팸 시퀀스, 게놈 목표 조작18,,1920에 액세스할 수의 범위를 더욱 확대 인식 진화.
여러 CRISPR Cas 시스템 게놈 기술 설계 목적을 위해 사용할 수 있지만, 기술의 대부분의 사용자는 여러 이유로 주로 연쇄 상 구 균 pyogenes (SpCas9)에서 Cas9 nuclease에 의존 했습니다. 첫째,이 상대적으로 단순히 NGG 팸, 더 복잡 한 PAMs 존재만 쪼개 다 수 다른 많은 Ca 단백질 달리 필요 합니다. 둘째, 인간의 세포21,,2223,24에 성공적으로 배포 하는 첫 번째 Ca endonuclease 이다. 셋째, SpCas9까지 날짜에 최고의 특징이 효소 이다. 연구자 다른 Cas nuclease 사용 하고자 하는 경우 그 또는 그녀가 종종 것 디자인 실험 및 다른 효소 SpCas9에 비해 다른 생물학 문맥에서 얼마나 잘 수행 하는 최선의 방법에 대 한 명확한.
다른 CRISPR Cas 시스템의 상대적인 성능에 선명도 제공 하려면 우리 최근 포도 상 구 균 (SaCas9), 에서 Cas9 효소에서에서 Cas9 효소 5 개의 Ca endonucleases-SpCas9의 체계적인 비교를 수행 했습니다. Neisseria meningitidis (NmCas9) (AsCas12a)에 Acidaminococcus sp. BV3L6에서에서 Cas12a의 효소 및 Cas12a 효소 Lachnospiraceae 박테리아 ND2006에서에서 (LbCas12a)25. 공정한 비교를 위해 우리는 다른 실험 조건 및 대상 사이트의 동일한 세트를 사용 하 여 다양 한 Cas nucleases 평가. 연구 또한 delineated 디자인 매개 변수 각 CRISPR Cas 시스템 기술의 사용자를 위한 유용한 참고 자료 역할을. 여기, 더 나은 CRISPR Cas 사용 하도록 연구 활성화 시스템, 다른 Cas9 및 Cas12a 효소 ( 그림 1참조)로 최적의 게놈 엔지니어링에 대 한 단계별 프로토콜 제공. 프로토콜 뿐만 아니라 포유류 세포에서 게놈 성공적인 엔지니어링 결과의 가능성을 최대화 하기 또한 중요 한 디자인 고려 하지만 실험 내용을 포함 합니다.
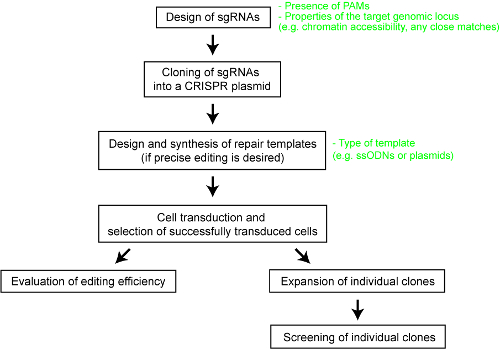
그림 1 : 게놈을 생성 하는 워크플로 개요 편집 인간의 세포 라인. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
Protocol
Representative Results
Discussion
CRISPR-Cas 시스템 엔지니어 유전자 및 식물 및 동물의 transcriptomes 강력 하 고 혁신적인 기술입니다. 많은 세균성 종의 게놈 및 transcriptome 목적44엔지니어링에 대 한 적응 수 있습니다 CRISPR Cas 시스템을 포함 하도록 발견 되었습니다. 연쇄 상 구 균 pyogenes (SpCas9)에서 Cas9 endonuclease은 배포할 성공적으로 인간 세포21,22,,…
Disclosures
The authors have nothing to disclose.
Acknowledgements
M.H.T.는 과학 기술 및 연구의 공동 위원회 사무실 그랜트 (1431AFG103)에 대 한 기관에 의해 지원 됩니다, 국가 의료 연구 위원회 (OFIRG/0017/2016) 부여, 국립 연구 재단 보조금 (NRF2013-THE001-046, NRF2013-THE001-093)는 교육부의 교육 1 단계 그랜트 (RG50/17 (S)), 난 양 기술 대학교, 난 양 기술 대학교에서 국제 유전자 공학 기계 (iGEM) 경쟁에 대 한 자금에서 시작 권한을 부여 합니다.
Materials
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | – | – | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
References
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. 유전학. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).

