In silico Modeling method voor computationele aquatische toxicologie van hormoonontregelende stoffen: een op software gebaseerde aanpak met QSAR toolbox
Summary
De kwantitatieve structuur-Activiteitsrelatie (QSAR) modellering is een representatieve bioinformatica-geassisteerde methode in de toxicologische screening. Dit protocol laat zien hoe de Risico’s van hormoonontregelende stoffen (EDs) in aquatische omgevingen op een computationeel wijze kunnen worden beoordeeld. Door gebruik te maken van de OESO QSAR toolbox implementeert het protocol een in silico-test voor het analyseren van de toxiciteit van hormoonontregelende stoffen in vissen.
Abstract
Computationele analyses van toxicologische processen maakt screening van chemische stoffen met een hoge doorvoer mogelijk en voorspelling van hun eindpunten in biologische systemen. Met name de modellen van de kwantitatieve structuur-activiteitsverhouding (QSAR) zijn in toenemende mate toegepast om de milieueffecten van een overvloed aan giftige stoffen te beoordelen. In de afgelopen jaren, sommige meer gemarkeerde soorten toxicantia zijn hormoonontregelende stoffen (EDs, die chemicaliën die kunnen interfereren met een hormoon-gerelateerde metabolisme). Omdat hormoonontregelende stoffen een aanzienlijke invloed kunnen hebben op de ontwikkeling en reproductie van dieren, is een snelle voorspellen van de nadelige effecten van EDs die in silico-technieken werken vereist Deze studie presenteert een in silico-methode om voorspellingsgegevens te genereren over de effecten van representatieve hormoonontregelende stoffen in aquatische gewervelde dieren, in het bijzonder vissoorten. Het protocol beschrijft een voorbeeld met behulp van de geautomatiseerde workflow van de QSAR toolbox-software die is ontwikkeld door de organisatie voor economische samenwerking en ontwikkeling (OESO) om acute ecotoxiciteits voorspellingen van EDs mogelijk te maken. Als gevolg hiervan worden de volgende gegevens bepaald: (1) berekening van de numerieke correlaties tussen de concentratie voor 50% van de letaliteit (LC50) en de verdelingscoëfficiënt octanol-water (Kow), (2) uitvoerprestaties waarbij de LC50 waarden bepaald in experimenten worden vergeleken met die gegenereerd door berekeningen, en (3) de afhankelijkheid van oestrogeen receptor bindende affiniteit op de relatie tussen Kow en LC50.
Introduction
Nieuwe ontwikkelingen in informatica en computationele technologie hebben de biologische wetenschappen gemachtigd met kwantitatieve methodologieën die hoge precisie en betrouwbaarheid bieden1. In het bijzonder hebben algoritmen die worden gebruikt in de moleculaire taxonomie en classificatie van eigenschappen geleid tot kwantitatieve structuur-activiteitsrelaties (QSAR) modellen2. Deze modellen correleren automatisch de chemische structuren en biologische activiteiten van een bepaalde chemische database en implementeren snel in silico-screening van een breed scala aan chemische substraten volgens hun medicinale of toxicologische acties3. QSAR-hulpmiddelen kunnen voorspellende toxiciteits profielen produceren als functie van vectoren van moleculaire descriptoren (d.w.z. fysisch-chemische parameters) van chemicaliën die van belang zijn voor het numeriek maken van categorische eindpunten4. Gewoonlijk wordt elk kwantitatief eindpunt weergegeven als een 2D teststappenvisualization versus wijzigingen in descriptor waarden. Vervolgens wordt een QSAR-model gegenereerd met behulp van (meervoudige) lineaire regressieanalyses. Zodra een gegevensset volledig is benut om een QSAR-model (de Trainingsset) te maken, wordt het model statistisch gevalideerd door de eindpunten te voorspellen van een groep chemicaliën die niet in de Trainingsset is opgenomen (de testset). Het model kan vervolgens worden gebruikt om de biologische activiteiten van niet-geteste verbindingen3te voorspellen.
Onder veel schadelijke chemicaliën, hormoonontregelende stoffen (EDs) zijn gemarkeerd als een groep van toxicantia die kunnen interfereren met talrijke hormoon-gerelateerde metabolismen in zoogdieren, amfibieën, en vis5,6. EDs zijn bekend voor het opwekken van een verscheidenheid van nadelige effecten, zoals kanker en misvormingen, door het blokkeren of wijzigen van normale hormonale trajecten of het activeren van abnormale hormoon synthese/afbraak signalen. Dientengevolge, deze hormoon-nabootsen chemicaliën kunnen perturb endocriene systemen zodanig dat biologische ontwikkeling en reproductie van Wildlife dierenpopulaties worden belemmerd. In het bijzonder zijn de ecotoxicologische effecten van hormoonontregelende stoffen uitgebreid onderzocht in aquatische gewervelde dieren, die bijna identieke hormoonreceptor structuren hebben met die van zoogdieren, waaronder mensen. Omdat alle hormonale acties optreden bij lage doses in vivo, is het voorspellen van de potentiële toxiciteiten van ED-kandidaten met behulp van Rapid in silico-screening van cruciaal belang voor de gezondheid van het publiek en het milieu.
QSAR-modellen op basis van de toxicologie van EDs zijn uitgevoerd met behulp van zowel 2D-als 3D-descriptoren (bekend als 2D en 3D QSAR, respectievelijk), die de ED ligand bindende affiniteiten van oestrogeen, androgeen en progesteron-receptoren7onthullen. Ondanks de zeer nauwkeurige voordelen van 3D QSAR, waarin conformationele en elektrostatische interacties worden overwogen, behoudt 2D QSAR zijn eigen robuustheid in directe wiskundige algoritmen, snelle berekeningen en extreem lage reken krachten. Bovendien zijn 2D-QSAR-modellen flexibel voor gebruik in een breed scala aan toepassingen, terwijl relatief nauwkeurige Voorspellings prestaties worden bereikt.
De OESO QSAR toolbox is momenteel een van de meest gebruikte computersoftware tools die gratis beschikbare en vooraf gebouwde QSAR-modellen8,9bieden. De Profiler maakt gebruik van 2D descriptor databases. Sinds de release van de eerste versie in 2008 is de software toegepast op het gebied van chemische en biologische industrieën, volksgezondheid en milieu veiligheid voor volledige of gedeeltelijke analyse van de potentiële Risico’s van natuurlijke en synthetische verbindingen, met bijzondere belangen in carcinogenese10,11,12, mutageniteit13,14,15en ontwikkelingstoxiciteit16. De toepassing op aquatische toxicologie is ook aangetoond, met focus op bioaccumulatie en biotransformatie17.
De QSAR toolbox is nuttig gebleken bij het voorspellen van de toxiciteit op korte termijn van een breed scala van chemicaliën17, evenals de oestrogeen receptor (er) bindende verwantschap van EDs18. De acute ecotoxiciteiten van hormoonontregelende stoffen in aquatische gewervelde dieren zijn echter niet geanalyseerd met behulp van de QSAR toolbox. In deze studie, een typische en facile protocol wordt gepresenteerd voor het uitvoeren van QSAR Modeling op de acute nadelige effecten van hormoonontregelende stoffen met een focus in de vissoorten. De studie toont aan dat de QSAR toolbox een zeer toegankelijke software is voor het berekenen en voorspellen van de letaliteit/sterfte van aquatische gewervelde dieren voor sommige representatieve hormoonontregelende stoffen. statistische behandelingsmethoden voor de afgeleide in silico-gegevenssets worden gepresenteerd. Figuur 1 toont het algemene schema voor de algemene werking van de QSAR toolbox. De in Figuur 2 getoonde workflow biedt eenvoudige instructies voor het gebruik van de in silico-assay om acute ecotoxiciteit van doelstoffen zoals hormoonontregelende chemicaliën te voorspellen.
Protocol
Representative Results
Discussion
De veelzijdigheid van de OESO QSAR toolbox als analytische software voor Ecotoxicologie wordt hier getoond met specifieke belangstelling voor de nadelige effecten van hormoonontregelende stoffen op aquatische gewervelde dieren. Daarnaast werd een eenvoudig en standaardprotocol aangetoond voor het voorspellen van acute toxiciteit (96-h LC50) van 74 representatieve EDs (tabel 1) voor vissoorten. Dit werd bereikt door het toepassen van categorie bouw, data gap vulling en er profilerings modules ingebed in de QSAR toolbox (Figuur 1, Figuur 2).
De lineaire correlatie tussen log10LC50 en log10Kow met een negatieve helling (zoals weergegeven in aanvullende figuur S1) is al lange tijd bekend als een standaard kwantitatieve relatie in QSAR-analyses25, waarbij hogere toxiciteit wordt aangetoond hoe meer hydrofobe een bepaalde chemische stof is. Zoals blijkt uit een eenvoudige berekening, is de algemene wiskundige relatie met vergelijking S1 en vergelijking S2 (aanvullende informatie) een geconverteerde uitdrukking uit de volgende Power functie26:
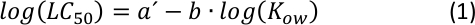

Vanaf het perceel van (vergelijking 2) kan het karakteriseren van een tussen bereik van Kow26 mogelijk zijn door de parameters a en b aan te passen, waarbij een bepaalde variatie in hydrofobiciteit (of hydrofiele) de eindpunt van acute toxiciteit.
Vergelijkende analyses tussen de computationele voorspellingen en experimentele waarnemingen op de LC50, zoals weergegeven in Figuur 3 en Figuur 4, zijn doorgaans gerapporteerd in studies van QSAR voor verschillende aquatische toxicanten, waaronder technische nonionische oppervlakteactieve stoffen27, triazoolfungiciden28en pesticide metabolieten21. Dit type retrospectieve validatie geeft informatie over de mate waarin een bepaalde QSAR-tool kan reiken in termen van vergelijkende prestaties voor experimentele resultaten. In deze studie van acute toxiciteit in vis, de QSAR toolbox bleek te bieden beschermende voorspellingen voor meer dan 90% van geteste EDs in alle vissen en in een enkele soort, Pimephales promelas.
Verdere identificatie van de drie uitschieter chemicaliën in Figuur 3 en Figuur 4, die een hogere voorspelde LC50 gemiddeld en minimaal, respectievelijk, is gebleken. Eerste, de 3 ‘, 5, 7-trihydroxy-4 ‘, 6-dimethoxyisoflavone is een soort flavonoïde (meer in het bijzonder, een isoflavone), die wordt beschouwd als over het algemeen veilig en gebruikt in kruidengeneesmiddelen; echter, het heeft nog steeds oestrogeen-gerelateerde bezorgdheid29 en kan leiden tot acute toxiciteit waarschijnlijk door middel van oxidatieve fosforylering ontkoppeling30. Vervolgens is de 1,4-benzenediol, genaamd hydroquinone, een fenol verbinding die een niet-specifieke en cytotoxische immuunrespons in vissen kan veroorzaken31. Tot slot, de 4-hexylphenol is gekend om te exposeren voldoende positieve oestrogene activiteit worden geclassificeerd als een ED32. Het is goed bestudeerd dat de belangrijkste reden van de acute toxiciteit van Hydrochinon is de reductie-oxidatie (redox) fietsen. De Hydrochinon wordt geoxideerd tot Benzochinon en teruggebracht tot semi-quinon of Hydrochinon herhaaldelijk, met afputten van cofactoren en het genereren van reactieve zuurstof soorten33. De andere twee chemicaliën kunnen diepere onderzoeken vereisen om hun werkingsmechanismen in acute ecotoxiciteit te onthullen met behulp van moleculaire docking benaderingen zoals die gebruikt door Panche et al.34, die niet kan worden gedekt door de QSAR toolbox.
EDs interfereren met het endocriene systeem voornamelijk door middel van fysisch-chemische interacties met steroïde receptoren zoals oestrogeen en androgeen receptoren, die van groot belang zijn in QSAR Modeling studies35. Gezien dit, de QSAR toolbox is robuust in termen van facile en snelle classificatie van ER bindende affiniteiten voor een set van chemicaliën op basis van alleen op de 2D descriptoren van moleculaire structuren. Toen dit ER Profiler systeem werd toegepast op onze lijst van hormoonontregelende stoffen, werd er geen duidelijke correlatie gevonden tussen ER bindingsaffiniteit en hydrofobiciteit (aanvullende figuur S2). Dit resultaat kan worden verklaard door het feit dat de vorming van een steroïde-receptor complex is niet een direct gevolg van een hydrofobe hechting bijdrage, maar moet gepaard gaan met een conformationele verandering in de actieve-site receptor structuur36. De receptor binding kan ook worden veroorzaakt door waterstof binding en π-stacking.
Bovendien kan de positie van elke chemische groep op het molecuul de receptor binding beïnvloeden, zelfs als de hydrofobiciteit en het aantal van de waterstof-obligatie acceptoren-donoren hetzelfde blijven. Ten tweede produceerde de ER Profiler tegengestelde trends tussen voorspelde en experimentele LC50 -gemiddelde niveaus met een toenemende er-bindingsaffiniteit (Figuur 5). Dit kan zijn omdat de letaliteit van ouders in een acute toxiciteitstest niet te wijten is aan ER binding, maar eerder aan narcose in de meeste gevallen, of aan redox fietsen in het geval van hydroquinone. Er is bijvoorbeeld een uitgebreidere analyse, inclusief de chronische toxiciteit, vereist voor een grotere set hormoonontregelende stoffen om voorspellende beperkingen van de huidige versie van de QSAR toolbox te definiëren.
Dit voorbereidend onderzoek kan ook gevolgen hebben voor de volksgezondheid, omdat steroïden (androgenen, oestrogenen, progestines, en corticoïden) en hun receptoren vertonen vergelijkbare of zelfs identieke macromoleculaire structuren over gewervelde dieren5. Deze soorten analoge endocriene signaleringssystemen kunnen werken met behulp van een gemeenschappelijk mechanisme in belangrijke gebeurtenissen van EDs5. Niettemin zijn aanvullende en complementaire methodologieën nodig om dit enorme en complexe aspect te verlichten [bijvoorbeeld door het uitvoeren van computationele modellering van absorptie, distributie, metabolisme en uitscheiding (ADME) en/of ongunstige uitkomst Pathway (AOP)]38. Bovendien, omdat de meeste wetenschappelijke en publieke bezorgdheid over de nadelige effecten van hormoonontregelende stoffen verband houden met hun chronische toxiciteiten, het verbeteren van de databases en algoritmen in de QSAR toolbox en het produceren van betrouwbare lange termijn ecotoxicologie voorspellingen voor EDs zijn beide nodig.
Deze paper demonstreert de toepassing van de QSAR toolbox om ecotoxicologische LC50 waarden voor vissen te vergelijken met Log10Kow waarden van EDs. in het gehele protocol leidt dit tot zwakke verhoudingen tussen de twee parameters, werd onthuld door eerdere studies (bijv., Kim et al.39) dat Log10Kow is niet een goede directe voorspeller van aquatische LC50. Ondanks deze beperking biedt dit protocol een algemene beoordeling of “vignet” om te beschrijven hoe het dashboard voor een bepaald doel moet worden gebruikt, omdat het een geldige toepassing is voor het gebruik van de QSAR toolbox voor het onderzoeken van correlaties tussen LC50 (of er-binding -affiniteit) en log10Kow, of als een hulpmiddel voor snelle acute ecotoxiciteit screening. Niettemin moet worden opgemerkt dat (1) het verlichten van het verband tussen oestrogeen receptor binding en chronische toxiciteit, in plaats van acute toxiciteit (letaliteit), relevanter is zodat duidelijkere correlaties kunnen worden gevonden, en (2) de androgeen receptor, samen met die van oestrogeen, speelt ook een cruciale rol in reproductietoxiciteit. Daarom is het vereist voor de toekomstige versie van de QSAR toolbox om de Voorspellings functies in het licht van die twee punten te verbeteren.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dit onderzoek werd gesteund door de National Research Council of Science & Technology (NST) Grant door de Zuid-Koreaanse overheid (MSIP) (nr. CAP-17-01-KIST Europe) en project 11911.
Materials
| Adobe Acrobat Reader DC | Adobe Systems Software Ireland Limited | NA | Required to view prediction and category report |
| Computer | System: Microsoft Corporation | NA | Recommended system properties: (i) system type: 64 bit, Microsoft Windows 7 or newer, (ii) processor: I5 at 2.4 GHz or faster processor or equivalent AMD CPU, (iii) Installed memory (RAM): 6 GB of RAM, (iv) Hard Disk Drive (HDD): 20 GB free hard drive space |
| Microsoft Editor | Microsoft Corporation | NA | Required to upload a substance list of CAS numbers (batch mode) to the OECD QSAR Toolbox as .txt file (text file) |
| Microsoft Excel 2016 | Microsoft Corporation | NA | Required to export data from OECD QSAR Toolbox as .cvs, .xls or .xlsx files |
| OECD QSAR Toolbox version 4.0 or newer | Organisation for Economic Co-operation and Development |
NA | Required to run OECD QSAR Toolbox Automated Workflows; free download: https://qsartoolbox.org/download/ |
| OriginPro 9 | OriginLab Corporation | NA | Optional program for data analysis; similar tools possible |
References
- Najarian, K., Najarian, S., Gharibzadeh, S., Eichelberger, C. N. . Systems Biology and Bioinformatics: A Computational Approach. , (2009).
- Fujita, T., Iwasa, J., Hansch, C. A new substituent constant, π, derived from partition coefficient. Journal of the American Chemical Society. 86, 5175-5180 (1964).
- Roy, K., Kar, S., Das, R. N. . Understanding the Basics of QSAR for Applications in Pharmaceutical Sciences and Risk Assessment. , (2015).
- Raies, A. B., Bajic, V. B. In silico toxicology: computational methods for the prediction of chemical toxicity. WIREs Computational Molecular Science. 6, 147-172 (2016).
- Hayes, T. B. Welcome to the revolution: integrative biology and assessing the impact of endocrine disruptors on environmental and public health. Integrative Compuational Biology. 45, 321-329 (2005).
- Schug, T. T., et al. Minireview: endocrine disruptors: past lessons and future directions. Molecular Endocrinology. 30, 833-847 (2016).
- Devillers, J., Marchand-Geneste, N., Carpy, A., Porcher, J. M. SAR and QSAR modeling of endocrine disruptors. SAR QSAR Environmental Research. 17, 393-412 (2006).
- Dimitrov, S. D., et al. QSAR Toolbox – workflow and major functionalities. SAR QSAR Environmental Research. 27, 203-219 (2016).
- Yordanova, D., et al. Automated and standardized workflows in the OECD QSAR Toolbox. Computational Toxicology. 10, 89-104 (2019).
- Mombelli, E., Devillers, J. Evaluation of the OECD (Q)SAR Application Toolbox and Toxtree for predicting and profiling the carcinogenic potential of chemicals. SAR QSAR Environmental Research. 21, 731-752 (2010).
- Devillers, J., Mombelli, E., Samsera, R. Structural alerts for estimating the carcinogenicity of pesticides and biocides. SAR QSAR Environmental Research. 22, 89-106 (2011).
- Li, C., et al. Identifying unknown by-products in drinking water using comprehensive two-dimensional gas chromatography-quadrupole mass spectrometry and in silico toxicity assessment. Chemosphere. 163, 535-543 (2016).
- Devillers, J., Mombelli, E. Evaluation of the OECD QSAR Application Toolbox and Toxtree for estimating the mutagenicity of chemicals. Part 1. Aromatic amines. SAR QSAR Environmental Research. 21, 753-769 (2010).
- Devillers, J., Mombelli, E. Evaluation of the OECD QSAR Application Toolbox and Toxtree for estimating the mutagenicity of chemicals. Part 2. α-β unsaturated aliphatic aldehydes. SAR QSAR Environmental Research. 21, 771-783 (2010).
- Kulkarni, S. A., Barton-Maclaren, T. S. Performance of (Q)SAR models for predicting Ames mutagenicity of aryl azo and benzidine based compounds. Journal of Environmental Science and Health Part C Environmental Carcinogenesis & Ecotoxicology Reviews. 32, 46-82 (2014).
- Craig, E. A., Wang, N. C., Zhao, Q. J. Using quantitative structure-activity relationship modeling to quantitatively predict the developmental toxicity of halogenated azole compounds. Journal of Applied Toxicology. 34, 787-794 (2014).
- Tebby, C., Mombelli, E., Pandard, P., Péry, A. R. Exploring an ecotoxicity database with the OECD (Q)SAR Toolbox and DRAGON descriptors in order to prioritise testing on algae, daphnids, and fish. Science of the Total Environment. 409, 3334-3343 (2011).
- Mombelli, E. Evaluation of the OECD (Q)SAR Application Toolbox for the profiling of estrogen receptor binding affinities. SAR QSAR Environmental Research. 23, 37-57 (2012).
- Verhaar, H. J. M., van Leeuwen, C. J., Hermens, J. L. M. Classifying environmental pollutants. 1: structure-activity relationships for prediction of aquatic toxicology. Chemosphere. 25, 471-491 (1992).
- Enoch, S. J., Hewitt, M., Cronin, M. T. D., Azam, S., Madden, J. C. Classification of chemicals according to mechanism of aquatic toxicity: an evaluation of the implementation of the Verhaar scheme in Toxtree. Chemosphere. 73, 243-248 (2008).
- Burden, N., Maynard, S. K., Weltje, L., Wheeler, J. R. The utility of QSARs in predicting acute fish toxicity of pesticide metabolites: a retrospective validation approach. Regulatory Toxicology and Pharmacology. 80, 241-246 (2016).
- Nendza, M., et al. Screening for potential endocrine disruptors in fish: evidence from structural alerts and in vitro and in vivo toxicological assays. Environmental Sciences Europe. 28, 26 (2016).
- Roncaglioni, A., Piclin, N., Pintore, M., Benfenati, E. Binary classification models for endocrine disrupter effects mediated through the estrogen receptor. SAR QSAR Environmental Research. 19, 697-733 (2008).
- Sosnovcová, J., Rucki, M., Bendová, H. Estrogen receptor binding affinity of food contact material components estimated by QSAR. Central European Journal of Public Health. 24, 241-244 (2016).
- Walker, J. D., Dearden, J. C., Schultz, T. W., Jaworska, J., Comber, M. H. I., Walker, J. D. QSARs for New Practitioners. QSARs for Pollution Prevention, Toxicity Screening, Risk Assessment, and Web Applications. , (2003).
- Sánchez-Bayo, F. From simple toxicological models to prediction of toxic effects in time). Ecotoxicology. 18, 343-354 (2009).
- Sjöström, M., Lindgren, &. #. 1. 9. 7. ;., Uppgård, L. L., Chen, F., Schüürmann, G. Joint Multivariate Quantitative Structure-Property and Structure-Activity Relationships for a Series of Technical Nonionic Surfactants. Quantitative Structure-Activity Relationships in Environmental Sciences-VII. , (1997).
- Ding, F., Guo, J., Song, W., Hu, W., Li, Z. Comparative quantitative structure-activity relationship (QSAR) study on acute toxicity of triazole fungicides to zebrafish. Chemistry Ecology. 27, 359-368 (2011).
- Galati, G., O’Brien, P. J. Potential toxicity of flavonoids and other dietary phenolics: significance for their chemopreventive and anticancer properties. Free Radical Biology in Medicine. 37, 287-303 (2004).
- Russom, C. L., Bradbury, S. P., Broderius, S. J. Predicting modes of action from chemical structure: acute toxicity in the fathead minnow (Pimephales promelas). Environmental Toxicology and Chemistry. 16, 948-967 (1997).
- Taysse, L., Troutaud, D., Khan, N. A., Deschaux, P. Structure-activity relationship of phenolic compounds (phenol, pyrocatechol and hydroquinone) on natural lymphocytotoxicity of carp (Cyprinus carpio). Toxicology. 98, 207-214 (1995).
- Nishihara, T., et al. Estrogenic activities of 517 chemicals by yeast two-hybrid assay. Journal of Health Science. 46, 282-298 (2000).
- Bolton, J. L., Trush, M. A., Penning, T. M., Dryhurst, G., Monks, T. J. Role of quinones in toxicology. Chemical Research in Toxicology. 13, 135-160 (2000).
- Panche, A. N., Diwan, A. D., Chandra, S. R. Flavonoids: an overview. Journal of Nutritional Science. 5, e47 (2016).
- Li, J., Gramatica, P. QSAR classification of estrogen receptor binders and pre-screening of potential pleiotropic EDCs. SAR QSAR Environmental Research. 21, 657-669 (2010).
- Bohl, M. . Molecular Structure and Biological Activity of Steroids. , (2017).
- Kaminuma, T., Takai-Igarashi, T., Nakano, T., Nakata, K. Modeling of signaling pathways for endocrine disruptors. BioSystems. 55, 23-31 (2000).
- Lillicrap, A., et al. Alternative approaches to vertebrate ecotoxicity tests in the 21st century: a review of developments over the last 2 decades and current status. Environmental Toxicology and Chemistry. 35, 2637-2646 (2016).
- Kim, J. W., et al. Acute toxicity of pharmaceutical and personal care products on freshwater crustacean (Thamnocephalus platyurus) and fish (Oryzias latipes). Journal of Toxicological Sciences. 34, 227-232 (2009).

