En Silico Modeling Method for Computational Aquatic Toxicology of Endocrine Disruptors: A Software-Based Approach Using QSAR Toolbox
Summary
El modelado cuantitativo de la relación estructura-actividad (QSAR) es un método representativo asistido por la bioinformática en el cribado toxicológico. Este protocolo demuestra cómo evaluar computacionalmente los riesgos de los disruptores endocrinos (ED) en entornos acuáticos. Utilizando la Caja de Herramientas QSAR de la OCDE, el protocolo implementa un ensayo in silico para analizar la toxicidad de los DE En los peces.
Abstract
Los análisis computacionales de los procesos toxicológicos permiten el cribado de alto rendimiento de sustancias químicas y la predicción de sus variables en sistemas biológicos. En particular, los modelos cuantitativos de relación estructura-actividad (QSAR) se han aplicado cada vez más para evaluar los efectos ambientales de una plétora de materiales tóxicos. En los últimos años, algunos tipos más destacados de tóxicos son disruptores endocrinos (ED, que son productos químicos que pueden interferir con cualquier metabolismo relacionado con las hormonas). Debido a que los ED pueden afectar significativamente el desarrollo y la reproducción de los animales, es necesario predecir rápidamente los efectos adversos de los DE mediante técnicas de silicona. Este estudio presenta un método in silico para generar datos de predicción sobre los efectos de los ED representativos en vertebrados acuáticos, particularmente especies de peces. El protocolo describe un ejemplo que utiliza el flujo de trabajo automatizado del software QSAR Toolbox desarrollado por la Organización para la Cooperación y el Desarrollo Económicos (OCDE) para permitir predicciones agudas de ecotoxicidad de los ED. Como resultado, se determinan los siguientes factores: (1) cálculo de las correlaciones numéricas entre la concentración del 50% de la letalidad (LC50) y el coeficiente de partición octanol-agua (Kow), (2) rendimientos de salida en los que la LC50 los valores determinados en los experimentos se comparan con los generados por los cálculos, y (3) la dependencia de la afinidad de unión del receptor de estrógeno en la relación entre Kow y LC50.
Introduction
Los nuevos desarrollos en informática y tecnología computacional han empoderado a las ciencias biológicas con metodologías cuantitativas que ofrecen alta precisión y fiabilidad1. En particular, los algoritmos utilizados en la taxonomía molecular y la clasificaciónde propiedades han dado lugar a modelos cuantitativos de relación estructura-actividad (QSAR) 2. Estos modelos correlacionan automáticamente las estructuras químicas y las actividades biológicas de una base de datos química determinada e implementan rápidamente en el cribado de silico de una amplia gama de sustratos químicos de acuerdo con sus acciones medicinales o toxicológicas3. Las herramientas QSAR pueden producir perfiles predictivos de toxicidad en función de los vectores de características de los descriptores moleculares (es decir, parámetros fisicoquímicos) de productos químicos de interés para crear numéricamente puntos finales categóricos4. Normalmente, cada punto final cuantitativo se muestra como una gráfica de dispersión 2D frente a los cambios en los valores del descriptor. A continuación, se genera un modelo QSAR mediante análisis de regresión lineal (múltiples). Una vez que un conjunto de datos se ha explotado completamente para construir un modelo QSAR (denominado conjunto de entrenamiento), el modelo se valida estadísticamente prediciendo los puntos finales de un grupo de productos químicos no incluidos en el conjunto de entrenamiento (denominado conjunto de pruebas). El modelo se puede utilizar para predecir las actividades biológicas de los compuestos no probados3.
Entre muchos productos químicos nocivos, los disruptores endocrinos (ED) se han destacado como un grupo de tóxicos quepueden interferir en numerosos metabolismos relacionados con las hormonas en mamíferos, anfibios y peces 5,6. Se sabe que los ED inducen una variedad de efectos adversos, como cánceres y malformaciones, bloqueando o alterando las vías hormonales normales o activando señales anormales de síntesis/degradación hormonal. Como consecuencia, estos productos químicos que imitan las hormonas pueden perturbar los sistemas endocrinos, de tal forma que el desarrollo biológico y la reproducción de las poblaciones de animales de vida silvestre se vean obstaculizados. En particular, los efectos ecotoxicológicos de las EPI han sido ampliamente investigados en vertebrados acuáticos, que tienen estructuras receptoras hormonales casi idénticas a las de los mamíferos, incluidos los seres humanos. Debido a que todas las acciones hormonales ocurren a dosis bajas in vivo, predecir las posibles toxicidades de los candidatos a ED que utilizan pruebas rápidas de detección de sislico es fundamental para la salud pública y ambiental.
Los modelos QSAR basados en la toxicología de los EDse se han llevado a cabo utilizando descriptores 2D y 3D (conocidos como QSAR 2D y3D, respectivamente), que revelan las afinidades de unión de ligandos ED de estrógeno, andrógenos y receptores de progesterona 7. A pesar de las ventajas de alta precisión de 3D QSAR, en el que se consideran las interacciones conformacionales y electrostáticas, 2D QSAR conserva su propia robustez en algoritmos matemáticos directos, cálculos rápidos y cargas computacionales extremadamente bajas. Además, los modelos 2D-QSAR son flexibles para su uso en una amplia gama de aplicaciones, al tiempo que logran un rendimiento de predicción relativamente preciso.
La Caja de Herramientas QSAR de la OCDE es actualmente una de lasherramientas de software informático más utilizadas, proporcionando modelos QSAR 8,9de libre disposición y preconstruidos. Su generador de perfiles utiliza bases de datos descriptores 2D. Desde el lanzamiento de la primera versión en 2008, el software se ha aplicado en los campos de las industrias químicas y biológicas, la salud pública y la seguridad ambiental para el análisis total o parcial de los riesgos potenciales de los compuestos naturales y sintéticos, con intereses especiales en la carcinogénesis10,11,12, mutagenicidad13,14,15, y toxicidad para el desarrollo16. También se ha demostrado la aplicación a la toxicología acuática, centrándose en la bioacumulación y la biotransformación17.
La caja de herramientas QSAR ha demostrado ser útil para predecir la toxicidad a corto plazo de una amplia gama de productos químicos17,así como las afinidades de unión del receptor de estrógeno (ER) de los ED1. Sin embargo, las ecotoxicidades agudas de los ED en los vertebrados acuáticos no se han analizado utilizando QSAR Toolbox. En este estudio, se presenta un protocolo típico y fácil para realizar modelos QSAR sobre los efectos adversos agudos de los DE con un enfoque en las especies de peces. El estudio muestra que QSAR Toolbox es un software altamente accesible para calcular y predecir la letalidad/mortalidad de los vertebrados acuáticos para algunos EDs representativos. La figura 1 muestra el esquema general para el funcionamiento general del cuadro de herramientas QSAR. El flujo de trabajo que se muestra en la Figura 2 proporciona instrucciones sencillas sobre cómo operar el ensayo in silico para predecir la ecotoxicidad aguda de sustancias diana como los productos químicos disruptores endocrinos.
Protocol
Representative Results
Discussion
La versatilidad de la caja de herramientas QSAR de la OCDE como software analítico para la ecotoxicología se muestra aquí con un interés específico en los efectos adversos de los productos químicos disruptores endocrinos en los vertebrados acuáticos. Además, se demostró un protocolo simple y estándar para predecir la toxicidad aguda (96-h LC50) de 74 EDs representativos (Tabla1) para las especies de peces. Esto se logró aplicando módulos de creación de categorías, llenado de brechas de datos y generación de perfiles de ER integrados en el cuadro de herramientas QSAR (Figura1, Figura 2).
La correlación lineal entre el registro10LC50 y el registro10KOW con una pendiente negativa (como se muestra en la figura suplementaria S1)ha sido conocida durante mucho tiempo como una relación cuantitativa estándar en los análisis QSAR25,donde mayor toxicidad se muestra cuanto más hidrófobo es un producto químico dado. Como se puede ver en un simple cálculo, la relación matemática general que incluye la Ecuación S1 y la Ecuación S2 (InformaciónSuplementaria)es una expresión convertida de la siguiente función de potencia26:
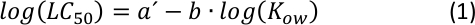

A partir de la gráfica de(Ecuación 2), caracterizar un rango intermedio de KOW26 puede ser posible ajustando los parámetros a y b, donde una cierta variación en hidrofobicidad (o hidrofilicidad) no cambia significativamente la punto final de toxicidad aguda.
Los análisis comparativos entre las predicciones computacionales y las observaciones experimentales sobre la LC50, como se muestra en la Figura 3 y la Figura 4,se han notificado típicamente en estudios de QSAR para diversos tóxicos acuáticos, incluyendo tensioactivos técnicos no iónicos27, fungicidas triazoles28y metabolitos plaguicidas21. Este tipo de validación retrospectiva proporciona información sobre hasta qué punto una determinada herramienta QSAR puede llegar en términos de rendimiento comparativo a los resultados experimentales. En este estudio de toxicidad aguda en peces, se demostró que QSAR Toolbox proporciona predicciones protectoras para más del 90% de los ED probados en todos los peces y en una sola especie, Pimephales promelas.
Se requiere una mayor identificación de los tres productos químicos atípicos en la Figura 3 y la Figura 4, que mostraron una LC50 pronosticada más alta en promedio y como mínimo, respectivamente. En primer lugar, el 3′,5,7-trihydroxy-4′,6-dimethoxyisoflavone es un tipo de flavonoide (más específicamente, una isoflavona), que se considera generalmente seguro y se utiliza en productos farmacéuticos herbarios; sin embargo, todavía tiene preocupaciones relacionadas con el estrógeno29 y puede causar toxicidad aguda probablemente a través de desacoplamiento de fosforilación oxidativa30. A continuación, el 1,4-benzenediol, llamado hidroquinona, es un compuesto fenólico que puede desencadenar una respuesta inmune no específica y citotóxica en los peces31. Finalmente, el 4-hexylphenol se ha sabido para exhibir suficiente actividad estrogénica positiva para ser clasificado como un ED32. Se ha estudiado bien que la razón principal de la toxicidad aguda de la hidroquinona es el ciclo de reducción-oxidación (redox). La hidroquinona se oxida a benzoquinona y se reduce de nuevo a semiquinona o hidroquinona repetidamente, con cofactores que agotan y generando especies reactivas de oxígeno33. Los otros dos productos químicos pueden requerir investigaciones más profundas para revelar sus mecanismos de acción en ecotoxicidad aguda utilizando enfoques de acoplamiento molecular como el utilizado por Panche et al.34, que no pueden ser cubiertos por la caja de herramientas QSAR.
Los ED interfieren con el sistema endocrino principalmente a través de interacciones fisicoquímicas con receptores de esteroides como los receptores de estrógeno y andrógenos, que son de considerable interés en los estudios de modelado QSAR35. Teniendo en cuenta esto, QSAR Toolbox es robusto en términos de clasificación fácil y rápida de afinidades de unión er para un conjunto de productos químicos basados únicamente en los descriptores 2D de las estructuras moleculares. Cuando este sistema de generador es de perfiles de ER se aplicó a nuestra lista de EDs, no se encontró una correlación clara entre la afinidad de unión de ER y la hidrofobicidad (FiguraSuplementaria S2). Este resultado puede explicarse por el hecho de que la formación de un complejo esteroide-receptor no es una consecuencia directa de una contribución de unión hidrófoba, sino que debe ir acompañada de un cambio de conformación en la estructura del receptor del sitio activo36. La unión del receptor también puede deberse a la unión al hidrógeno y al apilamiento de la s.
Además, la posición de cada grupo químico en la molécula puede afectar a la unión del receptor, incluso si la hidrofobicidad y el número de receptores-bonos de hidrógeno siguen siendo los mismos. En segundo lugar, el generador de perfiles de ER produjo tendencias contrarias entre los niveles medios de LC50 previstos y experimentales con una creciente afinidad de unión de ER (Figura5). Esto puede deberse a que la letalidad de los padres en una prueba de toxicidad aguda no se debe a la unión a Urgencias, sino más bien a la narcosis en la mayoría de los casos, o al ciclo de redox en el caso de la hidroquinona. Por ejemplo, se requiere un análisis más extenso, incluida la toxicidad crónica, para que un conjunto más grande de EDs defina las limitaciones predictivas de la versión actual de QSAR Toolbox.
Esta investigación preliminar también puede tener implicaciones para la salud pública porque los esteroides (andrógenos, estrógenos, progestinas, y corticoides) y sus receptores exhiben estructuras macromoleculares similares o incluso idénticas a través de vertebrados5. Estos tipos de sistemas de señalización endocrino análogos pueden funcionarutilizando un mecanismo común en eventos clave de los DATOS Electrónicos 5. Sin embargo, se requieren metodologías adicionales y complementarias para iluminar este vasto y complejo aspecto [por ejemplo, mediante la realización de modelos computacionales de absorción, distribución, metabolismo y excreción (ADME) y/o resultados adversos vía (AOP)]38. Además, debido a que la mayoría de las preocupaciones científicas y públicas planteadas sobre los efectos adversos de los DEE están relacionadas con sus toxicidades crónicas, mejorar las bases de datos y algoritmos en la caja de herramientas QSAR y producir ecotoxicología fiable a largo plazo las predicciones de los DE Son necesarias.
Este artículo demuestra la aplicación de QSAR Toolbox para comparar los valores ecotoxicológicos de LC50 para peces con los valores de registro10Kow de EDs. A lo largo del protocolo, resulta en relaciones débiles entre los dos parámetros, ya que ha sido revelado por estudios anteriores (por ejemplo, Kim et al.39) que el registro10Kow no es un buen predictor directo de LC50acuático. A pesar de esta limitación, este protocolo proporciona una revisión general o “viñeta” para describir cómo utilizar el panel para un propósito determinado, ya que es una aplicación válida para utilizar qSAR Toolbox para investigar correlaciones entre LC50 (o enlace ER afinidad) y el registro de10Kow, o como una herramienta para la detección rápida de ecotoxicidad aguda. No obstante, cabe señalar que (1) iluminar el vínculo entre la unión del receptor de estrógeno y la toxicidad crónica, en lugar de la toxicidad aguda (letalidad), es más relevante para que se puedan encontrar correlaciones más claras, y (2) el receptor de andrógenos, con la de estrógeno, también juega un papel crítico en la toxicidad reproductiva. Por lo tanto, es necesario que la versión futura del cuadro de herramientas QSAR mejore las funciones de predicción a la luz de esos dos puntos.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Esta investigación fue apoyada por la subvención del Consejo Nacional de Investigación de Ciencia y Tecnología (NST) por el gobierno de Corea del Sur (MSIP) (No. CAP-17-01-KIST Europe) y el Proyecto 11911.
Materials
| Adobe Acrobat Reader DC | Adobe Systems Software Ireland Limited | NA | Required to view prediction and category report |
| Computer | System: Microsoft Corporation | NA | Recommended system properties: (i) system type: 64 bit, Microsoft Windows 7 or newer, (ii) processor: I5 at 2.4 GHz or faster processor or equivalent AMD CPU, (iii) Installed memory (RAM): 6 GB of RAM, (iv) Hard Disk Drive (HDD): 20 GB free hard drive space |
| Microsoft Editor | Microsoft Corporation | NA | Required to upload a substance list of CAS numbers (batch mode) to the OECD QSAR Toolbox as .txt file (text file) |
| Microsoft Excel 2016 | Microsoft Corporation | NA | Required to export data from OECD QSAR Toolbox as .cvs, .xls or .xlsx files |
| OECD QSAR Toolbox version 4.0 or newer | Organisation for Economic Co-operation and Development |
NA | Required to run OECD QSAR Toolbox Automated Workflows; free download: https://qsartoolbox.org/download/ |
| OriginPro 9 | OriginLab Corporation | NA | Optional program for data analysis; similar tools possible |
References
- Najarian, K., Najarian, S., Gharibzadeh, S., Eichelberger, C. N. . Systems Biology and Bioinformatics: A Computational Approach. , (2009).
- Fujita, T., Iwasa, J., Hansch, C. A new substituent constant, π, derived from partition coefficient. Journal of the American Chemical Society. 86, 5175-5180 (1964).
- Roy, K., Kar, S., Das, R. N. . Understanding the Basics of QSAR for Applications in Pharmaceutical Sciences and Risk Assessment. , (2015).
- Raies, A. B., Bajic, V. B. In silico toxicology: computational methods for the prediction of chemical toxicity. WIREs Computational Molecular Science. 6, 147-172 (2016).
- Hayes, T. B. Welcome to the revolution: integrative biology and assessing the impact of endocrine disruptors on environmental and public health. Integrative Compuational Biology. 45, 321-329 (2005).
- Schug, T. T., et al. Minireview: endocrine disruptors: past lessons and future directions. Molecular Endocrinology. 30, 833-847 (2016).
- Devillers, J., Marchand-Geneste, N., Carpy, A., Porcher, J. M. SAR and QSAR modeling of endocrine disruptors. SAR QSAR Environmental Research. 17, 393-412 (2006).
- Dimitrov, S. D., et al. QSAR Toolbox – workflow and major functionalities. SAR QSAR Environmental Research. 27, 203-219 (2016).
- Yordanova, D., et al. Automated and standardized workflows in the OECD QSAR Toolbox. Computational Toxicology. 10, 89-104 (2019).
- Mombelli, E., Devillers, J. Evaluation of the OECD (Q)SAR Application Toolbox and Toxtree for predicting and profiling the carcinogenic potential of chemicals. SAR QSAR Environmental Research. 21, 731-752 (2010).
- Devillers, J., Mombelli, E., Samsera, R. Structural alerts for estimating the carcinogenicity of pesticides and biocides. SAR QSAR Environmental Research. 22, 89-106 (2011).
- Li, C., et al. Identifying unknown by-products in drinking water using comprehensive two-dimensional gas chromatography-quadrupole mass spectrometry and in silico toxicity assessment. Chemosphere. 163, 535-543 (2016).
- Devillers, J., Mombelli, E. Evaluation of the OECD QSAR Application Toolbox and Toxtree for estimating the mutagenicity of chemicals. Part 1. Aromatic amines. SAR QSAR Environmental Research. 21, 753-769 (2010).
- Devillers, J., Mombelli, E. Evaluation of the OECD QSAR Application Toolbox and Toxtree for estimating the mutagenicity of chemicals. Part 2. α-β unsaturated aliphatic aldehydes. SAR QSAR Environmental Research. 21, 771-783 (2010).
- Kulkarni, S. A., Barton-Maclaren, T. S. Performance of (Q)SAR models for predicting Ames mutagenicity of aryl azo and benzidine based compounds. Journal of Environmental Science and Health Part C Environmental Carcinogenesis & Ecotoxicology Reviews. 32, 46-82 (2014).
- Craig, E. A., Wang, N. C., Zhao, Q. J. Using quantitative structure-activity relationship modeling to quantitatively predict the developmental toxicity of halogenated azole compounds. Journal of Applied Toxicology. 34, 787-794 (2014).
- Tebby, C., Mombelli, E., Pandard, P., Péry, A. R. Exploring an ecotoxicity database with the OECD (Q)SAR Toolbox and DRAGON descriptors in order to prioritise testing on algae, daphnids, and fish. Science of the Total Environment. 409, 3334-3343 (2011).
- Mombelli, E. Evaluation of the OECD (Q)SAR Application Toolbox for the profiling of estrogen receptor binding affinities. SAR QSAR Environmental Research. 23, 37-57 (2012).
- Verhaar, H. J. M., van Leeuwen, C. J., Hermens, J. L. M. Classifying environmental pollutants. 1: structure-activity relationships for prediction of aquatic toxicology. Chemosphere. 25, 471-491 (1992).
- Enoch, S. J., Hewitt, M., Cronin, M. T. D., Azam, S., Madden, J. C. Classification of chemicals according to mechanism of aquatic toxicity: an evaluation of the implementation of the Verhaar scheme in Toxtree. Chemosphere. 73, 243-248 (2008).
- Burden, N., Maynard, S. K., Weltje, L., Wheeler, J. R. The utility of QSARs in predicting acute fish toxicity of pesticide metabolites: a retrospective validation approach. Regulatory Toxicology and Pharmacology. 80, 241-246 (2016).
- Nendza, M., et al. Screening for potential endocrine disruptors in fish: evidence from structural alerts and in vitro and in vivo toxicological assays. Environmental Sciences Europe. 28, 26 (2016).
- Roncaglioni, A., Piclin, N., Pintore, M., Benfenati, E. Binary classification models for endocrine disrupter effects mediated through the estrogen receptor. SAR QSAR Environmental Research. 19, 697-733 (2008).
- Sosnovcová, J., Rucki, M., Bendová, H. Estrogen receptor binding affinity of food contact material components estimated by QSAR. Central European Journal of Public Health. 24, 241-244 (2016).
- Walker, J. D., Dearden, J. C., Schultz, T. W., Jaworska, J., Comber, M. H. I., Walker, J. D. QSARs for New Practitioners. QSARs for Pollution Prevention, Toxicity Screening, Risk Assessment, and Web Applications. , (2003).
- Sánchez-Bayo, F. From simple toxicological models to prediction of toxic effects in time). Ecotoxicology. 18, 343-354 (2009).
- Sjöström, M., Lindgren, &. #. 1. 9. 7. ;., Uppgård, L. L., Chen, F., Schüürmann, G. Joint Multivariate Quantitative Structure-Property and Structure-Activity Relationships for a Series of Technical Nonionic Surfactants. Quantitative Structure-Activity Relationships in Environmental Sciences-VII. , (1997).
- Ding, F., Guo, J., Song, W., Hu, W., Li, Z. Comparative quantitative structure-activity relationship (QSAR) study on acute toxicity of triazole fungicides to zebrafish. Chemistry Ecology. 27, 359-368 (2011).
- Galati, G., O’Brien, P. J. Potential toxicity of flavonoids and other dietary phenolics: significance for their chemopreventive and anticancer properties. Free Radical Biology in Medicine. 37, 287-303 (2004).
- Russom, C. L., Bradbury, S. P., Broderius, S. J. Predicting modes of action from chemical structure: acute toxicity in the fathead minnow (Pimephales promelas). Environmental Toxicology and Chemistry. 16, 948-967 (1997).
- Taysse, L., Troutaud, D., Khan, N. A., Deschaux, P. Structure-activity relationship of phenolic compounds (phenol, pyrocatechol and hydroquinone) on natural lymphocytotoxicity of carp (Cyprinus carpio). Toxicology. 98, 207-214 (1995).
- Nishihara, T., et al. Estrogenic activities of 517 chemicals by yeast two-hybrid assay. Journal of Health Science. 46, 282-298 (2000).
- Bolton, J. L., Trush, M. A., Penning, T. M., Dryhurst, G., Monks, T. J. Role of quinones in toxicology. Chemical Research in Toxicology. 13, 135-160 (2000).
- Panche, A. N., Diwan, A. D., Chandra, S. R. Flavonoids: an overview. Journal of Nutritional Science. 5, e47 (2016).
- Li, J., Gramatica, P. QSAR classification of estrogen receptor binders and pre-screening of potential pleiotropic EDCs. SAR QSAR Environmental Research. 21, 657-669 (2010).
- Bohl, M. . Molecular Structure and Biological Activity of Steroids. , (2017).
- Kaminuma, T., Takai-Igarashi, T., Nakano, T., Nakata, K. Modeling of signaling pathways for endocrine disruptors. BioSystems. 55, 23-31 (2000).
- Lillicrap, A., et al. Alternative approaches to vertebrate ecotoxicity tests in the 21st century: a review of developments over the last 2 decades and current status. Environmental Toxicology and Chemistry. 35, 2637-2646 (2016).
- Kim, J. W., et al. Acute toxicity of pharmaceutical and personal care products on freshwater crustacean (Thamnocephalus platyurus) and fish (Oryzias latipes). Journal of Toxicological Sciences. 34, 227-232 (2009).

