מתרגם הריבוכמה אהדה לטיהור (מלכודת) כדי לחקור את התפתחות השורש של הספר הבסיסי בקנה מידה ספציפי לסוג תא
Summary
מתרגם הריבוכמה אהדה (מלכודת) מציעה את האפשרות לנתח תוכניות התפתחותיות עם עיבוד מינימלי של איברים ורקמות. הפרוטוקול מניב RNA באיכות גבוהה מתאים המיועדים עם חלבון פלורסנט ירוק (GFP)-יחידת משנה ריבוזומבית. כלים לניתוח במורד הזרם, כגון רביעיית-PCR או RNA-seq, חושפים פרופילי ביטויים ספציפיים לסוג תאים.
Abstract
במאמר זה, אנו מעניקים הוראות הידיים על מנת להשיג נתונים טרנסלסיים מסוגים שונים של השורש החדש של שורש השיטה באמצעות הריבותרגום האהדה (השמנה) שיטה ומיטוב רצופות הכנה לספרייה נמוכה.
כחומר מתחיל, אנו מעסיקים קווי צמחים לבטא GFP-tagged חלבון RPL18 באופן ספציפי סוג תא על ידי שימוש ביזמים נאותים. לפני החילוץ והפקת ה-RNA, הרקמה מוקפאת, השומרת על שלמות הרקמה ובמקביל מאפשרת ביצוע מחקרי סדרת זמן ברזולוציה גבוהה. בעיקר, מבנים קיר התא נשארים ללא פגע, אשר הוא חיסרון משמעותי בהליכים חלופיים כגון מיון המופעל על-ידי תאים בעלי קרינה פלואורסצנטית מבוסס הגישה הנשענת על רקמות לבודד אוכלוסיות תאים נפרדות. בנוסף, אין קיבעון רקמות הכרחי כמו לכידת לייזר מבוססי מיקרוניתוח טכניקות, אשר מאפשר RNA באיכות גבוהה לקבל.
עם זאת, דגימה מאוכלוסיות משנה של תאים ורק בידוד polysome הקשורים RNA מגביל באופן חמור תשואות RNA. לכן, יש צורך להחיל שיטות רגישות מספיק לספרייה לרכישת נתונים מוצלחת על-ידי RNA-seq.
השמנה מציעה כלי אידיאלי עבור מחקר הצמח כמו תהליכים התפתחותיים רבים כרוך תאים הקשורים לקיר ומכני איתות. השימוש ביזמים למקד אוכלוסיות תאים ספציפיות הוא גישור הפער בין איבר ורמת תא בודד, כי בתורו סובלים מרזולוציה קטנה או עלויות גבוהות מאוד. כאן, אנו להחיל את המלכודת ללמוד תקשורת תא התא במערך השורש הצדדי.
Introduction
מונע על ידי יישום הגוברת של טכניקות רצף הדור הבא, רזולוציה מרחבית בביולוגיה התפתחותית יכול להיות מוגבר. מחקרים עכשוויים מכוונים לניתוח רקמות למטה לסוגי תאים מיוחדים, אם לא ברמה אחת של תא1,2,3,4. למטרה זו, שפע של שיטות שונות המציאו במשך 50 השנים האחרונות (ראה איור 1a)5,6,7,8,9,10,11,12,13,14,15.
כלים רבים במדעי הצמח הינם עיבודים של טכניקות שהיו חלוץ במחקר בעלי חיים. זה לא המקרה לשיטה שאנו מציגים בפרוטרוט כאן. ב 2005, מצויד ברקע חזק בתרגום חלבון, מעבדה ביילי Serres להנדס ריבוזומלי חלבונים עבור האהדה הבאה טיהור16. לפיכך, הם יכולים להימנע מיצירת פרופיל רב פעמי ומעמיק לעבודה, אשר מבוסס על מעבר באמצעות שיפוע של סוכות ושימש להערכת תרגום הריבוזומים מאזשנות ה-60,18. השיטה מאז המכונה ריבולtional טיהור אהדה (מלכודת)16. לאחר לימודי העברה מוצלחים בצמחים, מלכודת היימן ואח ‘ לבעלי חיים19 ואחרים הרחיבו את היישום שלה ל-20שמרים, דרוסופילה21, קסנפוס22 ו-דג זברה23,24.
למרות השינוי הגנטי של מערכת המודל הוא תנאי מוקדם עבור השמנה, אשר מגביל את היישום שלה מינים הניתנים לטרנספורמציה גנטית, ניתן בו לרתום התנגדות זו כדי למקד את קבוצות המשנה של תאים, כי הם בעלי עניין מיוחד, ובכל זאת קשה מאוד לבודד מן הרקמה בשלמותה/איבר25 (למשל בצמחים, כל התאים מוחזקים במקומם דרך קירות התא היוצרים את הבסיס של שלד הידרוסטטי26. כדי לשחרר תא צמח ממטריקס זה, מדענים יש גם לחתוך פיזית את התא מתוך הרקמה המקיפה שלה דרך לכידת לייזר מיקרוניתוח (LCM)27 או ביצע העיכול אנזימטי של קירות התא28. בין התאים האחרונים, מה שנקרא פרוטואופרציה, האוכלוסייה של העניין היא בעלת תווית בדרך כלל וניתן להפריד באמצעות מיון תא המופעל על ידי קרינה פלואורסצנטית (FACS)7. LCM בדרך כלל דורש מדגם להיות קבוע מוטבע שעווה, אשר בסופו של דבר מתדרדר את האיכות של ה-RNA שלה29. FACS מבוססי שיטות להניב RNA באיכות גבוהה, אבל התהליך של protoplasting עצמו מציג הבדלים ביטוי גנים30 ורקמות עם קירות תא משני שונה ועבה הם לשמצה קשה לטפל. כמו-כן, מניחים על תהליכים התפתחותיים רבים בצמחים להסתמך על אותות מכנית ולכן השלמות של קיר התא היא בעלת חשיבות עליונה31. שתי שיטות, אשר להשתמש בקיצור דרך לעקוף בידוד התא על ידי הפעלה ברמה של נוקלאושני, הם מיון גרעיני המופעל באמצעות קרינה פלואורסצנטית (מאווררים) ובידוד של גרעינים מתויג סוגי תאים ספציפיים (שלמות). כמו במלכודת, הם משתמשים ביזמים ספציפיים לסוג תא כדי לסמן גרעינים, שלאחר מכן לקבל מועשר באמצעות מיון או למשוך למטה, בהתאמה8,15. האתגר העיקרי עבור כל הגישות הללו היא לקבל די חומר RNA מקבוצות המשנה של תאים ברקמה. כמו המלכודת לוכדת רק חלק של RNAs הסלולר, אוסף לדוגמה הוא צוואר בקבוק משמעותי. לפיכך, דרושות במיוחד פרוטוקולי הכנה לספריה הרגישים כדי להפיק נתונים באיכות גבוהה מכמויות קלט נמוכות.
מאז הקמתה, המלכודת כבר נעשה שימוש בשילוב עם מיקרו מערכים DNA או, כמו עלויות רצף ירד באופן משמעותי בשנים האחרונות, RNA-seq10,32,33. הרבה שאלות מחקר כבר הובהר כפי שנסקרו בסאבלוק ואח ‘.34. אנחנו משוכנעים כי דוחות יותר יעברו בשנים הקרובים כמו הטכניקה היא מאוד תכליתי בעת שילוב יזמים שונים כדי למקד סוגי תאים ספציפיים. בסופו של דבר, זה ייעשה גם בדרך inducible, והוא עשוי להיות משולב עם בדיקת תגובת הצמח לגורמי סטרס ביוטיים ובין רבים. בנוסף, כאשר קווים הטרנסגניים יציבה אינם זמינים, מערכות שעירות ביטוי השורש יש גם בהצלחה לבצע מלכודת עגבניות ותרופות35,36.
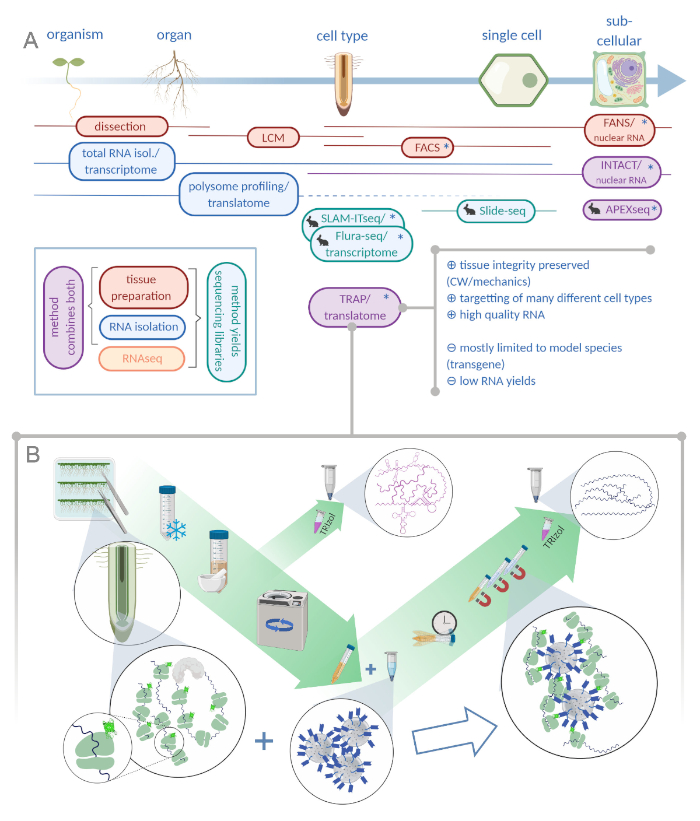
איור 1: מתרגם הריבוכמה אהדה (השמנה) משלים את תיק הניתוח “omics”. A. הגדלת רמות של דיוק אנליטי, עד תא יחיד או אפילו ברזולוציה subcellular יכול להיות מושגת על ידי שפע של שיטות או שילובים מהם. התוכנית מעניקה סקירה של הכלים הזמינים כעת בתחום הצמח ובעלי החיים. אוסף רקמות ברזולוציה התאית יכול להיות מושגת על ידי פרוטוקולים כמו LCM או FACS, אשר מצמידים לאחר מכן להמרה סטנדרטי או polysome בדיקת פרופיל/translatome. מלכודת ושלמות לשלב לכידת רקמות בידוד RNA כפי שהם מבוססים על התיוג האפיפי. עם זאת, דגימות שלמות רק גרעיני התא ומהווה, לכן, מקרה מיוחד של ניתוח ההמרה. סמל ארנב קטן מסמן שיטות שפותחו לאחרונה בשדה בעלי חיים: בעוד סלאם-itseq ו flura-seq להסתמך על יקוד מטבולית של rnas מחדש עם שינוי אורציל בתאים ביטוי האנזים מאשרני, שקופית-seq עושה שימוש בשקופית זכוכית מצופה עם ברקודים DNA המספקים מידע מיקומני ב הגישה תיוג הקרבה מלווה איפקס-seq כדי לדגום RNAs בתאי subcellular ספציפיים. בעיקר, רזולוציה מוגברת לעתים קרובות דורש את הדור של החומר הטרנסגניים (כוכביות) ושיטות אלה משמשות בעיקר עבור מינים מודל. המלכודת מתאימה במיוחד עבור מדעי הצמח לימודי מעורבים קיר תא (CW) או איתות מכונאי, כמו גם מינים תאים שקשה לשחרר מתוך מטריצה CW שלהם. ב. מפורט שלבי המעבדה הרטובה של הליך המלכודת: השתילים מבטאים חלבון מתויג מסוג gfp בסוגים שונים של תאים (כגון אנדודרסיס שורש) גדלים על מנות פטרי במשך שבעה ימים וחומר שורש שנקטפו על ידי הקפאת ההקפאה. מדגם סה כ בקרת RNA נאסף מתוך תמצית גולמי הומוגניים לפני הפלטינג פסולת באמצעות צנטריפוגה. חרוזים אנטי-GFP מגנטיים נוספים לתמצית נקי לבצע immunoprecipitation. לאחר הדגירה ושלושה צעדים לשטוף, ה-RNA polysome הקשורים (מלכודת/polysome RNA) הוא קיבל ישירות באמצעות כלורופורם פנול-הכלורופורם. LCM: לכידת לייזר מיקרודיסקציה, FACS/אוהדים: תא מופעל על-ידי קרינה פלואורסצנטית/מיון גרעיני, איפקס-seq: שיטה המבוססת על הנדסה ascorbate peroxidase, שלמות: בידוד של גרעינים מתויג סוגי תאים ספציפיים, סלאם-ITseq: thiol (SH)-האלקילציה מקושרת עבור רצף חילוף החומרים של RNA ברקמות, Flura-seq: fluorouracil-התווית ברצף RNA (נוצר עם Biorender.com) אנא לחץ כאן כדי להציג
המטרה של מאמר זה היא לספק תיאור מפורט של שיטת TRAP, כדי להדגיש שלבים קריטיים ולספק הדרכה לשיטת הכנה לספריה אפשרית.
ניסוי המלכודת הגנרית יהיה למעשה מורכב השלבים הבאים (ראה גם איור 1B): (1) הכנת חומר הצמח כולל שיבוט של ריבוכמה תיוג בניית, הייצור קו טרנסגניים ובחירה, גדל ו bulking של זרעים, עיקור וציפוי, ומתח יישום/טיפול (אופציונלי) ו לקצור רקמות; (2) אימונופיקציה, כולל המגון רקמות וניקוי של תמצית גסה, שטיפת חרוזים ואימונופיקציה, ולשטוף את הצעדים; (3) הערכת הוצאת רנ א ואיכות; ו (4) הכנה לספרייה.
שורש arabidopsis כבר מערכת מודל ללמוד פיתוח הצמח מאז המבוא שלה כמפעל דגם37,38. כאן, היישום של המלכודת הוא לראווה בהקשר של פיתוח השורש הצמח לרוחב. בצמחים, הצטברות של מערכת השורש כולה מסתמך על ביצוע של תוכנית זו ולכן חשוב מאוד להישרדותו של האורגניזם39. באראידאופזיס, שורשי הצדדיים מקורם ברקמת כלקרום העור השוכנת ליד כלי הדם ולכן הוא כינה את המוט הקרום (xpp; ראה איור 2 ג)40. כמה תאים XPP, אשר נמצאים עמוק בתוך השורש, לרכוש זהות תא מייסד, על גורם הורמונלי מקומי, להתחיל להתרבות על ידי נפיחות וחילוק אנטיליבאלי41. עם זאת, בשל נוכחותם של מטריצת קיר נוקשה התא, תהליך זה מפעילה לחץ מכני על הרקמות הסובבות. בפרט, אנדודראמיס הנמצא במעלה מושפע, כפי שהוא בדרך של ציר צמיחה שורש לרוחב42,43,44. אכן, החדש הקמתה החדשה יהיה צריך לצמוח דרך התאים האנדודרסטיות האלה (איור 2c2) בעוד תאי הקליפה והאפידרמיס הם רק דחפו הצידה הפריבדיום לבסוף לצאת45,46. העבודה האחרונה במעבדה שלנו הראתה כי האנדודרסטיות תורמת באופן פעיל כדי להכיל את השגשוג בקרום הלב. חסימה ממוקדת של איתות הורמונלי אנדועורי מספיק כדי לעכב גם את החלוקה הראשונה מאוד בתאים XPP47. לפיכך, התקשורת האנדוציציסטית מהווה מחסום מוקדם מאוד לפיתוח השורש הרוחבי בערידרופזיס. עם זאת, לא ידוע כיצד מבוצע הוצלב הזה. כדי לפתור את התעלומה הזאת, אנחנו בחרנו את הגישה השמנה-seq כדי למקד את התאים XPP וכלי העורי. כדי להעשיר את התאים בתוכנית השורש לרוחב, אנו לחקה את הגורם ההורמונלי על ידי החלת באופן שונה היישום מערכת הקלה אנלוגי (1-נפלוג חומצה אצטית, NAA)48, אשר באותו זמן מותר לפתור באופן זמני את השלב הראשוני של היווצרות השורש הצדדי.
Protocol
Representative Results
Discussion
אימות תבנית הלוקליזציה של RPL18
חיוני כדי למנוע פרשנות של נתונים מכל ניסוי השמנה היא תבנית הביטוי הנכון של יחידת המשנה הריבוזומתית המתויג. לכן, שילוב של GFP כמו תג אפירופה ל RPL18 מאוד באלגנטיות מאפשר אימות של דפוס הביטוי הרצוי ברציפות, מתחת השבר polysome מן הרקמה אותה. גישות פולשנית יותר ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
היינו רוצים להודות ז ‘ אן קלוד וואלזר של המרכז גיוון גנטי ציריך עבור ייעוץ מומחה מכריע בשלב מוקדם של פרויקט זה. העבודה במעבדה ורמר נתמך על ידי מענק מפרופסור SNF (PP00P3_157524) ו R’ צייד ציוד מענק (316030_164086) מן הקרן הלאומית המדע השוויצרי (SNF) הוענק JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

