Radiografia de Nêutrons e Tomografia Computadorizada de Sistemas Biológicos no Reator de Isótopos de Alto Fluxo do Oak Ridge National Laboratory
Summary
Este manuscrito descreve um protocolo para radiografia de nêutrons e tomografia computadorizada de amostras biológicas usando uma linha de luz CG-1D do High Flux Isotope Reactor (HFIR) para medir um implante metálico em um fêmur de rato, um pulmão de camundongo e um sistema raiz/solo de planta herbácea.
Abstract
Os nêutrons têm sido historicamente usados para uma ampla gama de aplicações biológicas empregando técnicas como espalhamento de nêutrons a baixo ângulo, eco de spin de nêutrons, difração e espalhamento inelástico. Ao contrário das técnicas de espalhamento de nêutrons que obtêm informações no espaço recíproco, a imagem de nêutrons baseada em atenuação mede um sinal no espaço real que é resolvido na ordem de dezenas de micrômetros. O princípio da imagem por nêutrons segue a lei de Beer-Lambert e é baseado na medição da atenuação de nêutrons em massa através de uma amostra. Maior atenuação é exibida por alguns elementos leves (mais notavelmente, hidrogênio), que são os principais componentes de amostras biológicas. Agentes de contraste como deutério, gadolínio ou compostos de lítio podem ser usados para aumentar o contraste de maneira semelhante à que é feita em imagens médicas, incluindo técnicas como imagens ópticas, ressonância magnética, raios-X e tomografia por emissão de pósitrons. Para sistemas biológicos, a radiografia de nêutrons e a tomografia computadorizada têm sido cada vez mais utilizadas para investigar a complexidade da rede radicular subterrânea de plantas, sua interação com os solos e a dinâmica do fluxo de água in situ. Além disso, esforços para entender detalhes de contraste em amostras animais, como tecidos moles e ossos, têm sido explorados. Este manuscrito se concentra nos avanços em bioimagem de nêutrons, como preparação de amostras, instrumentação, estratégia de aquisição de dados e análise de dados usando a linha de luz CG-1D de imagem de nêutrons do High Flux Isotope Reactor. As capacidades acima mencionadas serão ilustradas usando uma seleção de exemplos em fisiologia vegetal (planta herbácea/raiz/sistema solo) e aplicações biomédicas (fêmur de rato e pulmão de camundongo).
Introduction
O princípio da radiografia de nêutrons (nR) baseia-se na atenuação dos nêutrons através da matéria que atravessam. Ao contrário dos raios X que são espalhados pela nuvem de elétrons de um átomo, os nêutrons podem ser absorvidos ou espalhados por seu núcleo. Os nêutrons são sensíveis a elementos leves, como o hidrogênio (H), e podem, consequentemente, ser utilizados para aplicações biológicas radiográficas, como tecidos animais 1,2,3,4,5,6,7 ou humanos 8,9 e sistemas solo/radiculares abaixo do solo 10,11,12,13,14 ,15. A imagem por nêutrons é uma técnica complementar à imagem radiográfica, capaz de detectar elementos pesados16,17,18. O nR baseado em atenuação é governado pelos coeficientes de atenuação linear dos materiais dentro da amostra e pela espessura da amostra, conforme descrito pela lei de Beer-Lambert, que afirma que o feixe transmitido é diretamente proporcional à quantidade de material e ao comprimento do caminho através do material. Assim, a transmitância, T, pode ser calculada como:
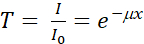 (1)
(1)
onde I 0 e I são, respectivamente, as intensidades incidente e transmissão do feixe; μ e x são o coeficiente de atenuação linear e a espessura de uma amostra homogênea, respectivamente. O coeficiente de atenuação μ é dado por:
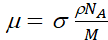 (2)
(2)
onde σ é a seção transversal de atenuação de nêutrons da amostra (espalhamento e absorção), ρ é sua densidade, NA é o número de Avogadro e M é sua massa molar.
O contraste na radiografia de amostras biológicas usando nêutrons de baixa energia (ou seja, energias abaixo de 0,5 eV) é principalmente devido a uma mudança na densidade de H (para uma espessura de amostra fixa). Isso se deve à probabilidade de interação de um nêutron com o núcleo H, que é maior do que com outros núcleos presentes em amostras biológicas, e ao fato de que a densidade do átomo H é primordial, pois é o átomo mais abundante em amostras biológicas.
Desde seus primórdios, a nR e a tomografia computadorizada por nêutrons (nCT) têm sido extensivamente utilizadas para aplicações em materiais e engenharia19,20,21,22,23. Os primeiros experimentos de demonstração da sensibilidade de nêutrons ao H em amostras biológicas começaram em meados da década de 195024 com as medidas de espécimes de plantas. O trabalho continuou até a década de 1960 com, por exemplo, a radiografia de tórax humano25 ou deratos26, na qual foi explorado o uso de agentes de contraste, como o óxido de gadolínio (Gd2O3). Além disso, hipotetizou-se que o contraste no tecido tumoral humano versus tecido normal era devido a um aumento local no conteúdo de H. Durante esses ensaios iniciais, concluiu-se que o aumento do fluxo de nêutrons e da resolução espacial melhoraria a qualidade da nR e provavelmente aumentaria sua popularidade como uma técnica complementar para aplicações industriais ou biomédicas. Os estudos mais recentes compreendem medidas de nR e nCT realizadas em espécimes de tecido cancerígeno1 e cortes de órgãos de animais 2,3,27 para aplicações biomédicas e forenses.
Localizado no Oak Ridge National Laboratory, Oak Ridge, TN, o High Flux Isotope Reactor (HFIR) é uma poderosa fonte de nêutrons que produz nêutrons por reação de fissão. Esses nêutrons têm energias da ordem de 2 MeV e são “resfriados” na piscina do reator por reações cinéticas com água pesada para atingir energias da ordem de 100-300 eV. A otimização de um experimento de nêutrons, seja espalhamento ou imagem, começa com a compreensão da fonte de nêutrons e das propriedades da linha de luz, como sua intensidade de feixe, distribuição de energia e o efeito de fundo (nêutrons rápidos, nêutrons atrasados, raios gama). No hall de guia frio HFIR, onde a linha de luz de imagem está localizada, os nêutrons são ainda mais “resfriados” por interações cinéticas com um moderador H líquido. Eles são então transportados em um sistema de guia curvo para longe da linha de visão da fonte, eliminando assim nêutrons rápidos e poluição gama. Como ilustrado na Figura 1, a linha de luz CG-1D para imagem de nêutrons28,29 é colocada em uma guia fria, implicando que a faixa de energia de nêutrons varia de alguns meV a algumas dezenas de eV (neste caso, o comprimento de onda de nêutrons utilizável correspondente varia de 0,8 a 10 Å) com um fluxo na faixa de 10a 7 n/(cm2∙s) na posição da amostra. Um sistema motorizado de abertura/difusor define a geometria pinhole do instrumento de imagem. Os nêutrons percorrem uma distância de 6,59 m em um tubo de voo cheio de hélio (He) com janelas de alumínio (Al) em cada extremidade. Os tubos de voo são usados para transportar nêutrons, limitando a dispersão do ar de tal forma que a perda na intensidade do feixe seja mínima. Para as medidas descritas neste manuscrito, o difusor é feito de um nanopó de óxido de alumínio (Al2O3) de 1 mm de espessura e 50 nm, envolto em um recipiente de Al. O difusor reduz os artefatos do feixe provenientes da guia de nêutrons (que são ampliados pela geometria pinhole de uma linha de luz de imagem), caso contrário, flutuações horizontais e verticais acentuadas de intensidade são visíveis na radiografia e a normalização dos dados torna-se um desafio. Para os experimentos ilustrados aqui, nêutrons são convertidos em luz usando um fluoreto de lítio-6 de 25 μm de espessura/fósforo de sulfeto de zinco (6LiF/ZnS:Ag).
A otimização da colimação depende da posição da amostra para o detector, da resolução espacial necessária e do tempo de aquisição. Quando a amostra fica a poucos cm de distância do cintilador, altas colimações (L/D acima de 800, onde L é a distância da abertura do orifício de diâmetro, D, e do detector) produzem melhor resolução espacial ao custo do fluxo de nêutrons. Baixa colimação (L/D abaixo de 800) é preferível para estudos dinâmicos in situ quando a resolução temporal prevalece sobre a espacial. Para as medidas descritas neste manuscrito, L/D e resolução espacial foram de aproximadamente 355 e 75 μm, respectivamente. A resolução temporal variou de acordo com a relação sinal-ruído (SNR). A amostra foi posicionada o mais próximo possível do cintilador para reduzir a distorção geométrica, como borramento. Estágios de translação e rotação estão disponíveis para fixar a amostra próxima aos detectores e realizar tomografia computadorizada (TC). CG-1D oferece três tipos de detectores: um dispositivo acoplado à carga (CCD) com 2048 pixels x 2048 pixels com um passo de pixel de 13,5 μm, um detector científico complementar de semicondutor de óxido de metal (sCMOS) com 2560 pixels x 2160 pixels com um passo de pixel de 6,5μm, e um detector de placa de microcanal (MCP)30,31 com 512 pixels x 512 pixels com um tamanho de pixel de 55μm. Nêutrons espalhados são absorvidos com borracha de boro de ~5 mm de espessura para proteger o chip do detector de ver nêutrons. Essa absorção gera raios gama que podem ser interrompidos pelo chumbo (Pb) colocado entre a borracha de boro e o detector. Cada detector é otimizado para um campo de visão diferente (FOV), bem como resoluções espaciais e temporais. Para as medidas de fêmur de rato e pulmão de camundongo, o detector CCD foi utilizado por sua grande capacidade de FOV (~ 7 cm x 7 cm) e resolução espacial razoável de aproximadamente 75μm. O nCT da raiz/sistema solo da planta foi realizado com o sCMOS, pois o objetivo era adquirir nCTs o mais rápido possível ao custo de FOV (que foi limitado a ~ 5 cm x 4,2 cm); assim, a resolução espacial evidentemente sofreu. Nesses detectores, os nêutrons são convertidos em luz ou em uma partícula alfa para fins de detecção. A rotação da amostra em torno de seu eixo vertical e a aquisição de radiografias em ângulos de rotação consecutivos permitem a aquisição de nCT. O modelo renderizado volumétrico tridimensional da amostra sob investigação é obtido utilizando-se o notebook interno Jupyter baseado em píton iMARS3D baseado em píton (FBP), pyMBIR ou um software comercial, todos descritos abaixo.
Finalmente, os nêutrons que não interagiram com a amostra ou com o detector são coletados em uma posição de parada do feixe aproximadamente 1 m a jusante do sistema detector para minimizar o ruído de fundo. O batente do feixe CG-1D tem 0,75 m de largura, 0,5 m de altura e 35 mm de espessura e é feito de B4C em epóxi. O batente do feixe é reforçado com 10 mm de carbonato de lítio enriquecido a 95% (6 Li2CO3) em um epóxi resistente ao fogo onde o feixe de nêutrons atinge, com uma cavidade revestida com 6Li, chumbo (Pb) e aço projetado para conter a alta taxa de raios gama secundários. O batente da viga é diretamente ligado à parede de blindagem de aço da linha de luz. Uma fotografia da linha de luz CG-1D é apresentada na Figura 2.
Três softwares de reconstrução foram utilizados para reconstruir os três dados experimentais em 3D, respectivamente. A reconstrução da amostra de pulmão de camundongo foi realizada com o Octopus32, um software de reconstrução comercial que utiliza FBP. O software Octopus fica em um PC servidor e pode ser usado para reconstruir dados coletados na linha de luz. Um software de reconstrução, chamado iMARS3D, está disponível na CG-1D. Ele é baseado no código fonte aberto TomoPY33 com recursos adicionais, como correção de inclinação automatizada, filtros de pós-processamento, etc. O iMARS3D inclui pré-processamento dos dados (subtração do fundo e ruído), corte, filtragem mediana (para corrigir golpes gama e pixels mortos), correção automatizada da flutuação da intensidade do feixe e correção da inclinação da amostra. Uma vez que os sinogramas são criados, o processamento de dados adicionais, como a remoção de artefatos de anel e suavização são uma opção. As diferentes etapas da reconstrução são salvas no servidor de análise (e posteriormente movidas na pasta compartilhada da proposta), enquanto as fatias 2D finais são imediatamente armazenadas na pasta compartilhada da proposta. O fêmur de ratos foi reconstruído com iMARS3D. A amostra de raiz/solo da planta foi pré-processada por mediana filtrando os dados usando TomoPY seguido de correção do eixo de inclinação usando a biblioteca SciPy de Python. A reconstrução foi realizada usando um pacote python desenvolvido internamente denominado pyMBIR (construído usando kernels da caixa de ferramentas ASTRA34) que implementa um conjunto de algoritmos tomográficos desde a linha de base FBP até técnicas avançadas de reconstrução iterativa baseadas em modelos35 que podem obter reconstruções de alta qualidade a partir de conjuntos de dados de nêutrons extremamente esparsos e barulhentos. Todos os volumes renderizados com base nas ferramentas de reconstrução acima mencionadas são representados em contraste de atenuação. Toda a visualização foi realizada utilizando-se o software de visualização comercial, segmentação e análise de dados AMIRA36.
Este manuscrito tem como objetivo demonstrar o procedimento de utilização de imagens de nêutrons (nR e nCT) na linha de luz HFIR CG-1D. Este estudo também ilustra as capacidades atuais de nR e nCT de última geração para amostras biológicas, especificamente um pulmão de camundongo, um osso de rato e sistemas de raiz/solo de plantas. O pulmão de camundongo foi escolhido para ilustrar a complementaridade de nêutrons para medir o tecido pulmonar, enquanto os raios X são mais sensíveis aos ossos. A amostra óssea, um fêmur de rato, tinha um implante de titânio (Ti), ilustrando o contraste entre o osso e o metal, e a oportunidade de ver a interface osso/metal (que é difícil de medir com radiografias, pois os metais os atenuam fortemente4). Finalmente, o sistema de água planta-raiz ilustra a capacidade tridimensional (3D) do nCT para medir sistemas raiz/solo in situ. Além disso, mostra as vantagens/desvantagens do uso de nR para amostras biológicas. Evidentemente, este método pode ser usado com segurança para medir a dinâmica da água em um sistema planta-raiz, mas não pode ser considerado como uma técnica de imagem animal vivo ou humano devido aos riscos associados à exposição à radiação, limitando os estudos a camundongos (mortos) ou medições semelhantes a patologias em que, por exemplo, uma amostra de tecido é ressecada de um paciente (animal ou humano) e preparada por fixação antes de ser medida em um feixe de nêutrons.
Protocol
Representative Results
Discussion
A radiografia de nêutrons e a TC de amostras biológicas são técnicas de imagem promissoras e complementares às radiografias ou à ressonância magnética. As etapas críticas na realização de um experimento de imagem de nêutrons de uma amostra biológica estão relacionadas à sua preparação e sua contenção na linha de luz. A otimização de um experimento é impulsionada pela pergunta científica a ser respondida. Se a questão científica requer alta resolução espacial para observar um fenômeno, então longos tempos de aquisição são necessários, e a desvantagem da nCT (com campo de visão de tamanho cm) é que leva horas para realizar uma varredura. Isso se deve principalmente à diferença no fluxo geral de nêutrons disponível em um reator em comparação com uma fonte síncrotron, onde as tomografias computadorizadas de raios X podem levar de segundos a minutos para alguns mm2 de campo de visão. Embora o método possa ser aplicado a amostras de tecido ex vivo extraídas de animais, não pode ser estendido in vivo a animais vivos ou humanos devido ao risco de exposição à radiação (como raios gama produzidos por nêutrons e interações de nêutrons com os átomos da amostra). No entanto, é adequado para a obtenção de imagens das interações raiz/solo da planta (Figura 7), como a dinâmica da absorção de água.
A vantagem do uso de nCT rápido para a dinâmica da planta é a sensibilidade ao H na água e a ausência de danos de radiação à planta, ao contrário da TC de raios X. Além disso, um contraste único pode ser observado a partir do uso de nêutrons em amostras de osso/metal, como um fêmur de rato, onde o metal é relativamente transparente em comparação com os tecidos circundantes (Figura 5), potencialmente evitando artefatos metálicos induzidos pela TC de raios-X39. Tecidos animais, como o pulmão de camundongos (Figura 6), mostram impressionante detecção de estrutura de tecidos moles porque os nêutrons são sensíveis ao H, mas a resolução espacial é um pouco o fator limitante nessas medidas. O contraste é fornecido pelos átomos H presentes nas amostras biológicas19,39.
Com o avanço de novas técnicas, como a interferometria de grade de nêutrons, e a melhoria na resolução espacial (alguns mícrons foram recentemente relatados42,43), a imagem por nêutrons pode oferecer ainda novos mecanismos de contraste para tecidos biológicos com melhor resolução espacial. A exploração de nêutrons de maior energia (para permitir a medição de amostras espessas) também promete a capacidade de medir seções maiores de um tecido animal, como um camundongo intacto, oferecendo assim novas possibilidades para a pesquisa biomédica.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Parte desta pesquisa utilizou recursos do Reator de Isótopos de Alto Fluxo, operado pela ORNL, e patrocinado pelo Departamento de Energia dos EUA, Office of Science, User Facilities, sob contrato DE-AC05-00OR22725 com a UT-Battelle, LLC. Parte desta pesquisa foi apoiada pelo ORNL através do programa Eugene Wigner Distinguished Staff Fellowship. Esta pesquisa também foi patrocinada pelo DOE Office of Science, Office of Biological and Environmental Research. Amostras femorais de ratos foram obtidas de experimentos realizados em colaboração com o Dr. Rick Sumner no Rush University Medical Center com financiamento obtido do NIH (R01AR066562) e do prêmio Orthopedic Research and Education Foundation-Smith and Nephew. A equipe quer agradecer às equipes de suporte HFIR que permitem o uso das linhas de luz de espalhamento de nêutrons.
Materials
| Aluminum containers | custom | Made from aluminum plates or tubing (alternate is quartz), plant and mouse sample | |
| Aluminum foil | Fisher | 01-213-100 | Mouse lung sample containment |
| Deionized water or deuterium oxide | Water or D2O can be used to enhance contrast, plant sample | ||
| Ethanol | Fisher | 04-355-223 | Mouse lung sample |
| Gauze sponges | CardinalHealth | Fully submerged in phosphate-buffered saline (PBS) and used to wrap samples, rat femur sample | |
| Growth chamber | Conviron | A1000 | Any growth chamber or greenhouse with controlled conditions would work, plant sample |
| Laboratory balance | Weighing plant system can be used to measure actual water content in the soils, plant sample | ||
| Pure silica sand | US Silica Co. | Flint#13 | Pure SiO2 provides low neutron attenuation compared to soils, plant sample |
| Sprague-Dawley Rats | Harlan | Order Code: 002-US | Rat femur sample |
| Titanium Rod | Goodfellow | TI007905 | Rat femur sample |
References
- Cekanova, M., Donnell, R., Bilheux, H., Bilheux, J. -. C. Neutron imaging: Detection of cancer using animal model. Proceedings of the 2014 Biomedical Sciences and Engineering Conference – 5th Annual ORNL Biomedical Sciences and Engineering Conference: Collaborative Biomedical Innovations – The Multi-Scale Brain: Spanning Molecular, Cellular, Systems, Cognitive, Behaviour. , (2014).
- Bilheux, H. Z., et al. Neutron imaging a. The Oak Ridge National Laboratory: Application to biological research. Proceedings of the 2014 Biomedical Sciences and Engineering Conference – 5th Annual ORNL Biomedical Sciences and Engineering Conference: Collaborative Biomedical Innovations – The Multi-Scale Brain: Spanning Molecular, Cellular, Systems, Cognitive, Behaviour. , (2014).
- Bilheux, H. Z., et al. A novel approach to determine post mortem interval using neutron radiography. Forensic Science International. 251, (2015).
- Isaksson, H., et al. Neutron tomographic imaging of bone-implant interface: Comparison with X-ray tomography. Bone. 103, 295-301 (2017).
- Le Cann, S., et al. Characterization of the bone-metal implant interface by Digital Volume Correlation of in-situ loading using neutron tomography. Journal of the Mechanical Behavior of Biomedical Materials. 75, 271-278 (2017).
- Burca, G., et al. Exploring the potential of neutron imaging for life sciences on IMAT. Journal of Microscopy. 272 (3), 242-247 (2018).
- Metzke, R. W., et al. Neutron computed tomography of rat lungs. Physics in Medicine and Biology. 56 (1), 1-10 (2011).
- Altieri, S., et al. Neutron autoradiography imaging of selective boron uptake in human metastatic tumours. Applied Radiation and Isotopes. 66 (12), 1850-1855 (2008).
- Altieri, S., Bortolussi, S., Bruschi, P., Pedroni, P., Zonta, A. Neutron radiography of human liver metastases after BPA infusion. Proceedings of 11th World congress on Neutron Capture Therapy. , (2004).
- Holz, M., Zarebanadkouki, M., Kaestner, A., Kuzyakov, Y., Carminati, A. Rhizodeposition under drought is controlled by root growth rate and rhizosphere water content. Plant and Soil. 423 (1-2), 429-442 (2018).
- Esser, H. G., Carminati, A., Vontobel, P., Lehmann, E. H., Oswald, S. E. Neutron radiography and tomography of water distribution in the root zone. Journal of Plant Nutrition and Soil Science. 173 (5), 757-764 (2010).
- Warren, J. M., et al. Neutron imaging reveals internal plant water dynamics. Plant and Soil. 366 (1-2), (2013).
- Dhiman, I., et al. Quantifying root water extraction after drought recovery using sub-mm in situ empirical data. Plant and Soil. 424, 73-89 (2018).
- Kroener, E., Zarebanadkouki, M., Kaestner, A., Carminati, A. Non-equilibrium dynamics of rhizosphere. Water Resources Research. 50 (8), 6479-6495 (2014).
- Moradi, A. B., et al. Three-dimensional visualization and quantification of water content in the rhizosphere. New Phytologist. 192 (3), 653-663 (2011).
- Banhart, J., et al. X-ray and neutron imaging – Complementary techniques for materials science and engineering. International Journal of Materials Research. 101 (9), 1069-1079 (2010).
- LaManna, J. M., Hussey, D. S., Baltic, E. M., Jacobson, D. L. Improving material identification by combining x-ray and neutron tomography. Proceedings 10391, Developments in X-Ray Tomography XI. 1039104, (2017).
- Tengattini, A., et al. NeXT-Gremoble, the Neutron and X-ray tomograph in Grenoble. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 968, 163939 (2020).
- Bilheux, H. Z., McGreevy, R. L., Anderson, I. S. . Neutron Imaging and Applications. , (2009).
- Zhang, P., Wittmann, F. H., Zhao, T. J., Lehmann, E. H., Vontobel, P. Neutron radiography, a powerful method to determine time-dependent moisture distributions in concrete. Nuclear Engineering and Design. 241 (12), 4758-4766 (2011).
- Lobo, R. M., Andrade, A. H. P., Castagnet, M. Hydride embrittlement in zircaloy components. Inac 2011 Int Nucl.Atlantic Conference. , 1-6 (2011).
- Kardjilov, N., et al. New trends in neutron imaging. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 605 (1-2), 13-15 (2009).
- Schillinger, B., et al. Detection systems for short-time stroboscopic neutron imaging and measurements on a rotating engine. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 542 (1-3), 142-147 (2005).
- Thewlis, J. Neutron Radiography. British Journal of Applied Physics. 7, 345-350 (1956).
- Anderson, J. Neutron Radiography in Man. British Journal of Radiology. 37, 957-958 (1964).
- Brown, M., Parks, P. B. Neutron radiography in biologic media: techniques, observations, and implications. American Journal of Roentgenology. 106 (3), 472-485 (1969).
- Metzke, R. W., Runck, H., Stahl, C. A., Schillinger, B., Calzada, E. Neutron computed tomography of rat lungs. Physics in Medicine and Biology. 56 (1), 1-10 (2011).
- Crow, L., et al. The CG1 instrument development test station at the high flux isotope reactor. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 634 (1), 71-74 (2011).
- Santodonato, L., et al. The CG-1D neutron imaging beamline at the Oak Ridge National Laboratory High Flux Isotope Reactor. Physics Procedia. 69, 104-108 (2015).
- Tremsin, A. S., et al. Improved efficiency of high resolution thermal and cold neutron imaging. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 628 (1), 415-418 (2011).
- Tremsin, A. S., Vallerga, J. V., McPhate, J. B., Siegmund, O. H. W. Optimization of high count rate event counting detector with microchannel plates and quad Timepix readout. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 787, 20-25 (2015).
- Vlassenbroeck, J., et al. Software tools for quantification of X-ray microtomography at the UGCT. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 580 (1), 442-445 (2007).
- Gürsoy, D., De Carlo, F., Xiao, X., Jacobsen, C. TomoPy: A framework for the analysis of synchrotron tomographic data. Journal of Synchrotron Radiation. 21 (5), 1188-1193 (2014).
- Pelt, D. M., et al. Integration of TomoPy and the ASTRA toolbox for advanced processing and reconstruction of tomographic synchrotron data. Journal of Synchrotron Radiation. 23 (3), 842-849 (2016).
- Venkatakrishnan, S. V., Cakmak, E., Billheux, H., Bingham, P., Archibald, R. K. Model-based iterative reconstruction for neutron laminography. Conference Record of 51st Asilomar Conference on Signals, Systems and Computers, ACSSC 2017. , (2018).
- Stalling, D., Westerhoff, M., Hege, H. C. Amira: A highly interactive system for visual data analysis. Visualization Handbook. 1, 749-767 (2005).
- Grünzweig, C., Frei, G., Lehmann, E., Kühne, G., David, C. Highly absorbing gadolinium test device to characterize the performance of neutron imaging detector systems. Review of Scientific Instruments. 78 (5), 1-4 (2007).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Meagher, M. J., Parwani, R. N., Virdi, A. S., Sumner, D. R. Optimizing a micro-computed tomography-based surrogate measurement of bone-implant contact. Journal of Orthopaedic Research. 36 (3), 979-986 (2018).
- Dalesio, L. R., et al. The experimental physics and industrial control system architecture: past, present, and future. Nuclear Inst. and Methods in Physics Research, A. 352 (1-2), 179-184 (1994).
- Bilheux, J., Lin, J. Y. Y., Bilheux, H. Z. Jupyter notebooks for neutron radiography data processing and analysis. Neutron Radiography-WCNR-11. 15, 198-204 (2020).
- Trtik, P., et al. Improving the spatial resolution of neutron imaging at Paul Scherrer Institut – The Neutron Microscope Project. Physics Procedia. 69, 169-176 (2015).
- Morgano, M., et al. Unlocking high spatial resolution in neutron imaging through an add-on fibre optics taper. Optics Express. 26 (2), 9-12 (2018).

