Détermination rapide de l’affinité anticorps-antigène par photométrie de masse
Summary
Nous décrivons une approche à molécule unique aux mesures d’affinité d’antigène-anticorps utilisant la photométrie de masse (MP). Le protocole basé sur mp est rapide, précis, utilise une très petite quantité de matériel, et ne nécessite pas de modification des protéines.
Abstract
Les mesures de la spécificité et de l’affinité des interactions antigène-anticorps sont d’une importance cruciale pour les applications médicales et de recherche. Dans ce protocole, nous décrivons la mise en œuvre d’une nouvelle technique à molécule unique, la photométrie de masse (MP), à cette fin. MP est une technique sans étiquette et sans immobilisation qui détecte et quantifie les masses moléculaires et les populations d’anticorps et de complexes anticorps à un niveau d’une seule molécule. MP analyse l’échantillon d’antigène-anticorps en quelques minutes, permettant la détermination précise de l’affinité contraignante et fournissant simultanément des informations sur la stoichiométrie et l’état oligomérique des protéines. Il s’agit d’une technique simple et simple qui ne nécessite que des quantités picomoles de protéines et pas de consommables coûteux. La même procédure peut être utilisée pour étudier la liaison protéines-protéines pour les protéines dont la masse moléculaire est supérieure à 50 kDa. Pour les interactions protéiques multivalentes, les affinités de plusieurs sites de liaison peuvent être obtenues en une seule mesure. Cependant, le mode de mesure à molécule unique et l’absence d’étiquetage imposent certaines limites expérimentales. Cette méthode donne les meilleurs résultats lorsqu’elle est appliquée à des mesures d’affinités d’interaction sous-micromolaire, d’antigènes d’une masse moléculaire de 20 kDa ou plus, et d’échantillons de protéines relativement purs. Nous décrivons également la procédure d’exécution des étapes de montage et de calcul requises à l’aide d’un logiciel d’analyse de données de base.
Introduction
Les anticorps sont devenus des outils omniprésents de la biologie moléculaire et sont largement utilisés dans les applications médicales et de recherche. En médecine, ils sont d’une importance cruciale dans le diagnostic, mais leurs applications thérapeutiques sont également en expansion et de nouvelles thérapies à base d’anticorps sont constamment développés1,2,3,4. Les applications scientifiques des anticorps incluent beaucoup de techniques indispensables de laboratoire telles que l’immunofluorescence5,l’immunoprécipice6,la cytométriede flux 7,ELISA, et le blotting occidental. Pour chacune de ces applications, l’obtention de mesures précises des propriétés contraignantes de l’anticorps, y compris l’affinité et la spécificité contraignantes, est d’une importance cruciale.
Depuis l’introduction du premier instrument commercial de résonance plasmon de surface (SPR) en 1990, les biocapteurs optiques sont devenus l’étalon-or de la caractérisation des anticorps, mais d’autres techniques, y compris l’ELISA, sont également couramment utilisées pour mesurer les affinités d’anticorps8,9. Ces méthodes nécessitent généralement l’immobilisation ou l’étiquetage des molécules analysées, ce qui peut potentiellement affecter l’interaction d’intérêt. Ils sont également relativement lents, impliquant plusieurs étapes d’analyse avant que les résultats puissent être recueillis pour l’analyse des données. Une méthode à molécule unique récemment mise au point, la photométrie de masse (MP), détecte les molécules directement en solution lorsqu’elles atterrissent à la surface du microscope coverslip10,11. La détection optique à base de diffusion de lumière que MP utilise ne nécessite pas d’étiquetage ou de modification des protéines. Les molécules protéiques individuelles sont enregistrées par le microscope de diffusion interférométrique comme des taches sombres apparaissant dans l’image (Figure 1D), et plusieurs milliers de molécules peuvent être détectées lors de l’acquisition de données d’une minute12. Le signal généré par chaque particule individuelle est quantifié, et sa valeur de contraste (obscurité relative) est calculée. Les valeurs de contraste interférométriques sont proportionnelles aux masses moléculaires des protéines, ce qui permet l’identification d’espèces liées et libres dans le mélange antigène-anticorps. Dans le même temps, en comptant les événements d’atterrissage moléculaire, MP mesure directement les populations d’espèces. Cela donne aux méthodes basées sur mp une capacité unique de quantifier indépendamment les affinités de plusieurs sites de liaison.
La liaison des molécules del’antigène (Ag)aux deux sites de liaison de l’anticorps intact (Ab) peut être décrite comme :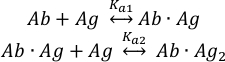
avec l’association d’équilibre constantes Ka1 et Ka2 définies comme :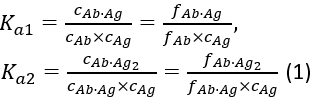
où ci et fi représentent la concentration et la fraction du composant i, respectivement. La concentration totale d’antigène( cAg)tot peut être exprimée comme: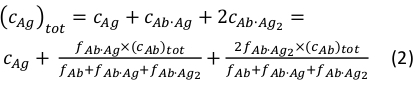
Étant donné que les concentrations totales de l’anticorps (cAb)tot etantigène( cAg)tot sont connus, cette équation peut être utilisée pour s’adapter directement aux fractions de composants expérimentaux obtenues à partir des mesures MP et calculer les constantes de l’association d’équilibre Ka1 et Ka2 (voir Information supplémentaire).
Les données MP peuvent également être utilisées pour estimer la cooperativité entre les deux sites de liaison d’anticorps11. Pour deux paratopes d’anticorps avec des constantes microscopiques identiques de liaison, les facteurs statistiques décrivant le processus de population de l’Ab· Ag et Ab· Ag2 complexes dictent que l’équilibre macroscopique apparent constantes Ka1 et Ka2 ne sera pas numériquement égal, et Ka1 = 4Ka2. Par conséquent, les valeurs expérimentales de Ka1 < 4Ka2 indiquent une cooperativité positive entre les deux sites de liaison d’anticorps. De même, Ka1 > 4Ka2 indique la cooperativité négative.
Les mesures mp de l’affinité de liaison antigène-anticorps sont rapides et nécessitent une petite quantité de matériel. Les distributions de masse MP utilisées pour les calculs constants d’équilibre fournissent des informations supplémentaires sur les propriétés de l’échantillon et permettent d’évaluer la pureté de l’échantillon, l’oligomérisation et l’agrégation en une seule expérience. La même méthode peut être utilisée pour mesurer la liaison protéine-protéine à haute affinité, et mp est particulièrement utile pour les études sur les interactions protéiques multi-valent. Les complexes multi-protéines ont généralement de grandes masses moléculaires, optimales pour la détection mp, et les données d’une molécule peuvent être utilisées pour mesurer la stoichiométrie et calculer les affinités de plusieurs sites de liaison simultanément. Ces informations sont généralement difficiles à obtenir à l’aide de méthodes en vrac.
Sans modifications, le protocole actuel convient aux mesures d’interactions sous-micromolaires relativement élevées avec des antigènes d’une masse moléculaire de 20 kDa ou plus. Pour des résultats optimaux, les stocks de protéines doivent être d’une grande pureté, mais il n’y a pas d’exigences spécifiques en matière de tampon. En utilisant MP, la liaison antigène-anticorps peut être évaluée en moins de cinq minutes. La collecte et l’analyse des données requises pour des calculs Kd précis peuvent être effectuées dans les 30 minutes.
Protocol
Representative Results
Discussion
Le protocole basé sur la photométrie de masse décrit ici fournit une méthode rapide et précise de mesure des affinités de liaison antigène-anticorps. L’analyse mp utilise une très petite quantité de matériel, et des informations supplémentaires , y compris la stoichiométrie, l’oligomérisation et la pureté , peuvent être évaluées à partir des mêmes données (figure 5). Sans modifications, cette méthode s’applique aux mesures des constantes de dissociation d’environ…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous remercions Keir Neuman pour sa lecture critique du manuscrit. Ce travail a été appuyé par le programme intra-muros de l’ICSLNH, nih.
Materials
| AcquireMP | Refeyn | MP data collection software | |
| Anti-human thrombin | Haematologic Technologies | AHT-5020 | RRID: AB_2864302 |
| Cotton-tipped applicators | Thorlabs | CTA10 | cotton optical swabs for lens cleaning |
| Coverslips 24×24 mm | Globe Scientific | 1405-10 | |
| Coverslips 24×50 mm | Fisher Scientific | 12-544-EP | |
| DiscoverMP | Refeyn | MP data processing software | |
| Forceps | Electron Microscopy Sciences | 78080-CF | soft-tipped forceps for coverslips handling |
| Human α-thrombin | Haematologic Technologies | HCT-0020 | |
| Immersion oil | Thorlabs | MOIL-30 | |
| Isopropanol | Alfa Aesar | 36644 | |
| Microsoft Excel | Microsoft | spreadsheet | |
| OneMP | Refeyn | Mass Photometry instrument | |
| Origin | OriginLab | scientific graphing software | |
| PBS | Corning | 46-013-CM | 10x stock |
| Syringe filter | Millipore | SLGSR33SS | buffer and sample filtering |
References
- Francis, R. J., et al. A phase I trial of antibody directed enzyme prodrug therapy (ADEPT) in patients with advanced colorectal carcinoma or other CEA producing tumours. British Journal of Cancer. 87 (6), 600-607 (2002).
- van Dyck, C. H. Anti-Amyloid-beta Monoclonal Antibodies for Alzheimer’s Disease: Pitfalls and Promise. Biological Psychiatry. 83 (4), 311-319 (2018).
- Vennepureddy, A., Singh, P., Rastogi, R., Atallah, J. P., Terjanian, T. Evolution of ramucirumab in the treatment of cancer – A review of literature. Journal of Oncology Pharmacy Practice. 23 (7), 525-539 (2017).
- Waldmann, T. A. Immunotherapy: past, present and future. Nature Medicine. 9 (3), 269-277 (2003).
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annual Review of Biochemistry. 78, 993-1016 (2009).
- Rosenberg, M. I. . Protein Analysis and Purification. , (2005).
- Picot, J., Guerin, C. L., Le Van Kim, C., Boulanger, C. M. Flow cytometry: Retrospective, fundamentals and recent instrumentation. Cytotechnology. 64 (2), 109-130 (2012).
- Khan, S. H., Farkas, K., Kumar, R., Ling, J. A versatile method to measure the binding to basic proteins by surface plasmon resonance. Analytical Biochemistry. 421 (2), 385-390 (2012).
- Lofgren, J. A., et al. Comparing ELISA and surface plasmon resonance for assessing clinical immunogenicity of panitumumab. The Journal of Immunology. 178 (11), 7467-7472 (2007).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Wu, D., Piszczek, G. Measuring the affinity of protein-protein interactions on a single-molecule level by mass photometry. Analytical Biochemistry. 592, 113575 (2020).
- Cole, D., Young, G., Weigel, A., Sebesta, A., Kukura, P. Label-free single-molecule imaging with numerical-aperture-shaped interferometric scattering microscopy. ACS Photonics. 4 (2), 211-216 (2017).
- Soltermann, F., et al. Quantifying protein-protein interactions by molecular counting with mass photometry. Angewandte Chemie International Edition in English. 59 (27), 10774-10779 (2020).
- Kemmer, G., Keller, S. Nonlinear least-squares data fitting in Excel spreadsheets. Nature Protocols. 5 (2), 267-281 (2010).

