Studie av proteindynamikk via nøytron spin ekko spektroskopi
Summary
Denne protokollen beskriver metoder for å undersøke strukturen og dynamikken til to modellproteiner som har en viktig rolle i menneskers helse. Teknikken kombinerer benk-topp biofysisk karakterisering med nøytronspinn ekkospektroskopi for å få tilgang til dynamikken på tids- og lengdeskalaer som er relevante for proteininterdomainbevegelser.
Abstract
De fleste menneskekroppsproteiners aktivitet og funksjonalitet er relatert til konfigurasjonsendringer av hele underdomener i proteinkrystallstrukturen. Krystallstrukturene bygger grunnlaget for enhver beregning som beskriver strukturen eller dynamikken til et protein, mesteparten av tiden med sterke geometriske begrensninger. Disse begrensningene fra krystallstrukturen er imidlertid ikke til stede i løsningen. Strukturen av proteinene i løsningen kan avvike fra krystallet på grunn av omorganiseringer av løkker eller underdomener på pico til nanosekund tidsskala (dvs. det interne proteindynamikktidsregimet). Det nåværende arbeidet beskriver hvordan sakte bevegelser på tidsskalaer på flere titalls nanosekunder kan nås ved hjelp av nøytronspredning. Spesielt er den dynamiske karakteriseringen av to store humane proteiner, et iboende uordnet protein som mangler en veldefinert sekundær struktur og et klassisk antistoffprotein, adressert ved nøytronspinnekkospektroskopi (NSE) kombinert med et bredt spekter av laboratoriekarakteriseringsmetoder. Ytterligere innsikt i proteindomenedynamikk ble oppnådd ved hjelp av matematisk modellering for å beskrive eksperimentelle nøytrondata og bestemme krysningen mellom kombinert diffusive og interne proteinbevegelser. Ekstraksjonen av det interne dynamiske bidraget til den mellomliggende spredningsfunksjonen oppnådd fra NSE, inkludert tidsskalaen for de forskjellige bevegelsene, tillater ytterligere syn på de mekaniske egenskapene til enkeltproteiner og mykheten til proteiner i deres nesten naturlige miljø i den overfylte proteinløsningen.
Introduction
Sonderingsdynamikk av myk materie med nøytroner
Å undersøke de dynamiske egenskapene til proteiner og peptider er en stor del av biofysisk forskning, og det finnes i dag mange velutviklede metoder for å få tilgang til et bredt spekter av energilandskap. Å relatere den eksperimentelt avslørte dynamikken til proteinene til deres biologiske funksjon er en langt vanskeligere oppgave, som krever komplekse matematiske modeller og datastøttede dynamikksimuleringer. Betydningen av nøytronspektroskopi for analyse av proteinbevegelser har blitt understreket i flere godt mottatte og anerkjente studier 1,2,3,4,5. Før du utforsker det mangfoldige energilandskapet for intern proteindynamikk, er det nødvendig med en kort oversikt over de dynamiske prosessene i myk materie og hvordan nøytroner kan få tilgang til dem.
Nøytronenes følsomhet overfor isotopisk konfigurasjon og typen interaksjoner de viser med myk materie, gjør nøytronspredning til en av de mest allsidige undersøkelsesteknikkene6. Det er et bredt spekter av korrelasjonslengdeskalaer og korrelasjonstider som nøytroner kan få tilgang til, fra kjernefysiske eksitasjoner og atomvibrasjoner til kollektive bevegelser og langsomme avslapningsprosesser som isotropiske rotasjoner og diffusive bevegelser. Når man undersøker de spredte nøytronene for deres energioverføring, kan tre hovedinteraksjoner skilles: den elastiske spredningen, der det ikke er energiutveksling mellom innkommende nøytron og partikkel i prøven; den uelastiske spredningen, med en stor, kvantifiserbar energiutveksling mellom nøytron og partikkel; og det særegne tilfellet av kvasi-elastisk spredning som betegner en svært liten energioverføring sammenlignet med den innfallende nøytronenergien 1,7. Disse interaksjonene gir presis informasjon om det undersøkte materialet og danner det teoretiske grunnlaget for et bredt spekter av nøytronspredningsteknikker.
I elastisk spredning registrerer detektoren retningene til nøytronene som et diffraksjonsmønster, som viser posisjonen til prøveatomene i forhold til hverandre. Informasjon om korrelasjonene mellom atomposisjoner er anskaffet (dvs. integrert intensitet S (Q) angående momentumoverføringen Q, som gjelder strukturell informasjon alene). Dette prinsippet danner grunnlaget for nøytrondiffraksjon8.
Kompleksitet oppstår når energioverføringen ikke lenger er null på grunn av eksitasjoner og interne svingninger i prøvematerialet. Dette danner grunnlaget for nøytronspektroskopi, der de spredte nøytronene undersøkes som en funksjon av både energioverføringen E og momentumoverføringen Q. Dynamisk og strukturell informasjon oppnås. Nøytronspektroskopi måler den samme integrerte intensiteten S (Q) for energioverføring (dvs. hastighetsendring av nøytronene på grunn av prøvespredning, S (Q, ω) = S (Q, E), som også refereres til som den dynamiske strukturfaktoren) 9.
For å beregne spredningen fra et materiale er det mer tilstrekkelig å bruke parkorrelasjonsfunksjonen 7,10. I diffraksjonstilfellet gir den statiske parkorrelasjonsfunksjonen G (r) sannsynligheten for å finne sentrum av en partikkel i en gitt avstand r fra sentrum av en annen partikkel. Spektroskopien generaliserer den statiske parkorrelasjonsfunksjonen og inkluderer energi/ frekvens / tid i spredningsligningen. Parkorrelasjonsfunksjonen G(r) blir en funksjon av tid G(r, t), som kan dekomponeres i en distinkt atomparkorrelasjonsfunksjon GD(r, t), og en selvkorrelasjonsfunksjon GS(r, t). Disse beskriver to typer korrelasjoner: parkorrelerte bevegelser av atomer som styrer den sammenhengende spredningen, og selvkorrelasjon som styrer den usammenhengendespredningen 10.
Koherent spredning er spredningen fra “gjennomsnittet” og avhenger av den relative fasen av de spredte bølgene. I det små vinkelspredningsregimet forstyrrer de spredte nøytronbølgene fra forskjellige spredningssentre (forskjellige atomer) konstruktivt (har lignende faser), og atomenes kollektive bevegelse observeres med sterk intensitetsforbedring. Koherent spredning beskriver i hovedsak spredningen av et enkelt nøytron fra alle kjernene i prøven10.
Når det ikke oppstår noen konstruktiv interferens mellom de spredte nøytronbølgene fra forskjellige sentre, følges et enkelt atom i tid, og selvkorrelasjonen mellom atomets posisjon på tid t = 0 og det samme atomet på tidspunktet t observeres. Dermed går informasjonen om atomenes relative posisjoner tapt, og fokuset er bare på lokale svingninger. Spredning fra lokale svingninger styrer usammenhengende spredning. Usammenhengende spredning er isotropisk, bidrar til bakgrunnssignalet og forringer signal-til-støy10,11.
Ved å kombinere alt det ovennevnte skiller vi fire store nøytronspredningsprosesser10: (1) elastisk koherent (måler korrelasjonene til atomposisjoner), (2) uelastisk koherent (måler kollektive bevegelser av atomer), (3) elastisk usammenhengende (bidrar til bakgrunnen, reduserer spredningsintensiteten ved Debye-Waller-faktor (DWF) og måler elastisk usammenhengende strukturfaktor (EISF), som beskriver geometrien til diffusive bevegelser i begrenset geometri, og (4) uelastisk usammenhengende (måler enkeltatomdynamikk og selvkorrelasjon).
Dynamikkprosesser som nøytroner kan få tilgang til i biologi spenner fra demping av lavfrekvente atom- og molekylære vibrasjoner, samspillet mellom løsningsmiddelmolekyler og biooverflater og diffusjonsprosesser i hydreringslaget av makromolekyler og begrenset geometri, til kortdistanse translasjonelle, rotasjons- og tumblingdiffusjonive bevegelser, og proteindomener og allosteriske bevegelser1 . Det store mangfoldet av nøytronmetoder og instrumenter for måling av proteindynamikk er basert på hvordan akromatiseringen av hendelsen eller den utgående nøytronstrålen oppnås og hvordan energianalysen av de spredte nøytronene utføres. Fra trippelakse til flytid, backscattering og spinn-ekko-spektrometre kan man utforske dynamiske prosesser med karakteristiske tider mellom 1 x 10-14 s og 1 x 10-6 s (femtosekund til mikrosekunder)12.
Oak Ridge National Laboratory, med sine to anerkjente nøytronkilder, Spallation Neutron Source – SNS13 og High Isotope Flux Reactor – HFIR14, har en av de beste suitene med spektrometre for å undersøke dynamikk i biomaterialer. Noen av de mest veltalende eksemplene inkluderer bruken av det kalde nøytronhakkerspektrometeret (CNCS) ved SNS15 for å undersøke dynamisk forstyrrelse av hydreringsvann rundt grønt fluorescerende protein i løsning16 eller subpikosekund kollektive vibrasjoner av flere proteiner17. Et tilbakevendende problem med uelastiske nøytronspredningsundersøkelser er at noen biologiske prosesser er for sakte til å bli observert. Uten ekstreme oppsett som fører til et stort tap av nøytronintensitet, er flytidsspektrometre begrenset til 10 μeV energioppløsning, tilsvarende en maksimal tidsskala på ~ 200 ps10,11. Dette er ikke tilstrekkelig til å observere store bevegelser i proteiner. Derfor er det ofte behov for instrumenter med høyere energioppløsning som backscattering-spektrometrene. Å kombinere time-of-flight og backscattering-teknikkene har vist seg å være kraftig for å undersøke endringen i den interne dynamikken til Cytochrome P450cam (CYP101), et enzym som katalyserer hydroksylerings kamfer18.
Mikroskopisk diffusivitet målt ved backscattering-spektrometeret ved SNS-BASIS19 var overraskende godt definert og kunne skilles i diffusiviteten til vann (hydrering, cytoplasmatisk og bulklignende vann) og diffusiviteten til cellebestanddeler i planariske flatorm, det første levende dyret som ble studert ved nøytronspredning20 . Backscattering er en høyoppløselig spektroskopisk teknikk, men den er også begrenset til flere μeV = flere nanosekunder, mens den langsomme dynamikken i biomaterialer også manifesterer seg som overlevelsestiden for korrelasjon mellom atomposisjon eller spinnorientering (f.eks. Avslapningsprosesser, som regelmessig skjer i tidsområdet ti til hundrevis av nanosekunder).
Nøytron spinn ekko spektroskopi (NSE) er den eneste nøytronspredningsteknikken som når så høy oppløsning. I motsetning til andre nøytronteknikker krever NSE ikke akromatisering av strålen siden den bruker den kvantemekaniske fasen til nøytronene, som er deres magnetiske øyeblikk. Manipuleringen av magnetiske øyeblikk tillater bruk av en bred nøytronstrålebølgelengdefordeling, mens teknikken er følsom for svært små nøytronhastighetsendringer i størrelsesorden 1 x 10-4. NSE har med hell blitt brukt til å undersøke den langsomme dynamikken til proteiner i løsning for mange proteiner. Blant disse mange pionerstudiene anerkjenner vi studiet av segmentfleksibiliteten til grisimmunoglobulin21; de koblede domenebevegelsene i Taq-polymerase22; domenebevegelsene i tetrameren av gjæralkohol dehydrogenase23; endringen av konformasjon i fosfoglyseratkinase ved substratbinding3; aktivering av domenebevegelser og dynamisk forplantning av allosteriske signaler i Na+/H+ utvekslingsregulatorisk kofaktor 1 (NHERF1) protein 4,24,25; dynamikken i en kompakt tilstand av kvikksølvionreduktase26; og diffusjonen av hemoglobin i røde blodlegemer27. To nyere studier i proteindynamikk har eksponert fleksibiliteten til humant antistoff Immunoglobulin G (IgG) som en entropisk fjær28 og egenskapene til løsemiddelbidrag til dynamikken til indre uordnet myelin basisk protein (MBP)5.
Denne artikkelen forklarer de grunnleggende prinsippene for NSE, de mange forberedende metodene som anbefales for en grundig proteindynamikkundersøkelse, samt metodikken og den eksperimentelle protokollen for NSE-datainnsamling ved NSE-spektrometeret ved SNS, SNS-NSE. Protokollen karakteriserer to proteiner: IgG, et vanlig humant antistoffprotein, og det indre uordnede proteinet MBP. De biofysiske implikasjonene, forskningsrelevansen til eksemplene og begrensningene i teknikken diskuteres kort.
NSE-spektroskopi, metoden for langsomme dynamikkmålinger
NSE er en polarisert teknikk som bruker nøytrontid for å måle utveksling av energi (tap av polarisasjon) på grunn av den kvasi-elastiske interaksjonen mellom nøytroner og atomer i en prøve. Kjernen i NSE-spektroskopi ligger to grunnleggende prinsipper: (1) nøytronspinnets evne til å presesere i magnetfeltet med en frekvens proporsjonal med magnetisk styrke 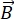 , nemlig Larmor-frekvensen29, og (b) spinn-ekkoet eller Hann-ekkoet, som representerer manipulering og refokusering av polarisasjonssignalet ved bruk av en serie radiofrekvenspulser30.
, nemlig Larmor-frekvensen29, og (b) spinn-ekkoet eller Hann-ekkoet, som representerer manipulering og refokusering av polarisasjonssignalet ved bruk av en serie radiofrekvenspulser30.
Det grunnleggende i NSE-prosessen kan oppsummeres i noen få enkle trinn 6,11 ved hjelp av figur 1. (1) Nøytronstrålen produsert av kilden (posisjon 1) polariseres (posisjon 2), styres og transporteres (posisjon 3), og kommer til inngangen til NSE-spektrometeret, hvor den roteres med 90 ° av den første pi-halvflipperen (posisjon 4). (2) Den polariserte strålen (f.eks. nøytronmagnetiske øyeblikk) blir vinkelrett på den første magnetens magnetfeltlinjer (første presesjonssone, posisjon 5) og begynner å presesere. (3) På slutten av magneten akkumulerer nøytronspinn en viss presesjonsvinkel proporsjonal med magnetfeltstyrken og flytiden som brukes inne (i utgangspunktet omvendt proporsjonal med nøytronhastigheten). De individuelle nøytronhastighetene er kodet innenfor presesjonsvinkelen på slutten av den første presesjonssonen. (4) Nær prøveposisjonen reverserer pi-flipperen (posisjon 6) retningen til spinnet med 180 °, og endrer tegnet på presesjonsvinkelen. (5) Nøytronene interagerer med prøvens molekyler (posisjon 7) og blir spredt. (6) De spredte nøytronene kommer inn og preseserer i den andre presesjonssonen (posisjon 8), men blir reversert orientert. (7) En annen pi-halv flipper (posisjon 9) brukes til å rotere orienteringen av spinnet fra vinkelrett til horisontal retning. Dette vil stoppe presesjonen, oversette presesjonsvinkelen φ til polarisasjon proporsjonal med cos (φ). (8) Analysatoren (posisjon 10) velger nøytronene basert på en orientering. Hvis interaksjonen med prøven er elastisk, vil nøytronets hastighet ikke endres. Nøytronene vil bruke like mye tid på å fly i første og andre presesjonssone, og de akkumulerte presesjonsvinklene blir fullstendig gjenvunnet. Den fulle polarisasjonen gjenopprettes på detektoren (posisjon 11) som et ekko av den opprinnelige polarisasjonen (dvs. spinn-ekko). (9) I NSE er imidlertid spredningen kvasi-elastisk, så en liten energiutveksling mellom nøytroner og prøvemolekyler fører til forskjellige nøytronhastigheter etter spredning av prøven. På grunn av de forskjellige hastighetene vil nøytronene bruke ekstra tid på å fly gjennom den andre presesjonssonen og vil ikke ha gjenopprettet presesjonsvinkelen på riktig måte. En partiell polarisasjon hentes på detektoren, og tapet av polarisasjon på grunn av spinnavslapping er proporsjonalt med cos-Fourier-transformasjonen av spektralfunksjonen S(Q, ω), den mellomliggende spredningsfunksjonen F(Q, t). (10) Tidsparameteren for funksjonen F(Q, t) er proporsjonal med presesjonens magnetfeltstyrke. Skanning av tap av polarisasjon som en funksjon av magnetfeltstyrke gir derfor en avslapningsfunksjon som avhenger av de dynamiske prosessene i prøven.

Figur 1: Fotografi av NSE-spektrometeret ved SNS (SNS-NSE) og nøytronfluebane skjematisk med de viktigste funksjonelle komponentene. Fra høyre til venstre: 1 = nøytronkilde; 2 = choppers-bender-polarizer-sekundær lukkersystem; 3 = stråletransport guider; 4 = pi/2 flipper for første 90° spinn-sving; 5 = første presesjonssone; 6 = pi flipper for 180 ° spin-turn; 7 = prøveområde og prøvemiljø (her vises kryoovnen); 8 = andre presesjonssone; 9 = pi/2 flipper for andre 90° spinn-sving; 10 = analysator; 11 = detektor. (Merk at deler av 3, samt 2 og 1, ligger bak den blå veggen inne i skjermingen; helikoptrene erstattes av en hastighetsvelger for reaktorbasert NSE). Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
NSE-spektroskopi gir en unik og detaljert oversikt over dynamikken til proteiner, som andre spektroskopiske teknikker ikke kan produsere. Målinger over en lengre tidsskala gir observasjoner av både proteinenes translasjons- og rotasjonsdiffusjon, som presentert her. Segmentdynamikken og andre interne svingninger avslører seg som et sterkt forfall av den sammenhengende spredningsfunksjonen S (Q, t) på kort tidsskala og er godt skilt fra de generelle diffusjonelle avslapningsprosessene. De viktigste begrensnin…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne forskningen brukte ressurser på Spallation Neutron Source (BL-15, BL-6, biologi og kjemi laboratorier), et DOE Office of the Science User Facility som drives av Oak Ridge National Laboratory. Denne forskningen brukte også ressurser ved MLZ-FRM2-reaktoren Garching (KWS-2, Phoenix-J-NSE) og JCNS1 ved Forschungszentrum Jülich GmbH, Tyskland. Forfatteren anerkjenner Dr. Ralf Biehl og Dr. Andreas Stadler for deres hjelp med modellering og deres bidrag til både IgG og MBP proteiner forskning, Dr. Piotr A. Żołnierczuk for NSE data reduksjon støtte, Dr. Changwoo Do for støtte med SANS målinger, og Rhonda Moody og Dr. Kevin Weiss for SNS biokjemi lab støtte.
Materials
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O – heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | – | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | – | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | – | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | – | small angle neutron instrument |
| Liquinox dish detergent | Alconox | – | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M – wax parafilm | Bemis | Parafilm M – 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | – | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | – | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | – | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | – | falling ball viscometer |
References
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).

