Untersuchung der Proteindynamik mittels Neutronen-Spin-Echo-Spektroskopie
Summary
Das vorliegende Protokoll beschreibt Methoden zur Untersuchung der Struktur und Dynamik von zwei Modellproteinen, die eine wichtige Rolle für die menschliche Gesundheit spielen. Die Technik kombiniert die biophysikalische Charakterisierung auf dem Labortisch mit der Neutronen-Spin-Echo-Spektroskopie, um auf die Dynamik auf Zeit- und Längenskalen zuzugreifen, die für Proteininterdomänenbewegungen relevant sind.
Abstract
Die Aktivität und Funktionalität der meisten menschlichen Körperproteine hängt mit Konfigurationsänderungen ganzer Subdomänen innerhalb der Proteinkristallstruktur zusammen. Die Kristallstrukturen bilden die Grundlage für jede Berechnung, die die Struktur oder Dynamik eines Proteins beschreibt, meist mit starken geometrischen Einschränkungen. Diese Einschränkungen aus der Kristallstruktur sind jedoch in der Lösung nicht vorhanden. Die Struktur der Proteine in der Lösung kann sich vom Kristall aufgrund von Umlagerungen von Schleifen oder Subdomänen auf der Pico-Nanosekunden-Zeitskala (d. h. dem internen Proteindynamik-Zeitregime) unterscheiden. Die vorliegende Arbeit beschreibt, wie Zeitlupe auf Zeitskalen von mehreren zehn Nanosekunden mittels Neutronenstreuung erreicht werden kann. Insbesondere die dynamische Charakterisierung von zwei großen menschlichen Proteinen, einem intrinsisch ungeordneten Protein, dem eine klar definierte Sekundärstruktur fehlt, und einem klassischen Antikörperprotein, wird durch Neutronen-Spin-Echo-Spektroskopie (NSE) in Kombination mit einer breiten Palette von Laborcharakterisierungsmethoden behandelt. Weitere Einblicke in die Dynamik der Proteindomäne wurden mithilfe mathematischer Modellierung erzielt, um die experimentellen Neutronendaten zu beschreiben und die Überschneidung zwischen kombinierten diffusiven und internen Proteinbewegungen zu bestimmen. Die Extraktion des internen dynamischen Beitrags zur aus NSE erhaltenen Zwischenstreufunktion, einschließlich der Zeitskala der verschiedenen Bewegungen, ermöglicht einen weiteren Einblick in die mechanischen Eigenschaften einzelner Proteine und die Weichheit von Proteinen in ihrer fast natürlichen Umgebung in der überfüllten Proteinlösung.
Introduction
Sondierung der Dynamik weicher Materie mit Neutronen
Die Untersuchung der dynamischen Eigenschaften von Proteinen und Peptiden ist ein wichtiger Teil der biophysikalischen Forschung, und es gibt heute viele gut entwickelte Methoden, um Zugang zu einer Vielzahl von Energielandschaften zu erhalten1. Die experimentell aufgedeckte Dynamik der Proteine mit ihrer biologischen Funktion in Beziehung zu setzen, ist eine weitaus schwierigere Aufgabe, die komplexe mathematische Modelle und computergestützte Dynamiksimulationen erfordert. Die Bedeutung der Neutronenspektroskopie für die Analyse von Proteinbewegungen wurde in mehreren gut aufgenommenen und weithin anerkannten Studienhervorgehoben 1,2,3,4,5. Bevor die vielfältige Energielandschaft der internen Proteindynamik erforscht wird, ist ein kurzer Überblick über die dynamischen Prozesse in weicher Materie erforderlich und wie Neutronen auf sie zugreifen können.
Die Empfindlichkeit von Neutronen gegenüber der Isotopenkonfiguration und die Art der Wechselwirkungen, die sie mit weicher Materie zeigen, macht die Neutronenstreuung zu einer der vielseitigsten Untersuchungstechniken6. Es gibt ein breites Spektrum von Korrelationslängenskalen und Korrelationszeiten, auf die Neutronen zugreifen können, von nuklearen Anregungen und atomaren Schwingungen bis hin zu kollektiven Bewegungen und langsamen Relaxationsprozessen wie isotropen Rotationen und diffusiven Bewegungen. Bei der Untersuchung der gestreuten Neutronen für ihren Energietransfer können drei Hauptwechselwirkungen unterschieden werden: die elastische Streuung, bei der kein Energieaustausch zwischen einfallendem Neutron und Teilchen in der Probe stattfindet; die inelastische Streuung mit einem großen, quantifizierbaren Energieaustausch zwischen Neutron und Teilchen; und der eigentümliche Fall der quasi-elastischen Streuung, der einen sehr kleinen Energietransfer im Vergleich zur einfallenden Neutronenenergie 1,7 bezeichnet. Diese Wechselwirkungen geben präzise Aufschluss über das untersuchte Material und bilden die theoretische Grundlage verschiedenster Neutronenstreutechniken.
Bei der elastischen Streuung zeichnet der Detektor die Richtungen der Neutronen als Beugungsmuster auf, das die Position der Probenatome relativ zueinander zeigt. Es werden Informationen über die Korrelationen atomarer Positionen gewonnen (d.h. integrierte Intensität S(Q) bezüglich der Impulsübertragung Q, die sich nur auf strukturelle Informationen bezieht). Dieses Prinzip bildet die Grundlage der Neutronenbeugung8.
Komplexität entsteht, wenn die Energieübertragung aufgrund von Anregungen und internen Schwankungen im Probenmaterial nicht mehr Null ist. Dies bildet die Grundlage der Neutronenspektroskopie, bei der die gestreuten Neutronen sowohl als Funktion des Energietransfers E als auch des Impulstransfers Q untersucht werden. Es werden dynamische und strukturelle Informationen gewonnen. Die Neutronenspektroskopie misst die gleiche integrierte Intensität S(Q) für den Energietransfer (d.h. Geschwindigkeitsänderung der Neutronen aufgrund der Probenstreuung, S(Q,ω) = S(Q, E), die auch als dynamischer Strukturfaktor bezeichnet wird)9.
Zur Berechnung der Streuung aus einem Material ist es sinnvoller, die Paarkorrelationsfunktion 7,10 zu verwenden. Im Beugungsfall gibt die statische Paarkorrelationsfunktion G(r) die Wahrscheinlichkeit an, den Mittelpunkt eines Teilchens in einem gegebenen Abstand r vom Zentrum eines anderen Teilchens zu finden. Die Spektroskopie verallgemeinert die statische Paarkorrelationsfunktion und bezieht Energie/Frequenz/Zeit in die Streugleichung mit ein. Die Paarkorrelationsfunktion G(r) wird zu einer Funktion der Zeit G(r, t), die in eine ausgeprägte Atompaarkorrelationsfunktion G D(r, t) und eine Selbstkorrelationsfunktion GS(r, t) zerlegt werden kann. Diese beschreiben zwei Arten von Korrelationen: paarkorrelierte Bewegungen von Atomen, die die kohärente Streuung steuern, und Selbstkorrelation, die die inkohärente Streuungsteuert 10.
Kohärente Streuung ist die Streuung vom “Durchschnitt” und hängt von der relativen Phase der gestreuten Wellen ab. Im Kleinwinkelstreuregime interferieren die gestreuten Neutronenwellen aus verschiedenen Streuzentren (verschiedene Atome) konstruktiv (haben ähnliche Phasen), und die kollektive Bewegung der Atome wird mit starker Intensitätsverstärkung beobachtet. Die kohärente Streuung beschreibt im Wesentlichen die Streuung eines einzelnen Neutrons aus allen Kernen in der Probe10.
Wenn keine konstruktive Interferenz zwischen den gestreuten Neutronenwellen aus verschiedenen Zentren auftritt, wird ein einzelnes Atom zeitlich verfolgt und die Selbstkorrelation zwischen der Position des Atoms zum Zeitpunkt t = 0 und demselben Atom zum Zeitpunkt t beobachtet. Somit geht die Information über die relativen Positionen von Atomen verloren, und der Fokus liegt nur auf lokalen Fluktuationen. Die Streuung aus lokalen Schwankungen steuert die inkohärente Streuung. Inkohärente Streuung ist isotrop, trägt zum Hintergrundsignal bei und verschlechtert das Signal-Rauschen10,11.
Wenn wir alle oben genannten Punkte kombinieren, unterscheiden wir vier Hauptneutronenstreuprozesse10: (1) elastisch kohärent (misst die Korrelationen atomarer Positionen), (2) inelastisch kohärent (misst kollektive Bewegungen von Atomen), (3) elastisch inkohärent (trägt zum Hintergrund bei, reduziert die Streuintensität durch den Debye-Waller-Faktor (DWF) und misst den elastischen inkohärenten Strukturfaktor (EISF), der die Geometrie diffusiver Bewegungen in begrenzter Geometrie beschreibt, und (4) inelastisch inkohärent (misst die Dynamik einzelner Atome und die Selbstkorrelation).
Dynamische Prozesse, auf die Neutronen in der Biologie zugreifen können, reichen von der Dämpfung niederfrequenter atomarer und molekularer Schwingungen, der Wechselwirkung von Lösungsmittelmolekülen mit Biooberflächen und Diffusionsprozessen in der Hydratationsschicht von Makromolekülen und begrenzter Geometrie bis hin zu kurzreichweitigen translationalen, rotationalen und taumelnden diffusiven Bewegungen sowie Proteindomänen und allosterischen Bewegungen1 . Die große Vielfalt an Neutronenmethoden und -instrumenten zur Messung der Proteindynamik basiert darauf, wie die Achromatisierung des einfallenden oder ausgehenden Neutronenstrahls erreicht wird und wie die Energieanalyse der gestreuten Neutronen durchgeführt wird. Von Dreiachsen- bis hin zu Flugzeit-, Rückstreu- und Spin-Echo-Spektrometern kann man dynamische Prozesse mit charakteristischen Zeiten zwischen 1 x 10-14 s und 1 x 10-6 s (Femtosekunden bis Mikrosekunden) untersuchen12.
Das Oak Ridge National Laboratory verfügt mit seinen beiden renommierten Neutronenquellen, der Spallation Neutron Source – SNS13 und dem High Isotope Flux Reactor – HFIR14, über eine der besten Suiten von Spektrometern zur Untersuchung der Dynamik in Biomaterialien. Einige der eloquentesten Beispiele sind die Verwendung des Kaltneutronen-Chopper-Spektrometers (CNCS) bei SNS15, um die dynamische Störung von Hydratationswasser um grün fluoreszierendes Protein in Lösung16 oder die kollektiven Schwingungen mehrerer Proteine17 unter Pikosekunden zu untersuchen. Ein wiederkehrendes Problem der Untersuchungen der inelastischen Neutronenstreuung ist, dass einige biologische Prozesse zu langsam sind, um beobachtet zu werden. Ohne extreme Setups, die zu einem enormen Verlust der Neutronenintensität führen, sind Time-of-Flight-Spektrometer auf eine Energieauflösung von 10 μeV begrenzt, was einer maximalen Zeitskala von ~200 ps10,11 entspricht. Dies reicht nicht aus, um großräumige Bewegungen in Proteinen zu beobachten. Daher werden häufig Instrumente mit höherer Energieauflösung wie die Rückstreuspektrometer benötigt. Die Kombination der Flugzeit- und Rückstreutechniken hat sich als leistungsfähig erwiesen, um die Veränderung der internen Dynamik von Cytochrom P450cam (CYP101) zu untersuchen, einem Enzym, das den Hydroxylierungskampfer18 katalysiert.
Die mikroskopische Diffusivität, die mit dem Rückstreuspektrometer an SNS-BASIS19 gemessen wurde, war überraschend gut definiert und konnte in die Diffusivität von Wasser (Hydratation, zytoplasmatisches und massenartiges Wasser) und die Diffusivität von Zellbestandteilen in planarischen Plattwürmern, dem ersten lebenden Tier, das durch Neutronenstreuunguntersucht wurde 20, getrennt werden. . Die Rückstreuung ist eine hochauflösende spektroskopische Technik, die aber auch auf mehrere μeV = mehrere Nanosekunden begrenzt ist, während sich die langsame Dynamik in Biomaterialien auch als Überlebenszeit der Korrelation zwischen atomaren Positionen oder Spinorientierungen manifestiert (z. B. Relaxationsprozesse, die regelmäßig im Zeitbereich von zehn bis Hunderten von Nanosekunden auftreten).
Die Neutronen-Spin-Echo-Spektroskopie (NSE) ist die einzige Neutronenstreutechnik, die eine so hohe Auflösung erreicht. Im Gegensatz zu anderen Neutronentechniken erfordert NSE keine Achromatisierung des Strahls, da es die quantenmechanische Phase der Neutronen verwendet, die ihre magnetischen Momente sind. Die Manipulation magnetischer Momente ermöglicht die Verwendung einer breiten Wellenlängenverteilung des Neutronenstrahls, während die Technik empfindlich auf sehr kleine Neutronengeschwindigkeitsänderungen in der Größenordnung von 1 x 10-4 reagiert. NSE wurde erfolgreich eingesetzt, um die langsame Dynamik von Proteinen in Lösung für viele Proteine zu untersuchen. Unter diesen vielen Pionierstudien erkennen wir die Untersuchung der segmentalen Flexibilität von Schweineimmunglobulin21 an; die gekoppelten Domänenbewegungen in Taq-Polymerase22; die Domänenbewegungen im Tetramer der Hefealkoholdehydrogenase23; die Änderung der Konformation in der Phosphoglyceratkinase bei Substratbindung3; die Aktivierung von Domänenbewegungen und die dynamische Ausbreitung allosterischer Signale im Na+/H+-Exchange Regulatory Cofactor 1 (NHERF1)-Protein4,24,25; die Dynamik eines kompakten Zustands der Quecksilberionenreduktase26; und die Diffusion von Hämoglobin in roten Blutkörperchen27. Zwei neuere Studien zur Proteindynamik haben die Flexibilität des humanen Antikörpers Immunglobulin G (IgG) als entropisches Frühjahr28 und die Eigenschaften des Lösungsmittelbeitrags zur Dynamik des intrinsisch ungeordneten Myelin-Basisproteins (MBP)5 aufgedeckt.
Der vorliegende Artikel erklärt die Grundprinzipien von NSE, die vielfältigen vorbereitenden Methoden, die für eine gründliche Untersuchung der Proteindynamik empfohlen werden, sowie die Methodik und das experimentelle Protokoll für die NSE-Datenerfassung am NSE-Spektrometer an der SNS, SNS-NSE. Das Protokoll charakterisiert zwei Proteine: IgG, ein normales menschliches Antikörperprotein, und das intrinsisch ungeordnete Protein MBP. Die biophysikalischen Implikationen, die Forschungsrelevanz der Beispiele und die Grenzen der Technik werden kurz diskutiert.
NSE-Spektroskopie, die Methode zur Messung der langsamen Dynamik
NSE ist eine polarisierte Technik, die die Neutronenflugzeit verwendet, um den Energieaustausch (Polarisationsverlust) aufgrund der quasi-elastischen Wechselwirkung zwischen Neutronen und Atomen in einer Probe zu messen. Im Kern der NSE-Spektroskopie liegen zwei Grundprinzipien: (1) die Fähigkeit des Neutronenspins, im Magnetfeld mit einer Frequenz proportional zur magnetischen Stärke 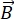 zu präzessieren, nämlich der Larmorfrequenz29, und (b) das Spin-Echo oder Hann-Echo, das die Manipulation und Neufokussierung des Polarisationssignals bei Anlegen einer Reihe von Hochfrequenzimpulsen30 darstellt.
zu präzessieren, nämlich der Larmorfrequenz29, und (b) das Spin-Echo oder Hann-Echo, das die Manipulation und Neufokussierung des Polarisationssignals bei Anlegen einer Reihe von Hochfrequenzimpulsen30 darstellt.
Die Grundlagen des NSE-Prozesses können in einigen einfachen Schritten 6,11 mithilfe von Abbildung 1 zusammengefasst werden. (1) Der von der Quelle erzeugte Neutronenstrahl (Position 1) wird polarisiert (Position 2), geführt und transportiert (Position 3) und erreicht den Eingang des NSE-Spektrometers, wo er vom ersten Pi-Halb-Flipper um 90° gedreht wird (Position 4). (2) Der polarisierte Strahl (z. B. neutronenmagnetische Momente) wird senkrecht zu den Magnetfeldlinien des ersten Magneten (erste Präzessionszone, Position 5) und beginnt zu präzessieren. (3) Am Ende des Magneten akkumulieren Neutronenspins einen bestimmten Präzessionswinkel, der proportional zur magnetischen Feldstärke und der im Inneren verbrachten Flugzeit ist (im Wesentlichen umgekehrt proportional zur Neutronengeschwindigkeit). Die einzelnen Neutronengeschwindigkeiten werden innerhalb ihres Präzessionswinkels am Ende der ersten Präzessionszone kodiert. (4) In der Nähe der Stichprobenposition kehrt der Pi-Flipper (Position 6) die Ausrichtung des Spins um 180° um und ändert das Vorzeichen des Präzessionswinkels. (5) Die Neutronen interagieren mit den Molekülen der Probe (Position 7) und werden gestreut. (6) Die gestreuten Neutronen treten in die zweite Präzessionszone (Position 8) ein und treten in sie vor, werden aber umgekehrt. (7) Ein weiterer Pi-Halb-Flipper (Position 9) wird verwendet, um die Ausrichtung des Spins von senkrecht zur horizontalen Richtung zu drehen. Dadurch wird die Präzession gestoppt, wodurch der Präzessionswinkel φ in eine Polarisation proportional zu cos(φ) übersetzt wird. (8) Der Analysator (Position 10) wählt die Neutronen anhand einer Orientierung aus. Wenn die Wechselwirkung mit der Probe elastisch ist, ändert sich die Geschwindigkeit des Neutrons nicht. Die Neutronen verbringen eine identische Menge an Zeit damit, in der ersten und zweiten Präzessionszone zu fliegen, und die akkumulierten Präzessionswinkel werden vollständig zurückgewonnen. Die volle Polarisation wird auf dem Detektor (Position 11) als Echo der ursprünglichen Polarisation (d.h. Spin-Echo) wiederhergestellt. (9) Bei NSE ist die Streuung jedoch quasi-elastisch, so dass ein kleiner Energieaustausch zwischen Neutronen und Probenmolekülen nach der Streuung durch die Probe zu unterschiedlichen Neutronengeschwindigkeiten führt. Aufgrund der unterschiedlichen Geschwindigkeiten werden die Neutronen eine zusätzliche Zeit damit verbringen, durch die zweite Präzessionszone zu fliegen und ihren Präzessionswinkel nicht richtig wiedererlangt zu haben. Eine partielle Polarisation wird auf dem Detektor abgerufen, und der Verlust der Polarisation aufgrund der Spinrelaxation ist proportional zur cos-Fourier-Transformation der Spektralfunktion S(Q, ω), der Zwischenstreufunktion F(Q, t). (10) Der Zeitparameter der Funktion F(Q, t) ist proportional zur Präzessionsmagnetfeldstärke. Die Abtastung des Polarisationsverlustes in Abhängigkeit von der magnetischen Feldstärke ergibt daher eine Relaxationsfunktion, die von den dynamischen Prozessen innerhalb der Probe abhängt.

Abbildung 1: Aufnahme des NSE-Spektrometers am SNS (SNS-NSE) und Neutronenflugbahnschema mit den wichtigsten Funktionskomponenten. Von rechts nach links: 1 = Neutronenquelle; 2 = Chopper-Bender-Polarizer-Sekundärverschlusssystem; 3 = Leitfäden für den Strahltransport; 4 = Pi/2 Flipper für erste 90° Drehung; 5 = erste Präzessionszone; 6 = Pi-Flipper für 180° Drehung; 7 = Probenfläche und Probenumgebung (hier ist der Kryoofen dargestellt); 8 = zweite Präzessionszone; 9 = Pi/2 Flipper für zweite 90° Drehung; 10 = Analysator; 11 = Detektor. (Beachten Sie, dass sich Teile von 3 sowie 2 und 1 hinter der blauen Wand in der Abschirmung befinden; die Chopper werden durch einen Geschwindigkeitswähler für reaktorbasierte NSE ersetzt.) Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protocol
Representative Results
Discussion
Die NSE-Spektroskopie liefert einen einzigartigen und detaillierten Blick auf die Dynamik von Proteinen, den andere spektroskopische Techniken nicht erzeugen können. Messungen über eine erweiterte Zeitskala ermöglichen Beobachtungen sowohl der translationalen als auch der Rotationsdiffusion der Proteine, wie hier dargestellt. Die segmentale Dynamik und andere interne Schwingungen entpuppen sich als starker Zerfall der kohärenten Streufunktion S(Q, t) auf einer kurzen Zeitskala und sind gut von den gesamten d…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Diese Forschung verwendete Ressourcen an der Spallation Neutron Source (BL-15, BL-6, Biologie und Chemielabors), einem DOE-Büro der Science User Facility, das vom Oak Ridge National Laboratory betrieben wird. Diese Forschung nutzte auch Ressourcen am MLZ-FRM2-Reaktor Garching (KWS-2, Phoenix-J-NSE) und am JCNS1 an der Forschungszentrum Jülich GmbH, Deutschland. Der Autor dankt Dr. Ralf Biehl und Dr. Andreas Stadler für ihre Hilfe bei der Modellierung und ihren Beitrag zur IgG- und MBP-Proteinforschung, Dr. Piotr A. Żołnierczuk für die Unterstützung der NSE-Datenreduktion, Dr. Changwoo Do für die Unterstützung bei SANS-Messungen und Rhonda Moody und Dr. Kevin Weiss für die Unterstützung des SNS-Biochemielabors.
Materials
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O – heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | – | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | – | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | – | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | – | small angle neutron instrument |
| Liquinox dish detergent | Alconox | – | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M – wax parafilm | Bemis | Parafilm M – 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | – | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | – | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | – | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | – | falling ball viscometer |
References
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).

