중성자 스핀 에코 분광법을 통한 단백질 역학 연구
Summary
본 프로토콜은 인간 건강에 중요한 역할을 하는 두 모델 단백질의 구조 및 역학을 조사하는 방법을 기술한다. 이 기술은 벤치 탑 생물 물리학 적 특성화와 중성자 스핀 에코 분광법을 결합하여 단백질 간 운동과 관련된 시간 및 길이 스케일의 역학에 액세스합니다.
Abstract
대부분의 인체 단백질의 활성 및 기능은 단백질 결정 구조 내에서 전체 하위 도메인의 구성 변화와 관련이 있습니다. 결정 구조는 단백질의 구조 또는 역학을 설명하는 모든 계산의 기초를 구축하며, 대부분의 경우 강력한 기하학적 제한이 있습니다. 그러나, 결정 구조로부터의 이러한 제한은 용액 내에 존재하지 않는다. 용액 중의 단백질의 구조는 피코 상의 루프 또는 서브도메인의 재배열로 인해 나노초 시간 스케일(즉, 내부 단백질 동역학 시간 체제)으로 인해 결정과 다를 수 있다. 본 연구는 중성자 산란을 사용하여 수십 나노초의 시간 척도에서 슬로우 모션에 어떻게 접근할 수 있는지를 설명한다. 특히, 잘 정의된 이차 구조와 고전적인 항체 단백질이 결여된 본질적으로 무질서한 단백질인 두 가지 주요 인간 단백질의 동적 특성화는 광범위한 실험실 특성화 방법과 결합된 중성자 스핀 에코 분광법(NSE)에 의해 해결됩니다. 단백질 도메인 역학에 대한 추가 통찰력은 실험 중성자 데이터를 설명하고 결합 된 확산 및 내부 단백질 운동 사이의 교차를 결정하기 위해 수학적 모델링을 사용하여 달성되었습니다. 다양한 움직임의 시간 척도를 포함하여 NSE로부터 얻은 중간 산란 기능에 대한 내부 동적 기여의 추출은 혼잡 한 단백질 용액에서 거의 자연 환경에서 단일 단백질의 기계적 특성과 단백질의 부드러움에 대한 추가 비전을 허용합니다.
Introduction
중성자를 이용한 연질 물질의 역학 조사
단백질과 펩티드의 동적 특성을 조사하는 것은 생물 물리학 연구의 주요 부분이며, 오늘날 광범위한 에너지 환경에 접근하기 위해 많은 잘 발달 된 방법이 존재합니다1. 실험적으로 밝혀진 단백질의 역학을 생물학적 기능과 관련시키는 것은 훨씬 더 어려운 작업이며, 복잡한 수학적 모델과 컴퓨터 지원 역학 시뮬레이션이 필요합니다. 단백질 운동의 분석을위한 중성자 분광법의 중요성은 널리 받아 들여지고 널리 인정 된 여러 연구에서 강조되었습니다 1,2,3,4,5. 내부 단백질 역학의 다양한 에너지 풍경을 탐구하기 전에, 연질 물질의 동적 과정에 대한 간략한 개요와 중성자가 어떻게 접근 할 수 있는지에 대한 간략한 개요가 필요합니다.
동위원소 구성에 대한 중성자의 민감도와 연질물질과의 상호 작용 유형은 중성자 산란을 가장 다양한 조사 기술 중 하나로 만듭니다6. 핵 여기 및 원자 진동에서부터 등방성 회전 및 확산 운동과 같은 느린 이완 과정에 이르기까지 중성자가 접근 할 수있는 상관 길이 척도와 상관 시간의 광범위한 스펙트럼이 있습니다. 흩어져있는 중성자를 에너지 전달을 조사 할 때, 세 가지 주요 상호 작용을 구별 할 수 있습니다 : 샘플에서 들어오는 중성자와 입자 사이에 에너지 교환이없는 탄성 산란; 중성자와 입자 사이의 크고 정량화 가능한 에너지 교환을 갖는 비탄성 산란; 및 입사 중성자 에너지 1,7에 비해 매우 작은 에너지 전달을 지정하는 준탄성 산란의 특이한 경우이다. 이러한 상호 작용은 조사 된 물질에 대한 정확한 정보를 제공하고 다양한 중성자 산란 기술의 이론적 기초를 형성합니다.
탄성 산란에서, 검출기는 중성자의 방향을 회절 패턴으로 기록하며, 이는 서로 상대적인 샘플 원자의 위치를 보여줍니다. 원자 위치의 상관 관계에 대한 정보가 획득됩니다 (즉, 구조 정보에만 관련된 운동량 전달 Q에 관한 통합 강도 S ( Q). 이 원리는 중성자 회절8의 기초를 형성한다.
복잡성은 샘플 재료의 여기 및 내부 변동으로 인해 에너지 전달이 더 이상 제로가 아닐 때 발생합니다. 이것은 중성자 분광법의 기초를 형성하며, 여기서 산란 중성자는 에너지 전달 E와 운동량 전달 Q의 함수로 조사됩니다. 동적 및 구조적 정보가 얻어집니다. 중성자 분광법은 에너지 전달을 위해 동일한 통합 강도 S(Q)를 측정합니다(즉, 샘플 산란으로 인한 중성자의 속도 변화, S(Q,ω) = S(Q, E), 이는 동적 구조 인자라고도 함)9.
재료로부터 산란을 계산하기 위해, 쌍 상관 함수 7,10을 사용하는 것이 더 적절하다. 회절의 경우, 정적 쌍 상관 함수 G(r)는 다른 입자의 중심으로부터 주어진 거리 r에서 입자의 중심을 찾을 확률을 제공합니다. 분광법은 정적 쌍 상관 함수를 일반화하고 산란 방정식에 에너지/주파수/시간을 포함합니다. 쌍 상관 함수 G(r)는 시간 G(r, t)의 함수가 되며, 이는 별개의 원자 쌍 상관 함수 GD(r, t) 및 자기 상관 함수 GS(r, t)로 분해될 수 있다. 이들은 두 가지 유형의 상관 관계를 설명합니다 : 일관된 산란을 지배하는 원자의 쌍 상관 운동과 일관성없는 산란10을 지배하는 자기 상관 관계.
일관된 산란은 “평균”으로부터의 산란이며 산란파의 상대적 위상에 달려 있습니다. 소각 산란 체제에서는 서로 다른 산란 중심 (다른 원자)에서 산란 된 중성자파가 건설적으로 간섭하고 (유사한 위상을 가짐) 원자의 집단 운동이 강한 강도 향상으로 관찰됩니다. 코히어런트 산란은 본질적으로 샘플10 내의 모든 핵으로부터 단일 중성자의 산란을 기술한다.
서로 다른 중심으로부터 산란된 중성자파 사이에 건설적인 간섭이 발생하지 않을 때, 단일 원자가 시간에 뒤따르고, 시간 t=0에서의 원자의 위치와 시간 t에서의 동일한 원자 사이의 자기-상관관계가 관찰된다. 따라서 원자의 상대적 위치에 대한 정보는 손실되고 초점은 국부적 인 변동에만 있습니다. 국소 변동으로 인한 산란은 일관성없는 산란을 지배합니다. 비일관성 산란은 등방성이며, 배경 신호에 기여하며, 신호 대 잡음(10,11)을 저하시킨다.
위의 모든 것을 결합하여 우리는 네 가지 주요 중성자 산란 과정을 구별합니다10 : (1) 탄성 일관성 (원자 위치의 상관 관계 측정), (2) 비탄성 일관성 (원자의 집단 운동 측정), (3) 탄성 불일치 (배경에 기여, Debye-Waller 요인 (DWF)에 의한 산란 강도 감소 및 탄성 불일치 구조 인자 (EISF)를 측정하여 제한된 기하학에서 확산 운동의 기하학을 설명하고, (4) 비탄성 일관성 (단일 원자 역학 및 자기 상관 관계 측정).
중성자가 생물학에서 접근할 수 있는 역학 과정은 저주파 원자 및 분자 진동의 감쇠, 용매 분자와 생체 표면의 상호작용, 거대분자와 제한된 기하학의 수화층에서의 확산 과정, 단거리 번역, 회전 및 텀블링 확산 운동, 단백질 도메인 및 알로스테릭 동작에 이르기까지 다양합니다1 . 단백질 역학을 측정하기위한 중성자 방법 및 장비의 광범위한 다양성은 입사 또는 나가는 중성자 빔의 무분화가 어떻게 달성되고 산란 된 중성자의 에너지 분석이 어떻게 수행되는지에 기반을두고 있습니다. 삼중 축에서 비행 시간, 후방 산란 및 스핀 에코 분광계에 이르기까지 1 x 10-14 초와 1 x 10-6 초 (펨토 초 ~ 마이크로 초)12 사이의 특성 시간으로 동적 프로세스를 탐색 할 수 있습니다.
오크 리지 국립 연구소는 두 개의 유명한 중성자 공급원 인 Spallation Neutron Source – SNS13 및 High Isotope Flux Reactor – HFIR14와 함께 바이오 재료의 역학을 조사하기위한 분광계의 최고의 스위트 룸 중 하나입니다. 가장 웅변적인 예들 중 일부는 용액16에서 녹색 형광 단백질 주위의 수화수의 동적 섭동 또는 몇몇 단백질(17)의 서브피코초 집단 진동을 조사하기 위해 SNS15에서 차가운 중성자 초퍼 분광계(CNCS)의 사용을 포함한다. 비탄성 중성자 산란 조사의 반복되는 문제는 일부 생물학적 과정이 관찰되기에는 너무 느리다는 것입니다. 중성자 강도의 엄청난 손실을 초래하는 극단적인 설정이 없다면, 비행 시간 분광계는 ~200ps 10,11의 최대 시간 스케일에 해당하는 10μeV 에너지 분해능으로 제한됩니다. 이것은 단백질의 대규모 움직임을 관찰하기에 충분하지 않습니다. 따라서 후방 산란 분광계와 같이 더 높은 에너지 분해능을 가진 장비가 종종 필요합니다. 비행 시간 및 후방 산란 기술을 결합하는 것은 히드록실 화 캄포어18을 촉매하는 효소인 시토크롬 P450cam(CYP101)의 내부 역학의 변화를 조사하는 데 강력한 것으로 입증되었습니다.
SNS-BASIS19 에서 후방 산란 분광계에 의해 측정된 현미경 확산도는 놀랍게도 잘 정의되었고, 물의 확산성(수화, 세포질 및 벌크 유사물)과 중성자 산란에 의해 연구된 최초의 살아있는 동물인 평면 편평충에서의 세포 구성성분의 확산성으로 분리될 수 있었다20 . 후방 산란은 고분해능 분광 기술이지만 수 μeV = 수 나노초로 제한되는 반면, 생체 재료의 느린 역학은 원자 위치 또는 스핀 배향 사이의 상관 관계의 생존 시간으로 나타납니다 (예 : 열 나노 초에서 수백 나노 초의 시간 범위에서 정기적으로 발생하는 이완 과정).
중성자 스핀 에코 분광법 (NSE)은 이러한 고해상도에 도달하는 유일한 중성자 산란 기술입니다. 다른 중성자 기술과는 달리, NSE는 자기 모멘트인 중성자의 양자 기계적 위상을 사용하기 때문에 빔의 무채색화를 필요로 하지 않습니다. 자기 모멘트의 조작은 광범위한 중성자 빔 파장 분포의 사용을 허용하는 반면, 기술은 1 x 10-4의 순서로 매우 작은 중성자 속도 변화에 민감합니다. NSE는 많은 단백질에 대한 용액에서 단백질의 느린 역학을 조사하는 데 성공적으로 사용되었습니다. 이러한 많은 선구자 연구 중에서, 우리는 돼지 면역 글로불린21의 세그먼트 유연성에 대한 연구를 인정합니다. Taq 중합효소22에서 결합된 도메인 운동; 효모 알콜 탈수소효소23의 사량체에서의 도메인 운동; 기질 결합시 포스포글리세레이트 키나아제에서의 입체 형태 변화3; 도메인 운동의 활성화 및 Na+/H+ 교환 조절 보조인자 1 (NHERF1) 단백질 4,24,25에서의 알로스테릭 신호의 동적 전파; 수은 이온 환원효소26의 콤팩트한 상태의 역학; 적혈구에서 헤모글로빈의 확산27. 단백질 역학에 대한 최근의 두 가지 연구는 엔트로픽 스프링28로서의 인간 항체 면역 글로불린 G (IgG)의 유연성과 본질적으로 무질서한 미엘린 기본 단백질 (MBP)5의 역학에 기여하는 용매의 특성을 드러냈다.
본 기사에서는 NSE의 기본 원칙, 철저한 단백질 역학 조사에 권장되는 여러 가지 준비 방법, SNS, SNS-NSE의 NSE 분광계에서 NSE 데이터 수집을위한 방법론 및 실험 프로토콜에 대해 설명합니다. 이 프로토콜은 두 가지 단백질을 특징으로합니다 : IgG, 일반 인간 항체 단백질 및 본질적으로 무질서한 단백질 MBP. 생물 물리학 적 함의, 실시예의 연구 관련성 및 기술의 한계가 간략하게 논의됩니다.
NSE 분광법, 느린 역학 측정을위한 방법
NSE는 중성자 비행 시간을 사용하여 샘플에서 중성자와 원자 간의 준 탄성 상호 작용으로 인한 에너지 교환 (분극 손실)을 측정하는 편광 기술입니다. NSE 분광법의 핵심에는 두 가지 기본 원칙이 있습니다 : (1) 중성자 스핀이 자기 강도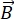 에 비례하는 주파수, 즉 Larmor 주파수 (29)를 가진 자기장에서 세차하는 능력, (b) 일련의 고주파 펄스 (30)를 적용 할 때 편광 신호의 조작 및 재초점을 나타내는 스핀 에코 또는 한 에코.
에 비례하는 주파수, 즉 Larmor 주파수 (29)를 가진 자기장에서 세차하는 능력, (b) 일련의 고주파 펄스 (30)를 적용 할 때 편광 신호의 조작 및 재초점을 나타내는 스핀 에코 또는 한 에코.
NSE 프로세스의 기본 사항은 그림 1을 사용하여 몇 가지 간단한 단계 6,11로 요약할 수 있습니다. (1) 소스(위치 1)에 의해 생성된 중성자 빔은 편광되고(위치 2), 안내되고, 운반되고(위치 3), NSE 분광계의 입구에 도착하며, 여기서 첫 번째 파이-하프 플리퍼(위치 4)에 의해 90° 회전한다. (2) 편광 빔 (예를 들어, 중성자 자기 모멘트)은 첫 번째 자석의 자기장 라인 (첫 번째 세차 영역, 위치 5)에 수직이되어 세차하기 시작합니다. (3) 자석의 끝에서, 중성자 스핀은 자기장 강도와 내부에서 소비되는 비행 시간에 비례하는 특정 세차각을 축적한다(기본적으로 중성자 속도에 반비례한다). 개별 중성자 속도는 첫 번째 세차전 구역의 끝에서 세차각 각도 내에서 인코딩됩니다. (4) 샘플 위치에 가까우면 파이 플리퍼 (위치 6)는 스핀의 방향을 180 ° 반전시켜 세차 각도의 부호를 변경합니다. (5) 중성자는 샘플의 분자(위치 7)와 상호작용하여 산란된다. (6) 흩어져있는 중성자는 두 번째 세차 구역 (위치 8)에 들어가고 세차하지만 역방향이된다. (7) 또 다른 파이 하프 플리퍼 (위치 9)는 스핀의 방향을 수직에서 수평 방향으로 회전시키는 데 사용됩니다. 이것은 세차운동을 멈추게 할 것이고, 세차전 각도 φ을 cos(φ)에 비례하는 편광으로 변환시킬 것이다. (8) 분석기(위치 10)는 하나의 배향에 기초하여 중성자를 선택한다. 샘플과의 상호 작용이 탄력적이면 중성자의 속도는 변하지 않습니다. 중성자는 첫 번째와 두 번째 세차 구역에서 동일한 시간을 비행하며 누적 된 세차 각도가 완전히 회복됩니다. 전체 편광은 검출기(위치 11) 상에서 원래의 편광(즉, 스핀-에코)의 에코로서 복원된다. (9) 그러나 NSE에서는 산란이 준 탄성이므로 중성자와 샘플 분자 간의 작은 에너지 교환은 샘플에 의한 산란 후 다른 중성자 속도로 이어집니다. 속도가 다르기 때문에 중성자는 두 번째 세차 구역을 통해 비행하는 데 추가 시간을 보내고 세차 각도를 제대로 회복하지 못합니다. 부분 분극이 검출기 상에서 검색되고, 스핀 이완으로 인한 편광의 손실은 스펙트럼 함수 S(Q, ω), 중간 산란 함수 F(Q, t)의 cos-Fourier-변환에 비례한다. (10) 함수 F(Q, t)의 시간 파라미터는 세차전 자기장 강도에 비례한다. 자기장 강도의 함수로서 분극의 손실을 주사하는 것은 샘플 내의 동적 과정에 의존하는 이완 기능을 산출한다.

그림 1: SNS(SNS-NSE)에서의 NSE 분광계와 가장 중요한 기능성 성분을 사용한 중성자 비행 경로 회로도의 사진. 오른쪽에서 왼쪽으로: 1 = 중성자 소스; 2 = 초퍼-벤더-편광판-2차 셔터 시스템; 3 = 빔 수송 가이드; 4 = 처음 90° 스핀 턴에 대한 pi/2 플리퍼; 5 = 첫 번째 세차운동장; 6 = 180° 스핀 턴을 위한 파이 플리퍼; 7 = 샘플 영역 및 샘플 환경(여기서, 냉동로가 도시됨); 8 = 두 번째 세차 구역; 9 = 두 번째 90° 스핀 턴에 대한 pi/2 플리퍼; 10 = 분석기; 11 = 검출기. (3과 2와 1의 일부는 차폐 내부의 파란색 벽 뒤에 위치하며, 초퍼는 원자로 기반 NSE의 속도 선택기로 대체됩니다.) 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
NSE 분광법은 다른 분광학적 기술이 생산할 수 없는 단백질의 역학에 대한 독특하고 상세한 시각을 제공합니다. 연장된 시간 척도에 걸친 측정은 여기에 제시된 바와 같이 단백질의 번역 및 회전 확산 둘 다에 대한 관찰을 제공한다. 분절 역학 및 기타 내부 진동은 짧은 시간 규모에서 일관된 산란 함수 S(Q, t)의 강한 붕괴로 나타나며 전체 확산 완화 과정과 잘 분리되어 있습니다. NSE 기술의 ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 연구는 Oak Ridge National Laboratory가 운영하는 과학 사용자 시설의 DOE 사무소 인 Spallation Neutron Source (BL-15, BL-6, Biology and Chemistry labs)의 자원을 사용했습니다. 이 연구는 또한 MLZ-FRM2 원자로 Garching (KWS-2, Phoenix-J-NSE)과 독일 Forschungszentrum Jülich GmbH의 JCNS1에서 자원을 사용했습니다. 저자는 Ralf Biehl 박사와 Andreas Stadler 박사가 모델링에 도움을 주고 IgG와 MBP 단백질 연구에 기여한 공로를 인정하고, NSE 데이터 감소 지원을 위한 Piotr A. Żołnierczuk 박사, SANS 측정 지원을 위한 Changwoo Do 박사, SNS 생화학 실험실 지원을 위한 Rhonda Moody와 Kevin Weiss 박사를 인정합니다.
Materials
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O – heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | – | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | – | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | – | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | – | small angle neutron instrument |
| Liquinox dish detergent | Alconox | – | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M – wax parafilm | Bemis | Parafilm M – 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | – | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | – | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | – | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | – | falling ball viscometer |
References
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).

