تصوير الخلايا الحية للصفائف أحادية الخلية (LISCA) - تقنية متعددة الاستخدامات لقياس الحركية الخلوية
Summary
نحن نقدم طريقة للحصول على دورات زمنية لمراسل مضان من خلايا واحدة باستخدام صفائف micropatterned. ويصف البروتوكول إعداد صفائف أحادية الخلية، وإعداد وتشغيل المجهر الفاصل الزمني لمسح الخلايا الحية، وأداة تحليل الصور مفتوحة المصدر للاختيار المسبق الآلي، والتحكم البصري وتتبع دورات زمنية مضانة متكاملة للخلايا لكل موقع التصاق.
Abstract
تصوير الخلايا الحية للصفائف أحادية الخلية (LISCA) هو طريقة متعددة الاستخدامات لجمع الدورات الزمنية للإشارات الفلورية من الخلايا الفردية في الإنتاجية العالية. بشكل عام، يعوق الحصول على دورات الوقت خلية واحدة من الخلايا المستزرعة من قبل حركة الخلية وتنوع أشكال الخلايا. صفائف صغيرة لاصقة توحيد ظروف خلية واحدة وتسهيل تحليل الصورة. تجمع LISCA بين الميكروبات أحادية الخلية مع الفحص المجهري الفاصل زمنيا ومعالجة الصور الآلية. هنا، نقوم بوصف الخطوات التجريبية لأخذ دورات زمنية للفلورسينس أحادية الخلية في شكل LISCA. نحن خلايا العدوى المنضمة إلى صفيف micropatterned باستخدام ترميز مرنا لتعزيز البروتين الفلوري الأخضر (eGFP) ورصد الحركية التعبير eGFP من مئات الخلايا بالتوازي عن طريق المسح الضوئي المجهر الفاصل الزمني. تتم معالجة مداخن بيانات الصورة تلقائيا بواسطة برامج مطورة حديثا تدمج كثافة الفلورسينس على معالم الخلايا المحددة لإنشاء دورات زمنية للفلورسينس أحادية الخلية. نحن نثبت أن دورات وقت التعبير eGFP بعد نقل الحمض النووي الريبي موصوفة بشكل جيد من خلال نموذج ترجمة حركي بسيط يكشف عن معدلات التعبير والتدهور في الحمض النووي الريبي. تتم مناقشة تطبيقات أخرى من LISCA لارتباطات وقت الحدث من علامات متعددة في سياق الإشارة إلى موت الخلايا المبرمج.
Introduction
في السنوات الأخيرة، أصبحت أهمية التجارب ذات الخلية الواحدة واضحة. تسمح البيانات من خلايا واحدة بالتحقيق في تقلب الخلية إلى الخلية ، وحل الارتباطات المعلمة داخل الخلية والكشف عن الحركية الخلوية التي لا تزال مخفية فيقياسات الفرقة 1و2و3. من أجل التحقيق في الحركية الخلوية لآلاف الخلايا المفردة بالتوازي ، هناك حاجة إلى نهج جديدة تمكن من مراقبة الخلايا في ظل ظروف موحدة على مدى فترة زمنية تصل إلى عدة ساعات إلى عدة أيام تليها تحليل البيانات الكمية 4. هنا ، نقدم تصوير الخلايا الحية للصفائف أحادية الخلية (LISCA) ، والذي يجمع بين استخدام الصفائف المجهرية مع المجهر الفاصل الزمني وتحليل الصور الآلي.
وقد تم إنشاء عدة طرق لتوليد صفائف الخلية الواحدة microstructured ونشرت في الأدب5،6. هنا، ونحن نصف بإيجاز Microscale البلازما التي بدأت نمط البروتين (μPIPP). كما تم العثور على بروتوكول مفصل لتصنيع صفيف خلية واحدة باستخدام μPIPP في المرجع7. استخدام صفائف وحيدة الخلية تمكن من محاذاة الآلاف من الخلايا على بقع التصاق موحدة تقديم البيئات الدقيقة المحددة لكل خلية، وبالتالي يقلل من مصدر للتغير التجريبي(الشكل 1A). وتستخدم صفائف أحادية الخلية لرصد المسارات الزمنية للعلامات الفلورية التي تهدف إلى الإشارة إلى مجموعة متنوعة من العمليات الخلوية. يسمح الفحص المجهري طويل الأجل في وضع الفاصل الزمني المسح الضوئي بمراقبة مساحة كبيرة من صفائف الخلية الواحدة وبالتالي أخذ عينات من بيانات الخلية الواحدة في إنتاجية عالية على مدى عدة ساعات أو حتى أيام. وهذا يولد مداخن خط الوقت من الصور من كل موقف من الصفيف (الشكل 1B). من أجل تقليل كمية كبيرة من بيانات الصورة واستخراج المقررات الزمنية ذات الخلية الواحدة المطلوبة بطريقة فعالة ، يلزم معالجة الصور الآلية التي تستفيد من تحديد مواقع الخلايا(الشكل 1C).
ويتمثل التحدي الذي تواجهه اللجنة في تكييف البروتوكولات التجريبية والأدوات الحاسوبية لتشكيل تقييم عالي الإنتاجية يولد بيانات كمية وقابلة للاستنساخ من الحركية الخلوية. في هذه المقالة نقدم وصفا خطوة بخطوة للأساليب الفردية وكيفية دمجها في اختبار LISCA. على سبيل المثال، نناقش المسار الزمني للتعبير المحسن عن البروتين الفلوري الأخضر (eGFP) بعد تسليم الحمض النووي الريبي الاصطناعي. يوصف التعبير eGFP بعد تسليم مرنا من قبل معادلات معدل رد الفعل النمذجة الترجمة وتدهور مرنا. إن تركيب وظيفة النموذج للمسار الزمني لتركيز eGFP مع قراءات LISCA لشدة الفلورسينس لكل خلية على حدة بمرور الوقت يؤدي إلى أفضل تقديرات لمعلمات النموذج مثل معدل تدهور الحمض النووي الريبي. ونتيجة لذلك، نناقش كفاءة تسليم الحمض النووي الريبي (mRNA) لاثنين من عوامل العدوى المختلفة القائمة على الدهون وكيف تختلف توزيعات المعلمات الخاصة بها.
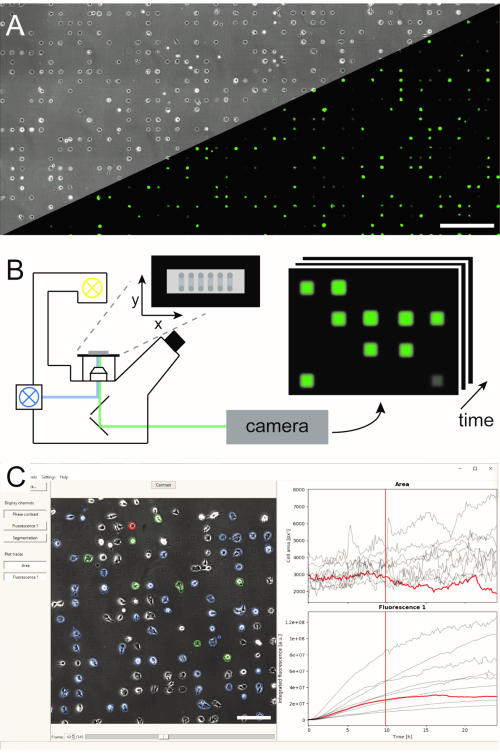
الشكل 1:تمثيل سير عمل LISCA الذي يجمع (أ) صفائف أحادية الخلية صغيرة النمط (B) المسح الضوئي الفاصل الزمني وتحليل الصورة الآلي (C) لسلسلة الصور المسجلة. تتكون صفائف الخلية الواحدة من نمط ثنائي الأبعاد من المربعات اللاصقة للخلايا مع مساحة مشتركة طاردة للخلايا تؤدي إلى ترتيب الخلايا على الميكروبيترن ، كما يمكن رؤيته في صورة تباين المرحلة وكذلك صورة الفلورية لخلايا التعبير عن eGFP (A). يتم تصوير المنطقة المجهرية بأكملها في وضع الفاصل الزمني المسح الضوئي التقاط الصور مرارا وتكرارا في سلسلة من المواقف(B). تتم معالجة سلسلة الصور المسجلة لقراءة كثافة الفلورسينس لكل خلية مع مرور الوقت (C). أشرطة المقياس: 500 ميكرومتر (A)، 200 ميكرومتر (C). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
Representative Results
Discussion
هنا وصفنا LISCA بأنها تقنية متعددة الاستخدامات لمتابعة الحركية الخلوية للتسميات الفلورية داخل الخلايا على مستوى الخلية الواحدة. من أجل إجراء تجربة LISCA ناجحة، يجب تأسيس كل خطوة من الخطوات الموضحة من مقطع البروتوكول بشكل فردي ومن ثم يجب دمج كافة الخطوات. كل واحد من الجوانب الرئيسية الثلاثة لل…
Disclosures
The authors have nothing to disclose.
Acknowledgements
وقد تم دعم هذا العمل من خلال منح من المؤسسة الألمانية للعلوم (DFG) إلى مركز البحوث التعاونية (SFB) 1032. يتم الاعتراف بامتنان بدعم وزارة التعليم والبحوث والتكنولوجيا الاتحادية الألمانية (BMBF) في إطار المشروع التعاوني 05K2018-2017-06716 Medisoft بالإضافة إلى منحة من بايريشفورشونجستفتونغ. تم دعم أنيتا رايزر من خلال زمالة DFG من خلال كلية الدراسات العليا للعلوم الحيوية الكمية في ميونيخ (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
References
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

