세포 역학을 정량화하는 다목적 기술인 단세포 어레이(LISCA)의 라이브 셀 이미징
Summary
마이크로패턴 어레이를 사용하여 단일 세포로부터 형광 리포터 타임 코스를 취득하는 방법을 제시합니다. 이 프로토콜은 단일 셀 어레이의 준비, 라이브 셀 스캐닝 시간 경과 현미경 검사법의 설정 및 작동 및 접착 부위당 세포 통합 형광 시간 과정의 자동화된 사전 선택, 시각적 제어 및 추적을 위한 오픈 소스 이미지 분석 도구에 대해 설명합니다.
Abstract
단일 세포 배열의 라이브 셀 이미징 (LISCA)는 높은 처리량의 개별 세포로부터 형광 신호의 시간 과정을 수집하는 다양한 방법입니다. 일반적으로, 배양 된 세포에서 단세포 시간 과정의 취득은 세포 운동성 및 세포 모양의 다양성에 의해 방해된다. 접착제 마이크로 어레이는 단일 셀 조건을 표준화하고 이미지 분석을 용이하게 합니다. LISCA는 단일 셀 마이크로어레이와 스캔 시간 경과 현미경 검사및 자동화된 이미지 처리를 결합합니다. 여기서는 LISCA 형식으로 단일 세포 형광 시간 과정을 취하는 실험 단계를 설명합니다. 당사는 향상된 녹색 형광 단백질(eGFP)을 위해 mRNA 인코딩을 사용하여 마이크로패턴 어레이에 부착된 세포를 트랜스펙트하고 스캔 시간 경과 현미경 검사를 통해 수백 개의 세포의 eGFP 발현 운동을 병렬로 모니터링합니다. 이미지 데이터 스택은 선택한 셀 윤곽에 형광 강도를 통합하여 단일 세포 형광 시간 과정을 생성하는 새로 개발된 소프트웨어에 의해 자동으로 처리됩니다. 우리는 mRNA 이식 후 eGFP 발현 시간 과정이 mRNA의 발현 및 저하 율을 드러내는 간단한 운동 번역 모델에 의해 잘 설명된다는 것을 보여줍니다. 사멸 신호의 맥락에서 여러 마커의 이벤트 시간 상관 관계에 대한 LISCA의 추가 응용 프로그램에 대해 설명합니다.
Introduction
최근 몇 년 동안, 단세포 실험의 중요성이 명백해지고 있다. 단일 세포로부터의 데이터는 세포 대 세포 가변성, 세포 내 파라미터 상관관계의 해상도 및 앙상블 측정1,2,3에숨겨져 있는 세포 역학의 검출을 조사할 수 있다. 수천 개의 단일 세포의 세포 역학을 병렬로 조사하기 위해서는 수 시간까지 며칠 동안 표준화된 조건하에서 세포를 모니터링할 수 있도록 하는 새로운 접근법이 필요하며, 이어서 정량적 데이터 분석 4. 여기서는 미세구조화 어레이의 사용과 시간 경과 현미경 검사및 자동화된 이미지 분석을 결합한 단일 셀 어레이(LISCA)의 라이브 셀 이미징을 소개합니다.
미세 구조화 된 단세포 어레이를 생성하기위한 몇 가지 방법이 수립되어 문헌5,6에출판되었다. 여기에서, 우리는 마이크로 스케일 플라즈마 개시 단백질 패턴화 (μPIPP)를 간단히 설명합니다. μPIPP를 이용한 단일 세포 어레이 제조의 상세한 프로토콜도 참조7에서발견된다. 단일 세포 어레이의 사용은 표준화된 접착 반점에 수천 개의 셀의 정렬을 가능하게 하여 각 세포에 대해 정의된 마이크로환경을 제시하여 실험적 가변성의 근원을 감소시킵니다(그림1A). 단일 세포 배열은 다양한 세포 과정을 나타내기 위해 목적으로 사용되는 형광 마커의 시간 과정을 모니터링하는 데 사용됩니다. 스캔 시간 경과 모드에서 장기 현미경 검사는 단일 셀 어레이의 넓은 영역을 모니터링하고 따라서 몇 시간 또는 며칠의 관측 시간에 걸쳐 높은 처리량의 단일 세포 데이터를 샘플링 할 수 있습니다. 이렇게 하면 배열의 각 위치에서 이미지의 시간 줄스택(그림 1B)이생성됩니다. 다량의 영상 데이터를 줄이고 원하는 단일 세포 형광 시간 과정을 효율적인 방법으로 추출하기 위해서는 셀의 위치지정(도 1C)을활용하는 자동화된 이미지 처리가 요구된다.
LISCA의 과제는 실험 프로토콜과 계산 도구를 조정하여 세포 역학의 정량적이고 재현 가능한 데이터를 생성하는 높은 처리량 분석기를 형성하는 것입니다. 이 문서에서는 개별 메서드와 LISCA 분석기에서 결합하는 방법에 대한 단계별 설명을 제공합니다. 예를 들어, 인공 mRNA 전달 후 향상된 녹색 형광 단백질(eGFP) 발현의 시간 과정에 대해 논의합니다. mRNA 전달 에 따른 eGFP 발현은 mRNA의 번역 및 분해를 모델링하는 반응 속도 방정식에 의해 설명된다. 시간이 지남에 따라 각 개별 셀에 대한 형광 강도의 LISCA 판독값에 eGFP 농도의 시간 과정에 대한 모델 기능을 피팅하면 mRNA 분해 속도와 같은 모델 파라미터의 가장 좋은 추정치를 산출합니다. 대표적인 결과로 우리는 두 개의 다른 지질 계 형 경화 에이전트의 mRNA 전달 효율과 매개 변수 분포가 어떻게 다른지에 대해 논의합니다.
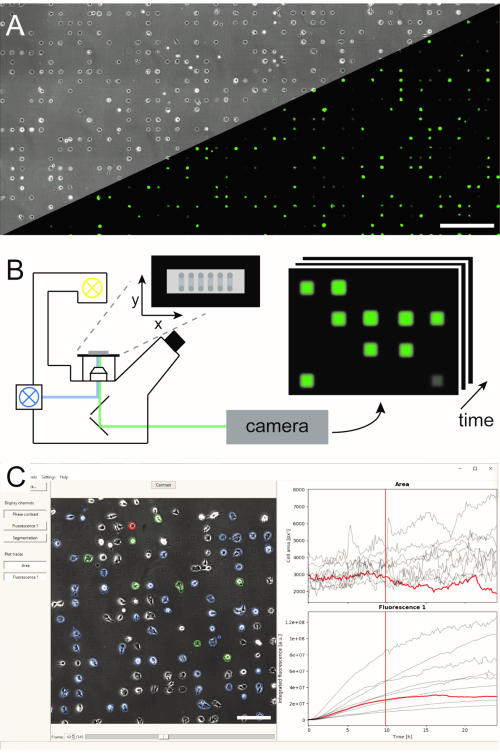
그림 1: LISCA 워크플로우의 표현 (A) 마이크로 패턴 단일 셀 어레이 (B) 스캔 시간 경과 현미경 및 (C) 기록 된 이미지 시리즈의 자동화 된 이미지 분석. 단세포 어레이는 세포-발충제 간 공간을 가진 세포-접착제 사각형의 2차원 패턴으로 구성되어 마이크로패턴상에 있는 세포의 배열로 이어지는데, 이는 상 대비 영상뿐만 아니라 eGFP 발현 세포의 형광 이미지에서 볼 수있다(A). 전체 미세 구조화 영역은 반복적으로 위치의 시퀀스에서 이미지를 촬영 스캔 시간 경과 모드에서 이미지(B). 기록된 이미지 시리즈는 시간이 지남에 따라 세포당 형광 강도를판독하기 위해 처리된다(C). 스케일 바: 500 μm(A),200 μm(C). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
여기서 우리는 LISCA를 단세포 수준에서 세포내 형광 라벨의 세포 역학을 따르는 다목적 기술로 기술했습니다. 성공적인 LISCA 실험을 수행하려면 프로토콜 섹션의 각 설명된 단계를 개별적으로 수립해야 하며 모든 단계를 결합해야 합니다. LISCA의 세 가지 주요 측면은 각각 중요한 단계를 특징으로합니다.
단세포 마이크로어레이 제작
마이크로어레이의 셀룰러 정렬은 …
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 작품은 독일 과학 재단 (DFG)에서 공동 연구 센터 (SFB) 1032에 보조금에 의해 지원되었다. 독일 연방 교육기술부(BMBF)의 협력 프로젝트 05K2018-06716 메디소프와 바이에리슈 포르스스티퉁의 보조금에 감사하게 인정받고 있다. 아니타 라이저는 정량적 생물 과학 뮌헨 (QBM)의 대학원을 통해 DFG 펠로우십에 의해 지원되었다.
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
References
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

