Live-celle bildebehandling av encellede matriser (LISCA) - en allsidig teknikk for å kvantifisere cellulær kinetikk
Summary
Vi presenterer en metode for oppkjøp av fluorescensreportertidskurs fra enkeltceller ved hjelp av mikropatterned arrays. Protokollen beskriver utarbeidelsen av encellede matriser, oppsett og drift av sanntidsskanningstidsforløpmikroskopi og et bildeanalyseverktøy med åpen kildekode for automatisert forhåndsvalg, visuell kontroll og sporing av celleintegrerte fluorescenstidskurs per adhesjonssted.
Abstract
Live-cell Imaging of Single-Cell Arrays (LISCA) er en allsidig metode for å samle inn tidsforløp for fluorescenssignaler fra individuelle celler i høy gjennomstrømning. Generelt er oppkjøpet av encellede tidskurs fra dyrkede celler hemmet av cellemotilitet og mangfold av celleformer. Selvklebende mikrokjeder standardiserer encellede forhold og letter bildeanalyse. LISCA kombinerer encellede mikroarrayer med skanning av tidsforløpmikroskopi og automatisert bildebehandling. Her beskriver vi de eksperimentelle trinnene for å ta encellede fluorescenstidskurs i ET LISCA-format. Vi transfekterer celler som holder seg til et mikropatterned array ved hjelp av mRNA-koding for forbedret grønt fluorescerende protein (eGFP) og overvåker eGFP-uttrykkskinetikken til hundrevis av celler parallelt via skanning av tidsforløpmikroskopi. Bildedatastakkene behandles automatisk av nyutviklet programvare som integrerer fluorescensintensitet over valgte cellekonturer for å generere encellede fluorescenstidskurs. Vi viser at eGFP-uttrykkstidskurs etter mRNA-transfeksjon er godt beskrevet av en enkel kinetisk oversettelsesmodell som avslører uttrykks- og nedbrytningshastigheter for mRNA. Videre anvendelser av LISCA for hendelsestidskorrelasjoner av flere markører i sammenheng med signalering av apoptose diskuteres.
Introduction
I de senere år har betydningen av encellede eksperimenter blitt tydelig. Data fra enkeltceller tillater undersøkelse av celle-til-celle-variasjon, oppløsningen av intracellulære parameterkorrelasjoner og påvisning av cellulær kinetikk som forblir skjult i ensemblemålinger1,2,3. For å undersøke cellulær kinetikk av tusenvis av enkeltceller parallelt, er det nødvendig med nye tilnærminger som gjør det mulig å overvåke cellene under standardiserte forhold over en tidsperiode på flere timer opptil flere dager etterfulgt av en kvantitativ dataanalyse 4. Her presenterer vi Live-cell Imaging of Single-Cell Arrays (LISCA), som kombinerer bruken av mikrostrukturerte matriser med tidsforløpmikroskopi og automatisert bildeanalyse.
Flere metoder for å generere mikrostrukturerte encellede matriser er etablert og publisert i litteratur5,6. Her beskriver vi kort mikroskala plasmainitiert proteinmønster (μPIPP). En detaljert protokoll for enkeltcellet matriseproduksjon ved hjelp av μPIPP finnes også i referanse7. Bruken av encellede matriser gjør det mulig å justere tusenvis av celler på standardiserte adhesjonspunkter som presenterer definerte mikromiljø for hver celle og dermed reduserer en kilde til eksperimentell variasjon (Figur 1A). Encellede matriser brukes til å overvåke tidsforløpene til fluorescerende markører med formål å indikere en rekke cellulære prosesser. Langsiktig mikroskopi i skannetidsforløpmodus gjør det mulig å overvåke et stort område av encellede matriser og dermed prøvetaking av encellede data i høy gjennomstrømning over en observasjonstid på flere timer eller til og med dager. Dette genererer tidslinjestakker med bilder fra hver plassering av matrisen (Figur 1B). For å redusere den store mengden bildedata og trekke ut de ønskede enkeltcellede fluorescenstidskursene på en effektiv måte, kreves automatisert bildebehandling som utnytter plasseringen av celler (Figur 1C).
Liscas utfordring er å tilpasse eksperimentelle protokoller og beregningsverktøy for å danne en analyse med høy gjennomstrømning som genererer kvantitative og reproduserbare data om cellulær kinetikk. I denne artikkelen gir vi en trinnvis beskrivelse av de enkelte metodene og hvordan de kombineres i en LISCA-analyse. Som et eksempel diskuterer vi tidsforløpet for forbedret grønt fluorescerende proteinuttrykk (eGFP) etter kunstig mRNA-levering. eGFP-uttrykk etter mRNA-levering er beskrevet ved reaksjonshastighetsligninger modelleringsoversettelse og nedbrytning av mRNA. Montering av modellfunksjonen for tidsforløpet av eGFP-konsentrasjon til LISCA-avlesningen av fluorescensintensiteten for hver enkelt celle over tid gir de beste estimatene av modellparametere som mRNA-nedbrytningshastigheten. Som et representativt resultat diskuterer vi mRNA-leveringseffektiviteten til to forskjellige lipidbaserte transfeksjonsmidler og hvordan deres parameterfordelinger er forskjellige.
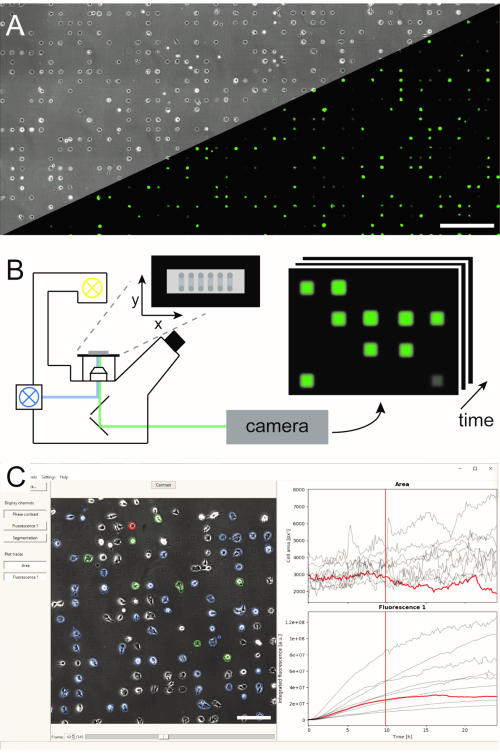
Figur 1: Representasjon av LISCA-arbeidsflyten som kombinerer (A) mikromønstrede encellede matriser (B) som skanner tidsforløpmikroskopi og (C) automatisert bildeanalyse av innspilte bildeserier. Encellede matriser består av et todimensjonalt mønster av celleklebende firkanter med et celleavvisende interspace som fører til et arrangement av cellene på mikropatternen, som det fremgår av fasekontrastbildet samt fluorescensbildet av eGFP som uttrykker celler (A). Hele det mikrostrukturerte området er avbildet i en tidsforløpmodus for skanning gjentatte ganger ved å ta bilder i en sekvens av posisjoner (B). Innspilte bildeserier behandles for å lese ut fluorescensintensiteten per celle over tid (C). Skalastenger: 500 μm (A), 200 μm (C). Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
Her beskrev vi LISCA som en allsidig teknikk for å følge cellulær kinetikk av intracellulære fluorescerende etiketter på encellet nivå. For å utføre et vellykket LISCA-eksperiment må hvert av de beskrevne trinnene i protokolldelen etableres individuelt, og deretter må alle trinnene kombineres. Hver av de tre viktigste aspektene ved LISCA har viktige trinn.
Encellet mikroarray fabrikasjon
Kvaliteten på mikroarrayen er avgjørende, da den cellulære justeringen på mikroar…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dette arbeidet ble støttet av tilskudd fra Den tyske vitenskapsstiftelsen (DFG) til Collaborative Research Center (SFB) 1032. Støtte fra det tyske føderale departementet for utdanning, forskning og teknologi (BMBF) under samarbeidsprosjektet 05K2018-2017-06716 Medisoft samt et stipend fra Bayerische Forschungsstiftung er takknemlig anerkjent. Anita Reiser ble støttet av et DFG Fellowship gjennom Graduate School of Quantitative Biosciences Munich (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
References
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

