Identificatie van de bindende eiwitten van kleine liganden met de differentiële radiale capillaire werking van ligandtest (DRaCALA)
Summary
De differentiële radiale capillaire werking van Ligand Assay (DRaCALA) kan worden gebruikt om kleine ligandbindende eiwitten van een organisme te identificeren met behulp van een ORFeome-bibliotheek.
Abstract
Het afgelopen decennium heeft enorme vooruitgang geboekt in het begrijpen van kleine signaalmoleculen in de bacteriële fysiologie. Met name de doeleiwitten van verschillende nucleotide-afgeleide secundaire boodschappers (NSM’s) zijn systematisch geïdentificeerd en bestudeerd in modelorganismen. Deze prestaties zijn voornamelijk te danken aan de ontwikkeling van verschillende nieuwe technieken, waaronder de capture compound-techniek en de differentiële radiale capillaire werking van ligandtest (DRaCALA), die werden gebruikt om systematisch doeleiwitten van deze kleine moleculen te identificeren. Dit artikel beschrijft het gebruik van de NSM’s, guanosine penta- en tetrafosfaten (p)ppGpp, als voorbeeld en videodemonstratie van de DRaCALA-techniek. Met behulp van DRaCALA werden 9 van de 20 bekende en 12 nieuwe doeleiwitten van (p)ppGpp geïdentificeerd in het modelorganisme Escherichia coli K-12, wat de kracht van deze test aantoont. In principe kan DRaCALA worden gebruikt voor het bestuderen van kleine liganden die kunnen worden geëtiketteerd door radioactieve isotopen of fluorescerende kleurstoffen. De kritische stappen, voor- en nadelen van DRaCALA worden hier besproken voor verdere toepassing van deze techniek.
Introduction
Bacteriën gebruiken verschillende kleine signaalmoleculen om zich aan te passen aan constant veranderende omgevingen1,2. Bijvoorbeeld, de auto-inducers, N-acylhomoserine lactones en hun gemodificeerde oligopeptiden, bemiddelen de intercellulaire communicatie tussen bacteriën om populatiegedrag te coördineren, een fenomeen dat bekend staat als quorum sensing2. Een andere groep kleine signaalmoleculen zijn de NSM’s, waaronder het veel bestudeerde cyclische adenosinemonofosfaat (cAMP), cyclisch di-AMP, cyclisch di-guanosinemonofosfaat (cyclisch di-GMP) en guanosine penta- en tetrafosfaat (p)ppGpp1. Bacteriën produceren deze NSM’s als reactie op verschillende stressomstandigheden. Eenmaal geproduceerd, binden deze moleculen zich aan hun doeleiwitten en reguleren ze verschillende fysiologische en metabolische routes om de ondervonden stress aan te kunnen en de bacteriële overleving te verbeteren. Daarom is identificatie van de doeleiwitten een onvermijdelijke voorwaarde voor het ontcijferen van de moleculaire functies van deze kleine moleculen.
Het afgelopen decennium is getuige geweest van een hausse aan kennis van deze kleine signaalmoleculen, voornamelijk als gevolg van verschillende technische innovaties die de doeleiwitten van deze kleine moleculen onthulden. Deze omvatten de afvangverbindingstechniek3,4,5, en de differentiële radiale capillaire werking van ligandtest (DRaCALA)6 die in dit artikel moet worden besproken.
Uitgevonden door Vincent Lee en collega’s in 20116, DRaCALA zet het vermogen van een nitrocellulosemembraan in om differentieel vrije en eiwitgebonden liganden vast te zetten. Moleculen zoals eiwitten kunnen zich niet verspreiden op een nitrocellulosemembraan, terwijl kleine liganden, zoals de NSM’s, dat wel kunnen. Door het NSM (bijv.ppGpp ) te mengen met het te testen eiwit en deze op het membraan te spotten, kunnen twee scenario’s worden verwacht(figuur 1):Als (p)ppGpp zich aan het eiwit bindt, wordt het radiolabel (p)ppGpp in het midden van de vlek door het eiwit behouden en zal het niet naar buiten diffuus zijn, wat een intens klein puntje geeft (d.w.z. sterk radioactief signaal) onder een fosforimager. Als (p)ppGpp zich echter niet aan het eiwit bindt, zal het vrij naar buiten diffuus zijn om een grote vlek met een uniform radioactief achtergrondsignaal te produceren.
Bovendien kan DRaCALA de interactie tussen een klein molecuul en een ongezuiverd eiwit in een hele cellysaat detecteren als het eiwit in voldoende hoeveelheid aanwezig is. Deze eenvoud maakt het gebruik van DRaCALA mogelijk bij het snel identificeren van eiwitdoelen met behulp van een ORFeome-expressiebibliotheek. Doeleiwitten van cAMP7, cyclische di-AMP8, cyclische di-GMP9,10en (p)ppGpp 11,12,13 zijn systematisch geïdentificeerd met behulp van DRaCALA. Dit videoartikel gebruikt (p)ppGpp als voorbeeld om de kritische stappen en overwegingen bij het uitvoeren van een succesvolle DRaCALA-screening te demonstreren en te beschrijven. Een meer grondige beschrijving van DRaCALA14 wordt ten zeerste aanbevolen om te lezen in combinatie met dit artikel voordat U DRaCALA uitvoert.
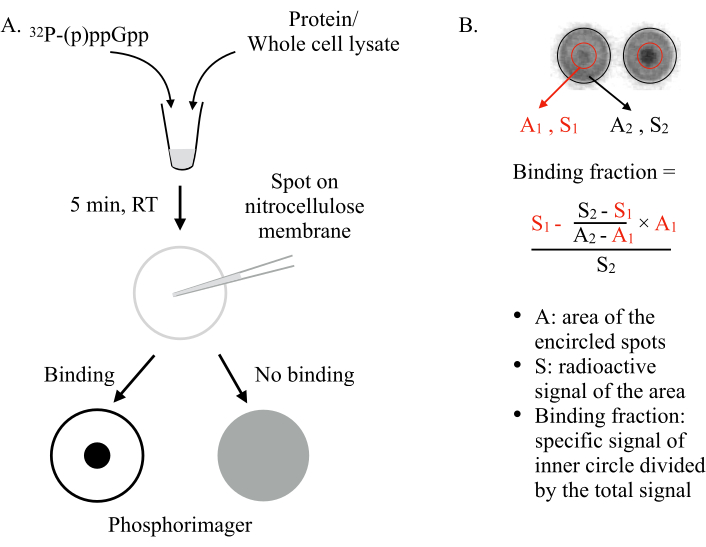
Figuur 1: Het principe van DRaCALA. (A) Schematisch van de DRaCALA-test. Zie de tekst voor meer informatie. B) Kwantificering en berekening van de bindende fractie. Zie de tekst voor meer informatie. Kortom, de DRaCALA-vlekken worden geanalyseerd door twee cirkels te tekenen die de hele vlek en de binnenste donkere stip omringen(d.w.z.de behouden (p)ppGpp vanwege de binding van het geteste eiwit). Het specifieke bindingssignaal is het radioactieve signaal van de binnenste cirkel (S1) na aftrek van het niet-specifieke achtergrondsignaal (berekend door A1 × ((S2-S1)/(A2-A1))). De bindingsfractie is het specifieke bindingssignaal gedeeld door het totale radioactieve signaal (S2). Afkortingen: DRaCALA = Differential Radial Capillary Action of Ligand Assay; (p)ppGpp = guanosine penta- en tetrafosfaten; RT = kamertemperatuur. Klik hier om een grotere versie van deze afbeelding te bekijken.
Protocol
Representative Results
Discussion
Een van de cruciale stappen bij het uitvoeren van DRaCALA-screening is het verkrijgen van goede hele cellysaten. Ten eerste moeten de geteste eiwitten in grote hoeveelheden en in oplosbare vormen worden geproduceerd. Ten tweede moet de lysis van cellen volledig zijn en moet de viscositeit van het lysaat minimaal zijn. De opname van lysozym en het gebruik van drie cycli van vries-dooi zijn vaak genoeg om cellen volledig te lyseren. Het vrijgegeven chromosomale DNA maakt het lysaat echter stroperig en genereert een hoog ac…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Het werk wordt ondersteund door een NNF-projectsubsidie (NNF19OC0058331) aan YEZ en het onderzoeks- en innovatieprogramma Horizon 2020 van de Europese Unie in het kader van de Marie Skłodowska-Curie-subsidieovereenkomst (Nº 801199) aan MLS.

Materials
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID – Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen – Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 – Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID – Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution – 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
References
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry–a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ‘,5 ‘-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).

