Identificazione delle proteine leganti di piccoli ligandi con l'azione capillare radiale differenziale del ligando Assay (DRaCALA)
Summary
Il Differential Radial Capillary Action of Ligand Assay (DRaCALA) può essere utilizzato per identificare piccole proteine leganti il ligando di un organismo utilizzando una libreria ORFeome.
Abstract
L’ultimo decennio ha visto enormi progressi nella comprensione di piccole molecole di segnalazione nella fisiologia batterica. In particolare, le proteine bersaglio di diversi messaggeri secondari derivati da nucleotidi (NSM) sono state sistematicamente identificate e studiate in organismi modello. Questi risultati sono dovuti principalmente allo sviluppo di diverse nuove tecniche tra cui la tecnica del composto di cattura e l’azione capillare radiale differenziale del saggio del ligando (DRaCALA), che sono state utilizzate per identificare sistematicamente le proteine bersaglio di queste piccole molecole. Questo documento descrive l’uso di NSM, guanosina penta- e tetrafosfati (p)ppGpp, come esempio e dimostrazione video della tecnica DRaCALA. Utilizzando DRaCALA, 9 su 20 note e 12 nuove proteine bersaglio di (p)ppGpp sono state identificate nell’organismo modello, Escherichia coli K-12, dimostrando la potenza di questo test. In linea di principio, DRaCALA potrebbe essere utilizzato per studiare piccoli ligandi che possono essere etichettati da isotopi radioattivi o coloranti fluorescenti. I passaggi critici, i pro e i contro di DRaCALA sono discussi qui per un’ulteriore applicazione di questa tecnica.
Introduction
I batteri utilizzano diverse piccole molecole di segnalazione per adattarsi ad ambienti in costante cambiamento1,2. Ad esempio, gli autoindoduttori, i lattoni N-acilomoserina e i loro oligopeptidi modificati, mediano la comunicazione intercellulare tra i batteri per coordinare il comportamento della popolazione, un fenomeno noto come quorum sensing2. Un altro gruppo di piccole molecole di segnalazione sono gli NSM, tra cui l’adenosina monofosfato ciclico (cAMP), il di-AMP ciclico, il monofosfato ciclico di di-guanosina (di-GMP ciclico) e i guanosina penta- e tetra fosfati (p)ppGpp1. I batteri producono questi NSM come risposta a una varietà di diverse condizioni di stress. Una volta prodotte, queste molecole si legano alle loro proteine bersaglio e regolano diverse vie fisiologiche e metaboliche per far fronte agli stress incontrati e migliorare la sopravvivenza batterica. Pertanto, l’identificazione delle proteine bersaglio è un prerequisito inevitabile per decifrare le funzioni molecolari di queste piccole molecole.
L’ultimo decennio ha visto un boom di conoscenza di queste piccole molecole di segnalazione, principalmente a causa di diverse innovazioni tecniche che hanno svelato le proteine bersaglio di queste piccole molecole. Questi includono la tecnica del composto di cattura3,4,5e l’azione capillare radiale differenziale del saggio del ligando (DRaCALA)6 da discutere in questo articolo.
Inventato da Vincent Lee e colleghi nel 20116,DRaCALA utilizza la capacità di una membrana nitrocellulosa di sequestrare in modo differenziale ligandi liberi e legati alle proteine. Molecole come le proteine non possono diffondersi su una membrana nitrocellulosa, mentre piccoli ligandi, come gli NSM, sono in grado di farlo. Mescolando l’NSM(ad esempio,ppGpp) con la proteina da testare e individuandoli sulla membrana, ci si possono aspettare due scenari (Figura 1): Se (p)ppGpp si lega alla proteina, il (p)ppGpp radiomarcato sarà trattenuto al centro dello spot dalla proteina e non si diffonderà verso l’esterno, dando un piccolo punto intenso (cioè., forte segnale radioattivo) sotto un fosforiger. Tuttavia, se (p)ppGpp non si lega alla proteina, si diffonderà liberamente verso l’esterno per produrre una grande macchia con segnale radioattivo di fondo uniforme.
Inoltre, DRaCALA può rilevare l’interazione tra una piccola molecola e una proteina nonpurata in un intero lisi cellulare se la proteina è presente in quantità sufficiente. Questa semplicità consente l’uso di DRaCALA nell’identificazione rapida di bersagli proteici utilizzando una libreria di espressioni ORFeome. Infatti, le proteine bersaglio di cAMP7, di-AMPciclico 8, ciclico di-GMP9,10e (p) ppGpp11,12,13 sono state sistematicamente identificate utilizzando DRaCALA. Questo articolo video utilizza (p)ppGpp come esempio per dimostrare e descrivere i passaggi critici e le considerazioni per eseguire uno screening DRaCALA di successo. Da notare, una descrizione più approfondita di DRaCALA14 è altamente raccomandata da leggere in combinazione con questo articolo prima di eseguire DRaCALA.
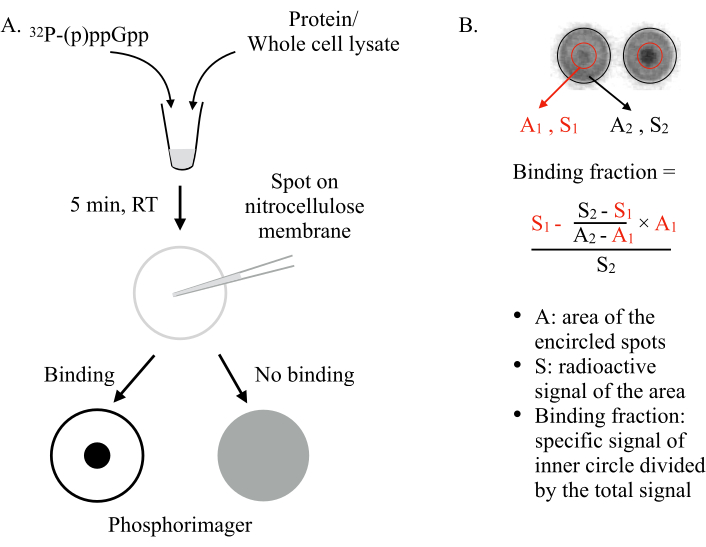
Figura 1: Il principio di DRaCALA. (A) Schema del saggio DRaCALA. Vedi il testo per i dettagli. (B) Quantificazione e calcolo della frazione vincolante. Vedi il testo per i dettagli. In breve, le macchie DRaCALA saranno analizzate disegnando due cerchi che circoscrivono l’intero punto e il punto scuro interno(cioèil (p)ppGpp trattenuto a causa del legame della proteina testata). Il segnale di legame specifico è il segnale radioattivo del cerchio interno (S1) dopo aver sottratto il segnale di fondo non specifico (calcolato da A1 × ((S2-S1)/(A2-A1))). La frazione di legame è il segnale di legame specifico diviso per il segnale radioattivo totale (S2). Abbreviazioni: DRaCALA = Differential Radial Capillary Action of Ligand Assay; (p)ppGpp = guanosina penta- e tetrafosfati; RT = temperatura ambiente. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Uno dei passaggi critici nell’esecuzione dello screening DRaCALA è quello di ottenere buoni lasati a cellule intere. In primo luogo, le proteine testate dovrebbero essere prodotte in grandi quantità e in forme solubili. In secondo luogo, la lisi delle cellule dovrebbe essere completa e la viscosità del lizzate deve essere minima. L’inclusione del lisozima e l’uso di tre cicli di congelamento-disgelo sono spesso sufficienti per far lisi completamente le cellule. Tuttavia, il DNA cromosomico rilasciato rende viscoso il …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Il lavoro è supportato da una sovvenzione del progetto NNF (NNF19OC0058331) a YEZ e dal programma di ricerca e innovazione Horizon 2020 dell’Unione europea nell’ambito della convenzione di sovvenzione Marie Skłodowska-Curie (Nº 801199) a MLS.

Materials
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID – Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen – Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 – Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID – Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution – 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
References
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry–a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ‘,5 ‘-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).

