Identifisere bindingsproteiner av små ligander med differensial radial kapillær virkning av Ligand Assay (DRaCALA)
Summary
Differensial radial kapillærvirkning av Ligand Assay (DRaCALA) kan brukes til å identifisere små ligandbindende proteiner av en organisme ved hjelp av et ORFeome-bibliotek.
Abstract
Det siste tiåret har det vært en enorm fremgang i forståelsen av små signalmolekyler i bakteriefysiologi. Spesielt er målproteinene til flere nukleotidavledede sekundærbud (NSMer) systematisk identifisert og studert i modellorganismer. Disse prestasjonene skyldes hovedsakelig utviklingen av flere nye teknikker, inkludert fangstsammensetningsteknikken og differensial radial kapillærvirkning av ligandanalyse (DRaCALA), som ble brukt til systematisk å identifisere målproteiner av disse små molekylene. Dette dokumentet beskriver bruken av NSMer, guanosin penta- og tetrafosfater (p)ppGpp, som et eksempel og en videodemonstrasjon av DRaCALA-teknikken. Ved hjelp av DRaCALA ble 9 av 20 kjente og 12 nye målproteiner av (p)ppGpp identifisert i modellorganismen, Escherichia coli K-12, som demonstrerer kraften i denne analysen. I prinsippet kan DRaCALA brukes til å studere små ligander som kan merkes av radioaktive isotoper eller fluorescerende fargestoffer. De kritiske trinnene, fordelene og ulempene ved DRaCALA diskuteres her for videre bruk av denne teknikken.
Introduction
Bakterier bruker flere små signalmolekyler for å tilpasse seg stadig skiftende miljøer1,2. For eksempel, autoindusere, N-acylhomoserine laktoser og deres modifiserte oligopeptider, formidler intercellulær kommunikasjon blant bakterier for å koordinere befolkningsadferd, et fenomen kjent som quorum sensing2. En annen gruppe små signalmolekyler er NSM-ene, inkludert det mye studerte sykliske adenosinmonofosfatet (cAMP), syklisk di-AMP, syklisk di-guanosinmonofosfat (syklisk di-GMP) og guanosin penta- og tetrafosfater (p)ppGpp1. Bakterier produserer disse NSM-ene som et svar på en rekke ulike stressforhold. Når de er produsert, binder disse molekylene seg til sine målproteiner og regulerer flere forskjellige fysiologiske og metabolske veier for å takle de påløpkede påkjenningene og forbedre bakteriell overlevelse. Derfor er identifisering av målproteinene en uunngåelig forutsetning for å dechiffrere molekylære funksjoner til disse små molekylene.
Det siste tiåret har vært vitne til en boom av kunnskap om disse små signalmolekylene, hovedsakelig på grunn av flere tekniske innovasjoner som avduket målproteinene til disse små molekylene. Disse inkluderer fangstsammensetningsteknikken3,4,5og differensial radial kapillærvirkning av ligandanalyse (DRaCALA)6 som skal diskuteres i dette dokumentet.
Oppfunnet av Vincent Lee og medarbeidere i 20116, DRaCALA distribuerer evnen til en nitrocellulosemembran til differensialt sequester frie og proteinbundne ligander. Molekyler som proteiner kan ikke spre seg på en nitrocellulosemembran, mens små ligander, som NSM-ene, er i stand til det. Ved å blande NSM (f.eks.ppGpp) med proteinet som skal testes og oppdage dem på membranen, kan to scenarier forventes (Figur 1): Hvis (p)ppGpp binder seg til proteinet, vil den radiomerkede (p)ppGpp beholdes i midten av stedet av proteinet og vil ikke spre seg utover, noe som gir en intens liten prikk (dvs. radioaktivt signal) under en fosforimager. Men hvis (p)ppGpp ikke binder seg til proteinet, vil det spre seg fritt utover for å produsere et stort sted med ensartet bakgrunn radioaktivt signal.
Videre kan DRaCALA oppdage samspillet mellom et lite molekyl og et uoppgjort protein i en hel cellelyse hvis proteinet er tilstede i tilstrekkelig mengde. Denne enkelheten gjør det mulig å bruke DRaCALA i raskt å identifisere proteinmål ved hjelp av et ORFeome-uttrykksbibliotek. Faktisk er målproteiner av cAMP7, syklisk di-AMP8, syklisk di-GMP9,10og (p) ppGpp11,12,13 systematisk identifisert ved hjelp av DRaCALA. Denne videoartikkelen bruker (p)ppGpp som et eksempel for å demonstrere og beskrive de kritiske trinnene og vurderingene ved å utføre en vellykket DRaCALA-screening. Vær oppmerksom på at en grundigere beskrivelse av DRaCALA14 anbefales på det sterkeste å lese i kombinasjon med denne artikkelen før du utfører DRaCALA.
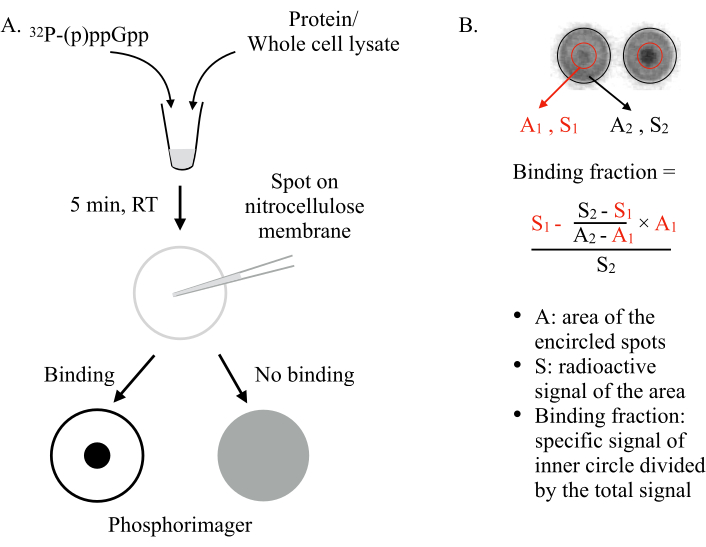
Figur 1: Prinsippet om DRaCALA. (A) Skjematisk for DRaCALA-analysen. Se teksten hvis du vil ha mer informasjon. (B) Kvantifisering og beregning av bindingsfraksjonen. Se teksten hvis du vil ha mer informasjon. Kort sagt vil DRaCALA-flekkene bli analysert ved å tegne to sirkler som omskriver hele stedet og den indre mørke prikken (dvs.beholdt (p) ppGpp på grunn av bindingen av det testede proteinet). Det spesifikke bindingssignalet er det radioaktive signalet til den indre sirkelen (S1) etter at det ikke-spesifikke bakgrunnssignalet er trukket fra (beregnet med A1 × ((S2-S1)/(A2-A1))). Bindingsfraksjonen er det spesifikke bindingssignalet delt på det totale radioaktive signalet (S2). Forkortelser: DRaCALA = Differensial radial kapillær virkning av Ligand Assay; (p)ppGpp = guanosin penta- og tetrafosfater; RT = romtemperatur. Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
Et av de kritiske trinnene for å utføre DRaCALA-screening er å oppnå gode hele cellelys. For det første bør de testede proteinene produseres i store mengder og i oppløselige former. For det andre bør lysis av celler være komplett, og lysatets viskositet må være minimal. Inkluderingen av lysozyme og bruk av tre sykluser med fryse-tining er ofte nok til å lyse celler helt. Imidlertid gjør det frigjorte kromosomale DNA lysatet viskøs og genererer høy bakgrunnsbindingssignal, noe som resulterer i falske positi…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Arbeidet støttes av et NNF Project Grant (NNF19OC0058331) til YEZ, og EUs forsknings- og innovasjonsprogram Horizon 2020 under Marie Skłodowska-Curie-tilskuddsavtalen (Nº 801199) til MLS.

Materials
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID – Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen – Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 – Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID – Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution – 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
References
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry–a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ‘,5 ‘-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).

