DNA张力探针用于绘制免疫细胞的瞬时皮牛顿受体力
Summary
本文描述了使用基于DNA的张力探针对免疫细胞施加的受体力进行成像的详细方案。这种方法可以实时映射受体力>4.7pN,并且可以随着时间的推移积分力。
Abstract
在两个相邻细胞之间的连接处以及细胞与细胞外基质之间的连接处传递的机械力对于调节从发育到免疫学的许多过程至关重要。因此,开发在分子尺度上研究这些力的工具至关重要。我们小组开发了一套分子张力传感器,用于量化和可视化细胞产生并传递到特定配体的力。最灵敏的一类分子张力传感器由核酸茎环发夹组成。这些传感器使用荧光团-淬灭剂对来报告DNA发夹在力下的机械延伸和展开。DNA发夹张力传感器的一个挑战是,它们是可逆的,在张力终止时快速重新折叠发夹,因此很难记录瞬态力。在本文中,我们描述了制备DNA张力传感器的协议,这些传感器可以“锁定”并防止重新折叠,以实现机械信息的“存储”。这允许记录高瞬态微微顿力,随后可以通过添加去除锁的互补核酸来“擦除”。这种在实时张力映射和机械信息存储之间切换的能力揭示了弱的、短暂的和不太丰富的力,这些力通常被T细胞用作其免疫功能的一部分。
Introduction
免疫细胞通过不断爬行和扫描靶细胞表面的抗原来防御病原体和癌细胞,并在其表面镶嵌1,2。抗原识别在T细胞受体(TCR)和靶细胞表面表达的肽主要组织相容性复合物MHC(pMHC)复合物结合时开始。由于TCR-pMHC识别发生在两个移动细胞之间的连接处,因此长期以来一直怀疑它经历了机械力。此外,这导致了TCR激活的机械传感器模型,这表明TCR力有助于其功能3,4。为了了解机械力何时、何地以及如何促进T细胞功能,必须开发工具来可视化T细胞传递的分子力。传统上,牵引力显微镜(TFM)和微柱阵列等方法用于研究细胞力5,6。然而,全聚焦方式(TFM)和微柱阵列的力灵敏度为纳牛顿(nN)尺度,因此通常不足以研究细胞受体传递的分子微孔(pN)力7。为了提高检测的力和空间分辨率,我们的实验室率先开发了分子张力探头,最初是使用聚乙二醇(PEG)聚合物7合成的。分子张力探针由可伸缩的分子“弹簧”(PEG、蛋白质、DNA)组成,两侧是荧光基团和淬灭剂,并固定在表面上。施加在探针末端的力导致其延伸,分离荧光团和淬灭剂,从而产生强烈的荧光信号(图1A)8,9,10。
在过去的十年中,我们开发了一个包含不同类别分子张力探针的库,其中包含由核酸11、蛋白质10 和聚合物8 制成的弹簧元件。其中,基于DNA的张力探头提供最高的信噪比和最大的力灵敏度,可在几pN到~20 pN11之间轻松调谐。我们已经使用这些实时DNA张力探针来研究许多不同细胞类型产生的分子力,包括成纤维细胞,癌细胞,血小板和免疫细胞11,12,13。本手稿将描述在表面上合成和组装DNA张力探针的方案,以使用常规荧光显微镜以pN力分辨率绘制分子受体力。虽然目前的程序包括对核酸进行化学修饰以引入荧光报告基因(图1B),但重要的是要注意,许多修饰和纯化步骤可以外包给定制DNA合成公司。因此,DNA张力探针技术是容易的,并且可以被更广泛的细胞生物学和机械生物学界所使用。
简而言之,为了组装DNA张力传感器,将DNA发夹杂交到一个臂上的荧光配体链和另一个臂上的淬灭锚链,然后固定在玻璃基板上(图1C,实时张力)。在没有机械力的情况下,发夹闭合,因此荧光被淬灭。然而,当施加的机械力大于F1/2 (平衡时的力导致50%的展开概率)时,发夹机械熔化,并产生荧光信号。
在实时DNA张力传感器的基础上,我们还描述了映射累积力的协议,这对于研究免疫细胞受体与其天然配体之间的相互作用特别有用。这是因为免疫受体通常显示短寿命的键3,14。使用“锁定”链对累积的力进行成像,该链优先结合以打开DNA发夹并允许存储与机械拉动事件相关的荧光信号(图1C,锁定张力)。锁定链旨在结合在机械诱导的发夹熔化时暴露的隐形结合位点,并通过阻止发夹重新折叠将发夹锁定在打开状态,从而存储张力信号并生成累积的张力图。此外,锁定链设计有八核苷酸链,这使得脚趾介导的链位移反应及其完整的互补,即“解锁”链。通过添加解锁链,绑定的锁定链从发夹结构上剥离,擦除存储的张力信号并将发夹重置回实时状态。
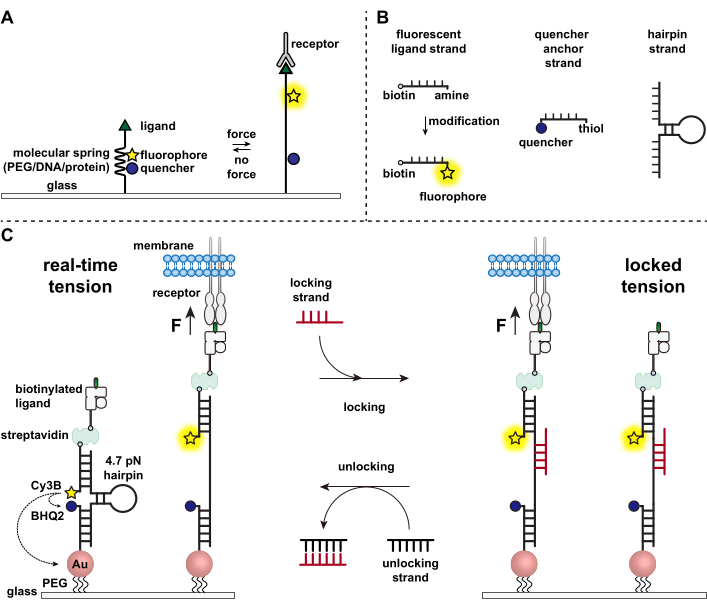
图 1:最先进的分子张力探头方案 。 (A)实时分子张力探针的一般设计,(B)基于DNA的张力探针构建的链,以及(C)工程化的基于DNA的张力探针及其在实时状态和锁定状态之间的切换。 请点击此处查看此图的大图。
主要方案由四个主要部分组成 – 寡核苷酸制备,表面处理,成像和数据分析。我们的实验室和其他实验室已经在幼稚和活化的OT-1 CD8 + T细胞,OT-II CD4 +细胞以及杂交瘤中成功证明了该协议,并且可以应用于询问不同的免疫细胞受体,包括T细胞受体,程序性细胞死亡受体(PD1)和淋巴细胞功能相关抗原1(LFA-1)力。本文将OT-1 CD8+幼稚T细胞用作示例细胞系。
Protocol
Representative Results
Discussion
通过此处提供的详细程序,可以制备DNA发夹张力探针底物,以绘制和量化免疫细胞产生的受体张力。当细胞被接种到DNA发夹张力探针底物上时,当受体以化学和机械方式感知配体时,它们会着陆,附着和扩散,后者由我们的探针检测到。然而,在某些情况下,细胞可能无法扩散(图7A)或无法产生张力信号。这通常是表面化学问题的结果,需要排除故障(表1).低…
Disclosures
The authors have nothing to disclose.
Acknowledgements
这项工作得到了NIH Grants R01GM131099,NIH R01GM124472和NSF CAREER 1350829的支持。我们感谢NIH四聚体设施的pMHC配体。这项研究得到了埃默里综合糖组学核心的部分支持。
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
References
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

