Tools voor de real-time beoordeling van een Pseudomonas aeruginosa-infectiemodel
Summary
Synthetisch cystic fibrosis sputum medium (SCFM2) kan worden gebruikt in combinatie met zowel confocale laserscanmicroscopie als fluorescentie-geactiveerde celsortering om bacteriële aggregaten met hoge resolutie te observeren. Dit artikel beschrijft methoden om geaggregeerde populaties te beoordelen tijdens antimicrobiële behandeling als een platform voor toekomstige studies.
Abstract
Pseudomonas aeruginosa (Pa) is een van de meest voorkomende opportunistische pathogenen geassocieerd met cystische fibrose (CF). Zodra Pa-kolonisatie is vastgesteld, vormt een groot deel van de infecterende bacteriën biofilms in het sputum van de luchtwegen. Van pa-biofilms geïsoleerd uit CF-sputum is aangetoond dat ze groeien in kleine, dichte aggregaten van ~ 10-1.000 cellen die ruimtelijk georganiseerd zijn en klinisch relevante fenotypen vertonen, zoals antimicrobiële tolerantie. Een van de grootste uitdagingen bij het bestuderen van hoe Pa-aggregaten reageren op de veranderende sputumomgeving is het gebrek aan nutritioneel relevante en robuuste systemen die de vorming van aggregaten bevorderen. Met behulp van een synthetisch CF-sputummedium (SCFM2) kan de levensgeschiedenis van Pa-aggregaten worden waargenomen met behulp van confocale laserscanmicroscopie (CLSM) en beeldanalyse met de resolutie van een enkele cel. Dit in vitro systeem maakt de observatie van duizenden aggregaten van verschillende grootte in real time, drie dimensies en op micronschaal mogelijk. Op individueel en populatieniveau vergemakkelijkt het vermogen om aggregaten te groeperen op fenotype en positie de observatie van aggregaten in verschillende ontwikkelingsstadia en hun reactie op veranderingen in de micro-omgeving, zoals antibioticabehandeling, die met precisie moeten worden gedifferentieerd.
Introduction
Pseudomonas aeruginosa (Pa) is een opportunistische ziekteverwekker die chronische infecties vaststelt bij immuungecompromitteerde personen. Voor mensen met de genetische ziekte cystic fibrosis (CF), kunnen deze infecties de loop van een leven duren. CF veroorzaakt de opbouw van een stroperig, voedingsrijk sputum in de luchtwegen, dat in de loop van de tijd wordt gekoloniseerd door een verscheidenheid aan microbiële pathogenen. Pa is een van de meest voorkomende CF-pathogenen, koloniseert de luchtwegen in de vroege kinderjaren en stelt moeilijk te behandelen infecties vast1. Pa blijft een belangrijk klinisch probleem en wordt beschouwd als een belangrijke doodsoorzaak bij mensen met CF, ondanks verbeterde therapieregimes in de afgelopen jaren2,3. Dit persistentiefenotype en toenemende antibioticatolerantie hebben Pa een plaats opgeleverd in een groep pathogenen die door zowel de Centers for Disease Control (CDC) als de Wereldgezondheidsorganisatie (WHO) zijn geïdentificeerd als onderzoeksprioriteiten voor de ontwikkeling van nieuwe therapeutische strategieën – de ESKAPE-pathogenen4.
Net als andere ESKAPE-pathogenen komt verworven antibioticaresistentie vaak voor bij Pa, maar er zijn ook veel intrinsieke eigenschappen die bijdragen aan de antimicrobiële tolerantie van Pa. Onder deze is het vermogen van Pa om aggregaten-zeer dichte clusters van ~ 10-1.000 cellen te vormen, die kunnen worden waargenomen bij meerdere infecties, waaronder CF-patiënt sputum5,6. Vergelijkbaar met Pa bestudeerd in andere biofilmsystemen, vertonen Pa-aggregaten klinisch relevante fenotypen zoals verhoogde resistentie tegen antibiotica en activering van cel-celcommunicatie (quorum sensing (QS)). Van aggregaten van Pa is bijvoorbeeld aangetoond dat ze QS-gereguleerd gedrag gebruiken om andere microben te bestrijden en antimicrobiële behandelingen zoals de productie van pyocyanine te verdragen7. Het vermogen om dergelijk gedrag te bestuderen biedt een opwindend inzicht in bacteriële ecosystemen in een omgeving die vergelijkbaar is met die waarin ze in het menselijk lichaam bestaan.
Een van de grootste uitdagingen bij het bestuderen van hoe Pa-aggregaten reageren op de veranderende sputumomgeving is het gebrek aan nutritioneel relevante en robuuste systemen die de vorming van aggregaten bevorderen. Veel van wat bekend is over Pa is ontdekt met behulp van in vitro systemen waarin cellen planktonisch groeien of in een karakteristieke aan het oppervlak gehechte, “paddenstoel” -architectuur die niet in vivo is waargenomen 8. Hoewel klassieke biofilmgroeimodellen, zoals stromingscellen of vaste agar, uitgebreide en waardevolle kennis hebben opgeleverd over bacterieel gedrag en mechanismen van antibioticatolerantie, vertalen deze bevindingen zich niet altijd in vivo. Veel in vitro modellen hebben een beperkt vermogen om de groeiomgeving van de menselijke infectieplaats na te bootsen, waardoor kostbare in vivo studies nodig zijn. Op hun beurt missen veel in vivo modellen de flexibiliteit en resolutie die in vitro technieken bieden.
Synthetische cystic fibrosis sputum (SCFM2) is ontworpen om een omgeving voor Pa-groei te bieden die vergelijkbaar is met die ervaren tijdens chronische infectie in de CF-long. SCFM2 omvat voedingsbronnen geïdentificeerd in slijmolijmoste CF sputa naast mucine, lipiden en DNA. Pa-groei in SCFM2 vereist een bijna identiek gen dat is ingesteld als dat nodig is voor groei in feitelijk sputum en ondersteunt natuurlijke Pa-aggregaatvorming 9,10. Na inenting vormen planktonische cellen aggregaten die in omvang toenemen door expansie. Individuele cellen (aangeduid als migranten) worden vrijgegeven uit aggregaten, migreren naar niet-gecoloniseerde gebieden en vormen nieuwe aggregaten10. Deze levensgeschiedenis kan worden waargenomen met behulp van CLSM en beeldanalyse met de resolutie van een enkele cel. Aggregaten van Pa gevormd in SCFM2 zijn van vergelijkbare grootte als die waargenomen in de CF-long10. Dit model maakt het mogelijk om meerdere aggregaten van verschillende grootte in real-time en in drie dimensies op micronschaal te observeren. Time-lapse microscopie maakt het mogelijk om duizenden (~ 50.000) aggregaten in één experiment te volgen. Het gebruik van beeldanalysesoftware maakt het mogelijk om geaggregeerde fenotypen uit micrografieën te kwantificeren, inclusief geaggregeerd volume, oppervlakte en positie in drie dimensies tot op 0,1 μm, zowel op individueel geaggregeerd als populatieniveau. Het vermogen om aggregaten te groeperen op fenotype en positie maakt de differentiatie van aggregaten in verschillende ontwikkelingsstadia met precisie mogelijk, evenals hun reactie op een veranderende micro-omgeving6,11.
De toepassing van SCFM2 om Pa-aggregaten te bestuderen in lage volumes en high-throughput assays maken het een flexibel, kosteneffectief model. Als gedefinieerd medium biedt SCFM2 uniformiteit en reproduceerbaarheid op meerdere platforms, waardoor een nutritioneel en fysiek relevante methode wordt geboden om Pa-aggregaten in vitro te bestuderen 9. Toepassingen omvatten het gebruik ervan in combinatie met CLSM om ruimtelijke organisatie en antibioticatolerantie bij hoge resolutie te observeren (zoals beschreven in dit methodendocument). De mogelijkheid om experimenten uit te voeren die real-time gegevens op micronschaal opleveren, maakt de studie van intra-species en inter-species interacties mogelijk, omdat ze in vivokunnen voorkomen. SCFM2 is bijvoorbeeld eerder gebruikt om de ruimtelijke dynamiek van cel-celcommunicatie in geaggregeerde populaties te bestuderen via een netwerk van systemen die door Pa worden gebruikt om meerdere genen te reguleren die bijdragen aan virulentie en pathogenese6.
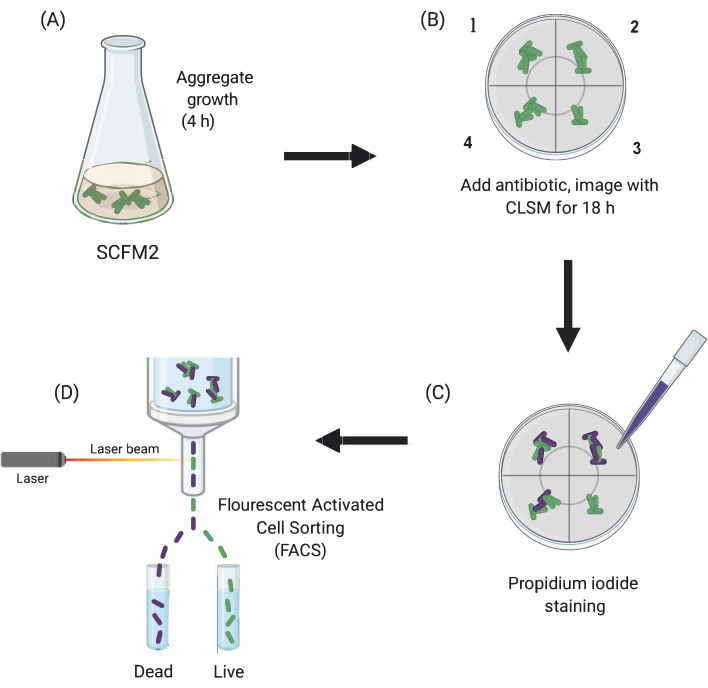
Figuur 1: Grafische weergave van de belangrijkste experimentele stappen. (A) SCFM2 wordt ingeënt met Pa-cellen en mag aggregaten vormen in een kweekschaal met glazen bodem. (B) Aggregaten worden overgebracht naar de confocale microscoop en antibioticum wordt toegevoegd. Afgebeeld zijn drie technische replicaties (kamers 1-3) en een controleput (4) van geënt SCFM2 zonder antibioticabehandeling. Aggregaten worden afgebeeld met behulp van CLSM in de loop van 18 h. (C) Na de eerste 18-h beeldvorming worden aggregaten behandeld met propidiumjodide om dode cellen te visualiseren en afgebeeld met CLSM (D) Aggregaten met het gewenste fenotype worden gescheiden van SCFM2 met behulp van FACS. Afkortingen: SCFM2 = synthetische cystic fibrosis sputum medium; Pa = Pseudomonas aeruginosa; CLSM = confocale laser scanning microscopie; FACS = fluorescentie-geactiveerde celsortering. Klik hier om een grotere versie van deze figuur te bekijken.
Hier wordt het nut van SCFM2 aangetoond om de impact van antibioticabehandeling op Pa-aggregaten in realtime te bestuderen, gevolgd door het gebruik van een celsorteerbenadering om populaties van aggregaten met verschillende fenotypen te isoleren voor downstream-analyse (figuur 1).
Protocol
Representative Results
Discussion
Dit werk heeft methodologieën geïntroduceerd die kunnen worden gecombineerd om bacteriële geaggregeerde populaties te bestuderen in de aan- en afwezigheid van antibioticabehandeling. Clsm met hoge resolutie maakt de visualisatie mogelijk van veranderingen in geaggregeerde biomassa en de structurele oriëntatie van aggregaten in realtime bij blootstelling aan antibiotica. Bovendien kunnen fysieke en structurele kenmerken van de biomassa die overblijven na behandeling met antibiotica worden gekwantificeerd, met als doel…
Disclosures
The authors have nothing to disclose.
Acknowledgements
S.E.D wordt ondersteund door start-upfondsen van de afdeling Moleculaire Geneeskunde, de Universiteit van Zuid-Florida, evenals een CFF-onderzoeksbeurs (DARCH19G0), de N.I.H (5R21AI147654 – 02 (PI, Chen)) en het USF Institute on Microbiomes. We bedanken het Whiteley-lab voor de voortdurende samenwerking met betrekking tot datasets met betrekking tot dit manuscript. Wij danken Dr. Charles Szekeres voor het faciliteren van FACS sortering. Figuren werden gemaakt door A.D.G en S.E.D met behulp van Biorender.com.
Materials
| Amino acids | |||
| Alanine | Acr s Organics s Organics |
56-41-7 | |
| Arginine HCl | MP | 1119-34-2 | |
| Asparagine | Acr s Organics s Organics |
56-84-8 | Prepared in 0.5 M NaOH |
| Cystine HCl | Alfa Aesar | L06328 | |
| Glutamic acid HCl | Acr s Organics s Organics |
138-15-8 | |
| Glycine | Acr s Organics s Organics |
56-40-6 | |
| Histidine HCl H2O | Alfa Aesar | A17627 | |
| Isoleucine | Acr s Organics s Organics |
73-32-5 | |
| Leucine | Alfa Aesar | A12311 | |
| Lysine HCl | Alfa Aesar | J62099 | |
| Methionine | Acr s Organics s Organics |
63-68-3 | |
| Ornithine HCl | Alfa Aesar | A12111 | |
| Phenylalanine | Acr s Organics s Organics |
63-91-2 | |
| Proline | Alfa Aesar | A10199 | |
| Serine | Alfa Aesar | A11179 | |
| Threonine | Acr s Organics s Organics |
72-19-5 | |
| Tryptophan | Acr s Organics s Organics |
73-22-3 | Prepared in 0.2 M NaOH |
| Tyrosine | Alfa Aesar | A11141 | Prepared in 1.0 M NaOH |
| Valine | Acr s Organics s Organics |
72-18-4 | |
| Antibiotic | |||
| Carbenicillin | Alfa Aesar | J6194903 | |
| Day-of Stocks | |||
| CaCl2 * 2H2O | Fisher Chemical | C79-500 | |
| Dextrose (D-glucose) | Fisher Chemical | 50-99-7 | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Fisher (Avanti Polar Lipids) | 4235-95-4 | shake 15-20 min at 37 °C to evaporate chloroform |
| FeSO4 * 7H2O | Acr s Organics s Organics |
7782-63-0 | this stock equals 1 mg/mL, MUST make fresh |
| L-lactic acid | Alfa Aesar | L13242 | pH stock to 7 with NaOH |
| MgCl2 * 6H2O | Acr s Organics s Organics |
7791-18-6 | |
| N-acetylglucosamine | TCI | A0092 | |
| Prepared solids | |||
| Porcine mucin | Sigma | M1778-100G | UV-sterilize |
| Salmon sperm DNA | Invitrogen | 15632-011 | |
| Stain | |||
| Propidium iodide | Alfa Aesar | J66764MC | |
| Salts | |||
| K2SO4 | Alfa Aesar | A13975 | |
| KCl | Alfa Aesar | J64189 | add solid directly to buffered base |
| KNO3 | Acr s Organics s Organics |
7757-79-1 | |
| MOPS | Alfa Aesar | A12914 | add solid directly to buffered base |
| NaCl | Fisher Chemical | S271-500 | add solid directly to buffered base |
| Na2HPO4 | RPI | S23100-500.0 | |
| NaH2PO4 | RPI | S23120-500.0 | |
| NH4Cl | Acr s Organics s Organics |
12125-02-9 | add solid directly to buffered base |
| Consumables | |||
| Conical tubes (15 mL) | Olympus plastics | 28-101 | |
| Conical tubes (50 mL) | Olympus plastics | 28-106 | |
| Culture tubes w/air flow cap | Olympus plastics | 21-129 | |
| 35 mm four chamber glass-bottom dish | CellVis | NC0600518 | |
| Luria Bertani (LB) broth | Genessee Scientific | 11-118 | |
| Phosphate-buffered saline (PBS) | Fisher Bioreagents | BP2944100 | |
| Pipet tips (p200) | Olympus plastics | 23-150RL | |
| Pipet tips (p1000) | Olympus plastics | 23-165RL | |
| Serological pipets (5 mL) | Olympus plastics | 12-102 | |
| Serological pipets (25 mL) | Olympus plastics | 12-106 | |
| Serological pipets (50 mL) | Olympus plastics | 12-107 | |
| Ultrapure water (RNAse/DNAse free); nanopure water | Genessee Scientific | 18-194 | Nanopure water used for preparation of solutions in Table 1 |
| Syringes (10 mL) | BD | 794412 | |
| Syringes (50 mL) | BD | 309653 | |
| 0.22 mm PES syringe filter | Olympus plastics | 25-244 | |
| PS cuvette semi-mico | Olympus plastics | 91-408 | |
| Software | |||
| Biorender | To prepare the figures | ||
| FacsDiva6.1.3 | Becton Dickinson, San Jose, CA | ||
| Imaris | Bitplane | version 9.6 | |
| Zen Black | |||
| Equipment | |||
| FacsAriallu | Becton Dickinson, San Jose, CA | ||
| LSM 880 confocal laser scanning microscope | Zeiss |
References
- Ramsay, K. A., et al. The changing prevalence of pulmonary infection in with fibrosis: A longitudinal analysis. Journal of Cystic Fibrosis. 16 (1), 70-77 (2017).
- Bessonova, L., et al. Data from the US and UK cystic fibrosis registries support disease modification by CFTR modulation with ivacaftor. Thorax. 73 (8), 731-740 (2018).
- Breuer, O., et al. Changing prevalence of lower airway infections in young children with cystic fibrosis. American Journal of Respiratory and Critical Care Medicine. 200 (5), 590-599 (2019).
- O’Donnell, J. N., Bidell, M. R., Lodise, T. P. Approach to the treatment of patients with serious multidrug-resistant Pseudomonas aeruginosa infections. Pharmacotherapy. 40 (9), 952-969 (2020).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Darch, S. E., et al. Spatial determinants of quorum signaling in a Pseudomonas aeruginosa infection model. Proceedings of the National Academy of Sciences of the United States of America. 115 (18), 4779-4784 (2018).
- Zhu, K., Chen, S., Sysoeva, T. A., You, L. Universal antibiotic tolerance arising from antibiotic-triggered accumulation of pyocyanin in Pseudomonas aeruginosa. PLoS Biology. 17 (12), 3000573 (2019).
- Ciofu, O., Tolker-Nielsen, T. Tolerance and resistance of Pseudomonas aeruginosa biofilms to antimicrobial agents-how P. aeruginosa can escape antibiotics. Frontiers in Microbiology. 10, 913 (2019).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proceedings of the National Academy of Sciences of the United States of America. 112 (13), 4110-4115 (2015).
- Darch, S. E., et al. Phage inhibit pathogen dissemination by targeting bacterial migrants in a chronic infection model. MBio. 8 (2), 00240 (2017).
- Jorth, P., et al. Regional isolation drives bacterial diversification within cystic fibrosis lungs. Cell Host & Microbe. 18 (3), 307-319 (2015).
- Palmer, K. L., Aye, L. M., Whiteley, M. Nutritional cues control Pseudomonas aeruginosa multicellular behavior in cystic fibrosis sputum. Journal of Bacteriology. 189 (22), 8079-8087 (2007).
- Davies, D. G., et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science. 280 (5361), 295-298 (1998).
- Hartmann, R., et al. Quantitative image analysis of microbial communities with BiofilmQ. Nature Microbiology. 6 (2), 151-156 (2021).
- Stacy, A., et al. Bacterial fight-and-flight responses enhance virulence in a polymicrobial infection. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7819-7824 (2014).

