Verktøy for sanntidsvurdering av en Pseudomonas aeruginosa infeksjonsmodell
Summary
Syntetisk cystisk fibrose sputum medium (SCFM2) kan brukes i kombinasjon med både konfokal laser skanning mikroskopi og fluorescens-aktivert celle sortering for å observere bakterielle aggregater ved høy oppløsning. Denne artikkelen beskriver metoder for å vurdere aggregerte populasjoner under antimikrobiell behandling som en plattform for fremtidige studier.
Abstract
Pseudomonas aeruginosa (Pa) er et av de vanligste opportunistiske patogenene forbundet med cystisk fibrose (CF). Når Pa-koloniseringen er etablert, danner en stor del av de smittende bakteriene biofilmer i luftveissputum. Pa biofilmer isolert fra CF sputum har vist seg å vokse i små, tette aggregater på ~ 10-1000 celler som er romlig organisert og viser klinisk relevante fenotyper som antimikrobiell toleranse. En av de største utfordringene med å studere hvordan Pa aggregater reagerer på det skiftende sputummiljøet, er mangelen på ernæringsmessig relevante og robuste systemer som fremmer aggregert dannelse. Ved hjelp av et syntetisk CF-sputummedium (SCFM2) kan livshistorien til Pa-aggregater observeres ved hjelp av konfokal laserskanningsmikroskopi (CLSM) og bildeanalyse ved oppløsningen til en enkelt celle. Dette in vitro-systemet tillater observasjon av tusenvis av aggregater av varierende størrelse i sanntid, tre dimensjoner og i mikronskalaen. På individ- og befolkningsnivå legger det å ha evnen til å gruppere aggregater etter fenotype og posisjon observasjon av aggregater på ulike utviklingsstadier og deres respons på endringer i mikromiljøet, som antibiotikabehandling, å bli differensiert med presisjon.
Introduction
Pseudomonas aeruginosa (Pa) er et opportunistisk patogen som etablerer kroniske infeksjoner hos immun-kompromitterte individer. For de med den genetiske sykdommen cystisk fibrose (CF), kan disse infeksjonene strekke seg over livets løpet. CF forårsaker oppbygging av et viskøs, næringsrikt sputum i luftveiene, som blir kolonisert av en rekke mikrobielle patogener over tid. Pa er en av de mest utbredte CF patogener, koloniserer luftveiene i tidlig barndom og etablerer vanskelige å behandle infeksjoner1. Pa er fortsatt et betydelig klinisk problem og regnes som en ledende årsak til dødelighet hos de med CF, til tross for forbedrede terapiregimer de siste årene2,3. Denne utholdenhet fenotypen og økende antibiotikatoleranse har gitt Pa en plass i en gruppe patogener identifisert av både Centers for Disease Control (CDC) og Verdens helseorganisasjon (WHO) som forskningsprioriteringer for utvikling av nye terapeutiske strategier – ESKAPE patogener4.
Som andre ESKAPE-patogener er er ervervet antibiotikaresistens vanlig hos Pa, men det er også mange iboende egenskaper som bidrar til Pa antimikrobiell toleranse. Blant disse er Pas evne til å danne aggregater-svært tette klynger på ~ 10-1000 celler, som kan observeres ved flere infeksjoner, inkludert CF pasient sputum5,6. I likhet med Pa studert i andre biofilmsystemer, viser Pa aggregater klinisk relevante fenotyper som økt resistens mot antibiotika og aktivering av cellecellekommunikasjon (quorumssensor (QS)). For eksempel har aggregater av Pa vist seg å bruke QS-regulert oppførsel for å bekjempe andre mikrober, samt tolerere antimikrobielle behandlinger som produksjon av pyocyanin7. Evnen til å studere slik atferd gir et spennende innblikk i bakterielle økosystemer i et miljø som ligner det de eksisterer i menneskekroppen.
En av de største utfordringene med å studere hvordan Pa aggregater reagerer på det skiftende sputummiljøet, er mangelen på ernæringsmessig relevante og robuste systemer som fremmer aggregert dannelse. Mye av det som er kjent om Pa har blitt oppdaget ved hjelp av in vitro-systemer der celler vokser planktonisk eller i en karakteristisk overflatemontert, “sopp” arkitektur som ikke har blitt observert in vivo8. Mens klassiske biofilmvekstmodeller, som strømningsceller eller fast agar, har gitt omfattende og verdifull kunnskap om bakteriell atferd og mekanismer for antibiotikatoleranse, oversetter disse funnene ikke alltid in vivo. Mange in vitro-modeller har en begrenset evne til å etterligne vekstmiljøet på det menneskelige infeksjonsstedet, noe som krever kostbare in vivo-studier. I sin tur mangler mange in vivo-modeller fleksibiliteten og oppløsningen som gis av in vitro-teknikker.
Syntetisk cystisk fibrose sputum (SCFM2) er designet for å gi et miljø for Pa vekst som ligner det som oppleves under kronisk infeksjon i CF-lungen. SCFM2 inkluderer ernæringskilder identifisert i ekspektorert CF-sputa i tillegg til mucin, lipider og DNA. Pa vekst i SCFM2 krever et nesten identisk gen satt til det som kreves for vekst i faktisk sputum og støtter naturlig Pa aggregertformasjon 9,10. Etter inokulering danner planktoniske celler aggregater som øker i størrelse gjennom ekspansjon. Enkeltceller (referert til som migranter) frigjøres fra aggregater, migrerer til ukoloniserte områder og danner nye aggregater10. Denne livshistorikken kan observeres ved hjelp av CLSM og bildeanalyse ved oppløsningen til en enkelt celle. Aggregater av Pa dannet i SCFM2 er av lignende størrelser som de som er observert i CF lunge10. Denne modellen tillater observasjon av flere aggregater av varierende størrelse i sanntid og i tre dimensjoner på mikronskalaen. Intervallmikroskopi gjør det mulig å spore tusenvis (~50 000) aggregater i ett eksperiment. Bruken av bildeanalyseprogramvare tillater kvantifisering av aggregerte fenotyper fra mikrografer, inkludert aggregert volum, overflateareal og posisjon i tre dimensjoner til nærmeste 0,1 μm, både på individnivå og befolkningsnivå. Å ha evnen til å gruppere aggregater etter fenotype og posisjon tillater differensiering av aggregater på forskjellige utviklingsstadier med presisjon, samt deres respons på et skiftende mikromiljø6,11.
Anvendelsen av SCFM2 for å studere Pa-aggregater i lavvolum- og høygjennomstrømningsanalyser gjør den til en fleksibel, kostnadseffektiv modell. Som et definert medium tilbyr SCFM2 ensartethet og reproduserbarhet på tvers av flere plattformer, og gir en ernæringsmessig og fysisk relevant metode for å studere Pa aggregater in vitro9. Anvendelser inkluderer bruk i kombinasjon med CLSM for å observere romlig organisering og antibiotikatoleranse ved høy oppløsning (som beskrevet i dette metodepapiret). Evnen til å utføre eksperimenter som gir sanntidsdata i mikronskala, gjør det mulig å studere intraarter og inter-arts interaksjoner som de kan forekomme in vivo. For eksempel har SCFM2 tidligere blitt brukt til å studere den romlige dynamikken i cellecellekommunikasjon i samlede populasjoner via et nettverk av systemer som brukes av Pa for å regulere flere gener som bidrar til virulens og patogenese6.
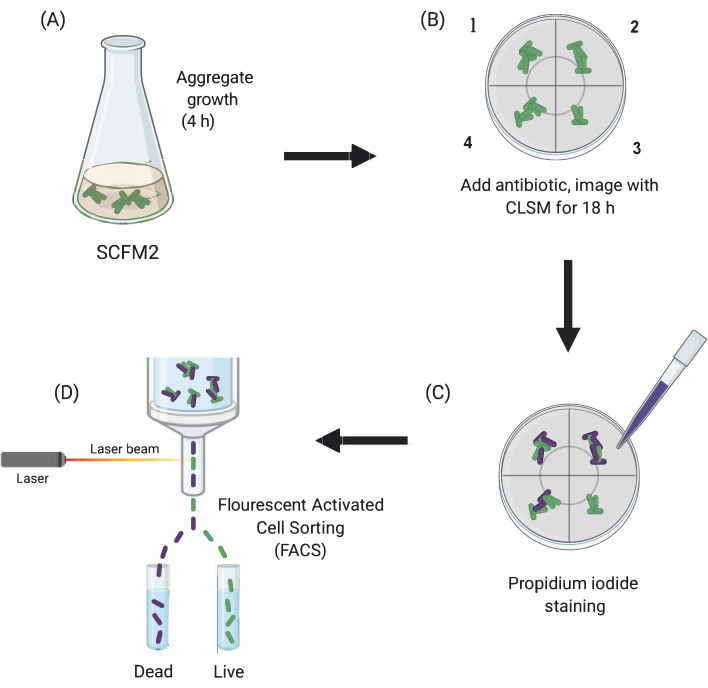
Figur 1: Grafisk fremstilling av de viktigste eksperimentelle trinnene. (A) SCFM2 er inokulert med Pa-celler og har lov til å danne aggregater i en glassbunnet kulturrett. (B) Aggregater overføres til det konfiske mikroskopet, og antibiotika tilsettes. Avbildet er tre tekniske replikeringer (kamre 1-3) og en kontrollbrønn (4) inokulert SCFM2 uten antibiotikabehandling. Aggregater er avbildet ved hjelp av CLSM i løpet av 18 h. (C) Etter den første 18-timers avbildningen behandles aggregater med propidiumjodid for å visualisere døde celler og avbildet ved hjelp av CLSM (D) Aggregater med ønsket fenotype er skilt fra SCFM2 ved hjelp av FACS. Forkortelser: SCFM2 = syntetisk cystisk fibrose sputum medium; Pa = Pseudomonas aeruginosa; CLSM = konfekt laser skanning mikroskopi; FACS = fluorescensaktivert cellesortering. Klikk her for å se en større versjon av denne figuren.
Her demonstreres nytten av SCFM2 for å studere effekten av antibiotikabehandling på Pa-aggregater i sanntid, etterfulgt av bruk av en cellesorteringstilnærming for å isolere populasjoner av aggregater med distinkte fenotyper for nedstrømsanalyse (figur 1).
Protocol
Representative Results
Discussion
Dette arbeidet har innført metoder som kan kombineres for å studere bakterielle aggregerte populasjoner i nærvær og fravær av antibiotikabehandling. Høyoppløselig CLSM tillater visualisering av endringer i aggregert biomasse og strukturell orientering av aggregater over sanntid når de utsettes for antibiotika. I tillegg kan fysiske og strukturelle egenskaper ved biomassen som forblir etter behandling med antibiotika kvantifiseres, med sikte på å korrelere disse observasjonene med fremtidige genuttrykksstudier v…
Disclosures
The authors have nothing to disclose.
Acknowledgements
S.E.D støttes av oppstartsmidler levert av Department of Molecular Medicine, University of South Florida, samt et CFF-forskningsstipend (DARCH19G0) N.I.H (5R21AI147654 – 02 (PI, Chen)) og USF Institute on Microbiomes. Vi takker Whiteley-laboratoriet for løpende samarbeid med datasett relatert til dette manuskriptet. Vi takker Dr. Charles Szekeres for å legge til rette for FACS-sortering. Tall ble opprettet av A.D.G og S.E.D ved hjelp av Biorender.com.
Materials
| Amino acids | |||
| Alanine | Acr s Organics s Organics |
56-41-7 | |
| Arginine HCl | MP | 1119-34-2 | |
| Asparagine | Acr s Organics s Organics |
56-84-8 | Prepared in 0.5 M NaOH |
| Cystine HCl | Alfa Aesar | L06328 | |
| Glutamic acid HCl | Acr s Organics s Organics |
138-15-8 | |
| Glycine | Acr s Organics s Organics |
56-40-6 | |
| Histidine HCl H2O | Alfa Aesar | A17627 | |
| Isoleucine | Acr s Organics s Organics |
73-32-5 | |
| Leucine | Alfa Aesar | A12311 | |
| Lysine HCl | Alfa Aesar | J62099 | |
| Methionine | Acr s Organics s Organics |
63-68-3 | |
| Ornithine HCl | Alfa Aesar | A12111 | |
| Phenylalanine | Acr s Organics s Organics |
63-91-2 | |
| Proline | Alfa Aesar | A10199 | |
| Serine | Alfa Aesar | A11179 | |
| Threonine | Acr s Organics s Organics |
72-19-5 | |
| Tryptophan | Acr s Organics s Organics |
73-22-3 | Prepared in 0.2 M NaOH |
| Tyrosine | Alfa Aesar | A11141 | Prepared in 1.0 M NaOH |
| Valine | Acr s Organics s Organics |
72-18-4 | |
| Antibiotic | |||
| Carbenicillin | Alfa Aesar | J6194903 | |
| Day-of Stocks | |||
| CaCl2 * 2H2O | Fisher Chemical | C79-500 | |
| Dextrose (D-glucose) | Fisher Chemical | 50-99-7 | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Fisher (Avanti Polar Lipids) | 4235-95-4 | shake 15-20 min at 37 °C to evaporate chloroform |
| FeSO4 * 7H2O | Acr s Organics s Organics |
7782-63-0 | this stock equals 1 mg/mL, MUST make fresh |
| L-lactic acid | Alfa Aesar | L13242 | pH stock to 7 with NaOH |
| MgCl2 * 6H2O | Acr s Organics s Organics |
7791-18-6 | |
| N-acetylglucosamine | TCI | A0092 | |
| Prepared solids | |||
| Porcine mucin | Sigma | M1778-100G | UV-sterilize |
| Salmon sperm DNA | Invitrogen | 15632-011 | |
| Stain | |||
| Propidium iodide | Alfa Aesar | J66764MC | |
| Salts | |||
| K2SO4 | Alfa Aesar | A13975 | |
| KCl | Alfa Aesar | J64189 | add solid directly to buffered base |
| KNO3 | Acr s Organics s Organics |
7757-79-1 | |
| MOPS | Alfa Aesar | A12914 | add solid directly to buffered base |
| NaCl | Fisher Chemical | S271-500 | add solid directly to buffered base |
| Na2HPO4 | RPI | S23100-500.0 | |
| NaH2PO4 | RPI | S23120-500.0 | |
| NH4Cl | Acr s Organics s Organics |
12125-02-9 | add solid directly to buffered base |
| Consumables | |||
| Conical tubes (15 mL) | Olympus plastics | 28-101 | |
| Conical tubes (50 mL) | Olympus plastics | 28-106 | |
| Culture tubes w/air flow cap | Olympus plastics | 21-129 | |
| 35 mm four chamber glass-bottom dish | CellVis | NC0600518 | |
| Luria Bertani (LB) broth | Genessee Scientific | 11-118 | |
| Phosphate-buffered saline (PBS) | Fisher Bioreagents | BP2944100 | |
| Pipet tips (p200) | Olympus plastics | 23-150RL | |
| Pipet tips (p1000) | Olympus plastics | 23-165RL | |
| Serological pipets (5 mL) | Olympus plastics | 12-102 | |
| Serological pipets (25 mL) | Olympus plastics | 12-106 | |
| Serological pipets (50 mL) | Olympus plastics | 12-107 | |
| Ultrapure water (RNAse/DNAse free); nanopure water | Genessee Scientific | 18-194 | Nanopure water used for preparation of solutions in Table 1 |
| Syringes (10 mL) | BD | 794412 | |
| Syringes (50 mL) | BD | 309653 | |
| 0.22 mm PES syringe filter | Olympus plastics | 25-244 | |
| PS cuvette semi-mico | Olympus plastics | 91-408 | |
| Software | |||
| Biorender | To prepare the figures | ||
| FacsDiva6.1.3 | Becton Dickinson, San Jose, CA | ||
| Imaris | Bitplane | version 9.6 | |
| Zen Black | |||
| Equipment | |||
| FacsAriallu | Becton Dickinson, San Jose, CA | ||
| LSM 880 confocal laser scanning microscope | Zeiss |
References
- Ramsay, K. A., et al. The changing prevalence of pulmonary infection in with fibrosis: A longitudinal analysis. Journal of Cystic Fibrosis. 16 (1), 70-77 (2017).
- Bessonova, L., et al. Data from the US and UK cystic fibrosis registries support disease modification by CFTR modulation with ivacaftor. Thorax. 73 (8), 731-740 (2018).
- Breuer, O., et al. Changing prevalence of lower airway infections in young children with cystic fibrosis. American Journal of Respiratory and Critical Care Medicine. 200 (5), 590-599 (2019).
- O’Donnell, J. N., Bidell, M. R., Lodise, T. P. Approach to the treatment of patients with serious multidrug-resistant Pseudomonas aeruginosa infections. Pharmacotherapy. 40 (9), 952-969 (2020).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Darch, S. E., et al. Spatial determinants of quorum signaling in a Pseudomonas aeruginosa infection model. Proceedings of the National Academy of Sciences of the United States of America. 115 (18), 4779-4784 (2018).
- Zhu, K., Chen, S., Sysoeva, T. A., You, L. Universal antibiotic tolerance arising from antibiotic-triggered accumulation of pyocyanin in Pseudomonas aeruginosa. PLoS Biology. 17 (12), 3000573 (2019).
- Ciofu, O., Tolker-Nielsen, T. Tolerance and resistance of Pseudomonas aeruginosa biofilms to antimicrobial agents-how P. aeruginosa can escape antibiotics. Frontiers in Microbiology. 10, 913 (2019).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proceedings of the National Academy of Sciences of the United States of America. 112 (13), 4110-4115 (2015).
- Darch, S. E., et al. Phage inhibit pathogen dissemination by targeting bacterial migrants in a chronic infection model. MBio. 8 (2), 00240 (2017).
- Jorth, P., et al. Regional isolation drives bacterial diversification within cystic fibrosis lungs. Cell Host & Microbe. 18 (3), 307-319 (2015).
- Palmer, K. L., Aye, L. M., Whiteley, M. Nutritional cues control Pseudomonas aeruginosa multicellular behavior in cystic fibrosis sputum. Journal of Bacteriology. 189 (22), 8079-8087 (2007).
- Davies, D. G., et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science. 280 (5361), 295-298 (1998).
- Hartmann, R., et al. Quantitative image analysis of microbial communities with BiofilmQ. Nature Microbiology. 6 (2), 151-156 (2021).
- Stacy, A., et al. Bacterial fight-and-flight responses enhance virulence in a polymicrobial infection. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7819-7824 (2014).

