Ferramentas para a avaliação em tempo real de um modelo de infecção por pseudomonas aeruginosa
Summary
O meio de escarro de fibrose cística sintética (SCFM2) pode ser utilizado em combinação com microscopia de varredura a laser confocal e classificação celular ativada por fluorescência para observar agregados bacterianos em alta resolução. Este artigo detalha métodos para avaliar populações agregadas durante o tratamento antimicrobiano como uma plataforma para estudos futuros.
Abstract
Pseudomonas aeruginosa (Pa) é um dos patógenos oportunistas mais comuns associados à fibrose cística (CF). Uma vez estabelecida a colonização pa, uma grande proporção das bactérias infectantes formam biofilmes dentro da escória das vias aéreas. Os biofilmes pa isolados da escarro cf têm se mostrado que crescem em pequenos agregados densos de ~10-1.000 células que são organizadas espacialmente e exibem fenótipos clinicamente relevantes, como a tolerância antimicrobiana. Um dos maiores desafios para estudar como os agregados pa respondem à mudança do ambiente de escarro é a falta de sistemas nutricionalmente relevantes e robustos que promovam a formação agregada. Utilizando um meio de escarro CF sintético (SCFM2), a história de vida dos agregados pa pode ser observada usando microscopia de varredura a laser confocal (CLSM) e análise de imagem na resolução de uma única célula. Este sistema in vitro permite a observação de milhares de agregados de tamanho variável em tempo real, três dimensões e na escala de mícingos. Nos níveis individual e populacional, ter a capacidade de agrupar agregados por fenótipo e posição facilita a observação de agregados em diferentes estágios de desenvolvimento e sua resposta a mudanças no microambiente, como o tratamento com antibióticos, para ser diferenciado com precisão.
Introduction
Pseudomonas aeruginosa (Pa) é um patógeno oportunista que estabelece infecções crônicas em indivíduos imuno-comprometidos. Para aqueles com a doença genética fibrose cística (CF), essas infecções podem percorrer o curso de uma vida. Cf causa o acúmulo de uma escarro viscosa e rica em nutrientes nas vias aéreas, que se torna colonizada por uma variedade de patógenos microbianos ao longo do tempo. Pa é um dos patógenos cf mais prevalentes, colonizando as vias aéreas na primeira infância e estabelecendo infecções difíceis de tratar1. O pa continua sendo um problema clínico significativo e é considerado uma das principais causas de mortalidade naqueles com CF, apesar da melhoria dos regimes terapêuticos nos últimos anos2,3. Esse fenótipo de persistência e o aumento da tolerância a antibióticos têm conquistado ao Pa um lugar em um grupo de patógenos identificados tanto pelos Centros de Controle de Doenças (CDC) quanto pela Organização Mundial da Saúde (OMS) como prioridades de pesquisa para o desenvolvimento de novas estratégias terapêuticas – os patógenos ESKAPE4.
Como outros patógenos ESKAPE, a resistência a antibióticos adquirida é comum no Pa,mas também existem muitas propriedades intrínsecas que contribuem para a tolerância antimicrobiana pa. Entre eles está a capacidade do Pa de formar aglomerados agregados altamente densos de ~10-1.000 células, que podem ser observadas em múltiplas infecções, incluindo a escarro do paciente CF5,6. Semelhante ao Pa estudado em outros sistemas de biofilme, os agregados pa apresentam fenótipos clinicamente relevantes, como aumento da resistência a antibióticos e ativação da comunicação celular-célula (sensoriamento de quórum (QS). Por exemplo, agregados de Pa têm sido mostrados para usar comportamentos regulados por QS para combater outros micróbios, bem como tolerar tratamentos antimicrobianos, como a produção de pyocyanin7. A capacidade de estudar tais comportamentos oferece uma visão emocionante dos ecossistemas bacterianos em um ambiente semelhante ao em que eles existem no corpo humano.
Um dos maiores desafios para estudar como os agregados pa respondem à mudança do ambiente de escarro é a falta de sistemas nutricionalmente relevantes e robustos que promovam a formação agregada. Muito do que se sabe sobre pa foi descoberto usando sistemas in vitro nos quais as células crescem pltipicamente ou em uma arquitetura característica anexada à superfície, “cogumelo” que não foi observada in vivo8. Embora modelos clássicos de crescimento de biofilmes, como células de fluxo ou ágar sólido, tenham gerado amplo e valioso conhecimento sobre comportamentos bacterianos e mecanismos de tolerância a antibióticos, esses achados nem sempre se traduzem in vivo. Muitos modelos in vitro têm uma capacidade limitada de imitar o ambiente de crescimento do local de infecção humana, exigindo estudos in vivo caros. Por sua vez, muitos modelos in vivo carecem da flexibilidade e resolução proporcionadas por técnicas in vitro.
A fibrose sintética sputum (SCFM2) foi projetada para fornecer um ambiente para o crescimento de Pa semelhante ao experimentado durante a infecção crônica no pulmão CF. O SCFM2 inclui fontes nutricionais identificadas em sputa cf gestante, além de mucina, lipídios e DNA. O crescimento de Pa no SCFM2 requer um conjunto genético quase idêntico ao necessário para o crescimento da escória real e suporta a formação agregada pa natural9,10. Após a inoculação, as células planctônicas formam agregados que aumentam de tamanho através da expansão. As células individuais (referidas como migrantes) são liberadas de agregados, migram para áreas não colonizadas e formam novos agregados10. Este histórico de vida pode ser observado usando CLSM e análise de imagem na resolução de uma única célula. Os agregados de Pa formados em SCFM2 são de tamanhos semelhantes aos observados no pulmãoCF 10. Este modelo permite a observação de múltiplos agregados de tamanho variável em tempo real e em três dimensões na escala de mícingos. A microscopia de lapso de tempo permite o rastreamento de milhares (~50.000) de agregados em um experimento. O uso de software de análise de imagens permite a quantificação de fenótipos agregados de micrografos, incluindo volume agregado, área de superfície e posição em três dimensões até os 0,1 μm mais próximos, tanto nos níveis agregado individual quanto populacional. Ter a capacidade de agrupar agregados por fenótipo e posição permite a diferenciação de agregados em diferentes estágios de desenvolvimento com precisão, bem como sua resposta a um microambiente em mudança6,11.
A aplicação do SCFM2 para estudar agregados pa em ensaios de baixo volume e alto rendimento torna-o um modelo flexível e econômico. Como um meio definido, o SCFM2 oferece uniformidade e reprodutibilidade em várias plataformas, fornecendo um método nutricional e fisicamente relevante para estudar agregados pa in vitro9. As aplicações incluem seu uso em combinação com o CLSM para observar a organização espacial e a tolerância a antibióticos em alta resolução (como descrito neste artigo de métodos). A capacidade de realizar experimentos que fornecem dados em tempo real, em escala de micron, permite o estudo de interações intra-espécies e interespécies, pois elas podem ocorrer in vivo. Por exemplo, o SCFM2 já foi usado para estudar a dinâmica espacial da comunicação celular em populações agregadas através de uma rede de sistemas utilizados pela Pa para regular múltiplos genes que contribuem para a virulência e a patogênese6.
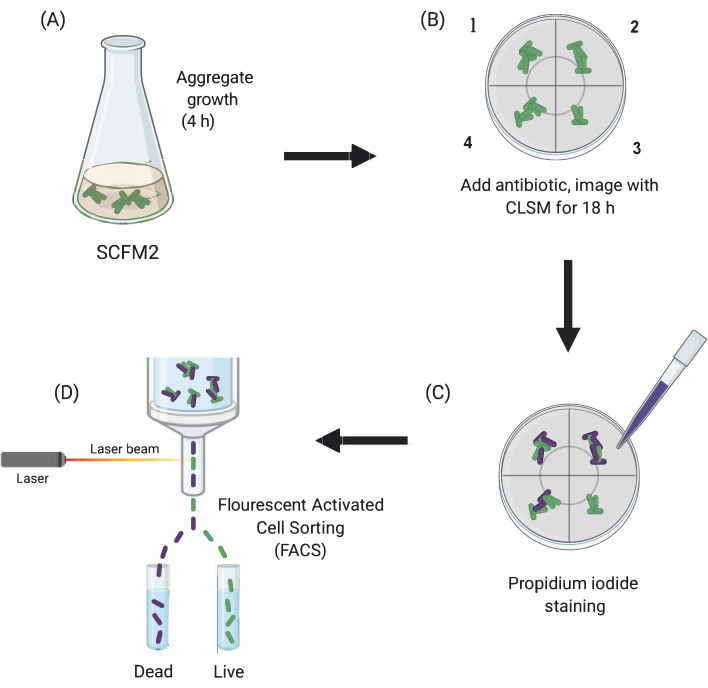
Figura 1: Representação gráfica dos principais passos experimentais. (A) SCFM2 é inoculado com células Pa e permitido formar agregados em um prato de cultura com fundo de vidro. (B) Os agregados são transferidos para o microscópio confocal, e o antibiótico é adicionado. São retratadas três réplicas técnicas (câmaras 1-3) e um poço de controle (4) de SCFM2 inoculado sem tratamento antibiótico. Agregados são imagens usando CLSM ao longo de 18 h. (C) Após a imagem inicial de 18h, os agregados são tratados com iodeto de propídio para visualizar células mortas e imagens usando CLSM (D) Agregados com fenótipo desejado são separados do SCFM2 usando FACS. Abreviaturas: SCFM2 = meio de escarro de fibrose cística sintética; Pa = Pseudomonas aeruginosa; CLSM = microscopia de varredura a laser confocal; FACS = classificação celular ativada por fluorescência. Clique aqui para ver uma versão maior desta figura.
Aqui, demonstra-se a utilidade do SCFM2 para estudar o impacto do tratamento antibiótico nos agregados de pa em tempo real, seguida pelo uso de uma abordagem de classificação celular para isolar populações de agregados com fenótipos distintos para análise a jusante(Figura 1).
Protocol
Representative Results
Discussion
Este trabalho introduziu metodologias que podem ser combinadas para estudar populações bacterianas agregadas na presença e ausência de tratamento antibiótico. O CLSM de alta resolução permite a visualização de mudanças na biomassa agregada e a orientação estrutural dos agregados em tempo real quando expostos a antibióticos. Além disso, podem ser quantificadas as características físicas e estruturais da biomassa que permanecem após o tratamento com antibióticos, com o objetivo de correlacionar essas obse…
Disclosures
The authors have nothing to disclose.
Acknowledgements
A S.E.D é apoiada por fundos iniciantes fornecidos pelo Departamento de Medicina Molecular da Universidade do Sul da Flórida, bem como uma bolsa de pesquisa CFF (DARCH19G0) o N.I.H (5R21AI147654 – 02 (PI, Chen)) e o UsF Institute on Microbiomes. Agradecemos ao laboratório whiteley pela colaboração contínua envolvendo conjuntos de dados relacionados a este manuscrito. Agradecemos ao Dr. Charles Szekeres por facilitar a classificação da FACS. Os números foram criados pela A.D.G e S.E.D usando Biorender.com.
Materials
| Amino acids | |||
| Alanine | Acr s Organics s Organics |
56-41-7 | |
| Arginine HCl | MP | 1119-34-2 | |
| Asparagine | Acr s Organics s Organics |
56-84-8 | Prepared in 0.5 M NaOH |
| Cystine HCl | Alfa Aesar | L06328 | |
| Glutamic acid HCl | Acr s Organics s Organics |
138-15-8 | |
| Glycine | Acr s Organics s Organics |
56-40-6 | |
| Histidine HCl H2O | Alfa Aesar | A17627 | |
| Isoleucine | Acr s Organics s Organics |
73-32-5 | |
| Leucine | Alfa Aesar | A12311 | |
| Lysine HCl | Alfa Aesar | J62099 | |
| Methionine | Acr s Organics s Organics |
63-68-3 | |
| Ornithine HCl | Alfa Aesar | A12111 | |
| Phenylalanine | Acr s Organics s Organics |
63-91-2 | |
| Proline | Alfa Aesar | A10199 | |
| Serine | Alfa Aesar | A11179 | |
| Threonine | Acr s Organics s Organics |
72-19-5 | |
| Tryptophan | Acr s Organics s Organics |
73-22-3 | Prepared in 0.2 M NaOH |
| Tyrosine | Alfa Aesar | A11141 | Prepared in 1.0 M NaOH |
| Valine | Acr s Organics s Organics |
72-18-4 | |
| Antibiotic | |||
| Carbenicillin | Alfa Aesar | J6194903 | |
| Day-of Stocks | |||
| CaCl2 * 2H2O | Fisher Chemical | C79-500 | |
| Dextrose (D-glucose) | Fisher Chemical | 50-99-7 | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Fisher (Avanti Polar Lipids) | 4235-95-4 | shake 15-20 min at 37 °C to evaporate chloroform |
| FeSO4 * 7H2O | Acr s Organics s Organics |
7782-63-0 | this stock equals 1 mg/mL, MUST make fresh |
| L-lactic acid | Alfa Aesar | L13242 | pH stock to 7 with NaOH |
| MgCl2 * 6H2O | Acr s Organics s Organics |
7791-18-6 | |
| N-acetylglucosamine | TCI | A0092 | |
| Prepared solids | |||
| Porcine mucin | Sigma | M1778-100G | UV-sterilize |
| Salmon sperm DNA | Invitrogen | 15632-011 | |
| Stain | |||
| Propidium iodide | Alfa Aesar | J66764MC | |
| Salts | |||
| K2SO4 | Alfa Aesar | A13975 | |
| KCl | Alfa Aesar | J64189 | add solid directly to buffered base |
| KNO3 | Acr s Organics s Organics |
7757-79-1 | |
| MOPS | Alfa Aesar | A12914 | add solid directly to buffered base |
| NaCl | Fisher Chemical | S271-500 | add solid directly to buffered base |
| Na2HPO4 | RPI | S23100-500.0 | |
| NaH2PO4 | RPI | S23120-500.0 | |
| NH4Cl | Acr s Organics s Organics |
12125-02-9 | add solid directly to buffered base |
| Consumables | |||
| Conical tubes (15 mL) | Olympus plastics | 28-101 | |
| Conical tubes (50 mL) | Olympus plastics | 28-106 | |
| Culture tubes w/air flow cap | Olympus plastics | 21-129 | |
| 35 mm four chamber glass-bottom dish | CellVis | NC0600518 | |
| Luria Bertani (LB) broth | Genessee Scientific | 11-118 | |
| Phosphate-buffered saline (PBS) | Fisher Bioreagents | BP2944100 | |
| Pipet tips (p200) | Olympus plastics | 23-150RL | |
| Pipet tips (p1000) | Olympus plastics | 23-165RL | |
| Serological pipets (5 mL) | Olympus plastics | 12-102 | |
| Serological pipets (25 mL) | Olympus plastics | 12-106 | |
| Serological pipets (50 mL) | Olympus plastics | 12-107 | |
| Ultrapure water (RNAse/DNAse free); nanopure water | Genessee Scientific | 18-194 | Nanopure water used for preparation of solutions in Table 1 |
| Syringes (10 mL) | BD | 794412 | |
| Syringes (50 mL) | BD | 309653 | |
| 0.22 mm PES syringe filter | Olympus plastics | 25-244 | |
| PS cuvette semi-mico | Olympus plastics | 91-408 | |
| Software | |||
| Biorender | To prepare the figures | ||
| FacsDiva6.1.3 | Becton Dickinson, San Jose, CA | ||
| Imaris | Bitplane | version 9.6 | |
| Zen Black | |||
| Equipment | |||
| FacsAriallu | Becton Dickinson, San Jose, CA | ||
| LSM 880 confocal laser scanning microscope | Zeiss |
References
- Ramsay, K. A., et al. The changing prevalence of pulmonary infection in with fibrosis: A longitudinal analysis. Journal of Cystic Fibrosis. 16 (1), 70-77 (2017).
- Bessonova, L., et al. Data from the US and UK cystic fibrosis registries support disease modification by CFTR modulation with ivacaftor. Thorax. 73 (8), 731-740 (2018).
- Breuer, O., et al. Changing prevalence of lower airway infections in young children with cystic fibrosis. American Journal of Respiratory and Critical Care Medicine. 200 (5), 590-599 (2019).
- O’Donnell, J. N., Bidell, M. R., Lodise, T. P. Approach to the treatment of patients with serious multidrug-resistant Pseudomonas aeruginosa infections. Pharmacotherapy. 40 (9), 952-969 (2020).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Darch, S. E., et al. Spatial determinants of quorum signaling in a Pseudomonas aeruginosa infection model. Proceedings of the National Academy of Sciences of the United States of America. 115 (18), 4779-4784 (2018).
- Zhu, K., Chen, S., Sysoeva, T. A., You, L. Universal antibiotic tolerance arising from antibiotic-triggered accumulation of pyocyanin in Pseudomonas aeruginosa. PLoS Biology. 17 (12), 3000573 (2019).
- Ciofu, O., Tolker-Nielsen, T. Tolerance and resistance of Pseudomonas aeruginosa biofilms to antimicrobial agents-how P. aeruginosa can escape antibiotics. Frontiers in Microbiology. 10, 913 (2019).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proceedings of the National Academy of Sciences of the United States of America. 112 (13), 4110-4115 (2015).
- Darch, S. E., et al. Phage inhibit pathogen dissemination by targeting bacterial migrants in a chronic infection model. MBio. 8 (2), 00240 (2017).
- Jorth, P., et al. Regional isolation drives bacterial diversification within cystic fibrosis lungs. Cell Host & Microbe. 18 (3), 307-319 (2015).
- Palmer, K. L., Aye, L. M., Whiteley, M. Nutritional cues control Pseudomonas aeruginosa multicellular behavior in cystic fibrosis sputum. Journal of Bacteriology. 189 (22), 8079-8087 (2007).
- Davies, D. G., et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science. 280 (5361), 295-298 (1998).
- Hartmann, R., et al. Quantitative image analysis of microbial communities with BiofilmQ. Nature Microbiology. 6 (2), 151-156 (2021).
- Stacy, A., et al. Bacterial fight-and-flight responses enhance virulence in a polymicrobial infection. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7819-7824 (2014).

