Идентификация альтернативного сплайсинга и полиаденилирования в данных RNA-seq
Summary
Альтернативное сплайсинг (AS) и альтернативное полиаденилирование (APA) расширяют разнообразие изоформ транскриптов и их продуктов. Здесь мы описываем биоинформационные протоколы для анализа объемных анализов РНК-seq и 3′-концевого секвенирования для обнаружения и визуализации AS и APA, варьирующихся в зависимости от экспериментальных условий.
Abstract
Наряду с типичным анализом RNA-Seq для измерения дифференциальной экспрессии генов (DGE) в экспериментальных / биологических условиях, данные RNA-seq также могут быть использованы для изучения других сложных регуляторных механизмов на уровне экзонов. Альтернативное сплайсинг и полиаденилирование играют решающую роль в функциональном разнообразии гена, генерируя различные изоформы для регулирования экспрессии генов на посттранскрипционном уровне, и ограничение анализа всем генным уровнем может пропустить этот важный регуляторный слой. Здесь мы демонстрируем подробный пошаговый анализ для идентификации и визуализации использования дифференциального экзона и полиаденилирования в разных условиях с использованием биопроводника и других пакетов и функций, включая DEXSeq, diffSplice из пакета Limma и rMATS.
Introduction
RNA-seq широко использовался на протяжении многих лет, как правило, для оценки дифференциальной экспрессии генов и открытия генов1. Кроме того, он также может быть использован для оценки различного использования на уровне экзонов из-за генов, экспрессирующих различные изоформы, что способствует лучшему пониманию регуляции генов на посттранскрипционном уровне. Большинство эукариотических генов генерируют различные изоформы путем альтернативного сплайсинга (AS) для увеличения разнообразия экспрессии мРНК. События AS можно разделить на различные паттерны: пропуск полных экзонов (SE), где («кассетный») экзон полностью удаляется из стенограммы вместе с его фланкирующими интронами; альтернативный (донорский) 5-футовый выбор места сращивания (A5SS) и альтернативный 3-дюймовый (акцепторный) выбор места сращивания (A3SS), когда два или более участков сращивания присутствуют на обоих концах экзона; удержание интронов (RI), когда интрон сохраняется в зрелом транскрипте мРНК, и взаимное исключение использования экзона (MXE), где только один из двух доступных экзонов может быть сохранен за один раз 2,3. Альтернативное полиаденилирование (АПА) также играет важную роль в регулировании экспрессии генов с использованием альтернативных поли(А) сайтов для генерации нескольких изоформ мРНК из одного транскрипта4. Большинство участков полиаденилирования (pAs) расположены в 3′ нетранслируемой области (3′ UTR), генерируя изоформы мРНК с различными длинами UTR 3′. Поскольку 3′ UTR является центральным узлом для распознавания регуляторных элементов, различные длины 3′ UTR могут влиять на локализацию, стабильность и трансляцию мРНК5. Существует класс 3′-концевых анализов секвенирования, оптимизированных для обнаружения APA, которые отличаются деталями протокола6. Конвейер, описанный здесь, предназначен для PolyA-seq, но может быть адаптирован для других протоколов, как описано.
В данном исследовании мы представляем конвейер методов дифференциального анализа экзонов 7,8 (рисунок 1), которые можно разделить на две широкие категории: основанные на экзонах (DEXSeq9, diffSplice10) и событийные (реплицированный многомерный анализ сплайсинга транскриптов (rMATS)11). Методы, основанные на экзонах, сравнивают изменение складки в разных условиях отдельных экзонов с мерой общего изменения складки генов, чтобы вызвать дифференциально экспрессированное использование экзонов, и на основе этого вычисляют меру активности АС на уровне гена. Методы, основанные на событиях, используют считывание переходов, охватывающих экзон-интрон, для обнаружения и классификации конкретных событий сплайсинга, таких как пропуск экзона или удержание интронов, и различают эти типы AS в выходных данных3. Таким образом, эти методы обеспечивают взаимодополняющие взгляды для полного анализа AS 12,13. Мы выбрали DEXSeq (на основе пакета DESeq214 DGE) и diffSplice (на основе пакета Limma10 DGE) для исследования, поскольку они являются одними из наиболее широко используемых пакетов для дифференциального анализа сплайсинга. rMATS был выбран в качестве популярного метода для анализа на основе событий. Другим популярным методом, основанным на событиях, является MISO (Смесь изоформ)1. Для APA мы адаптируем подход, основанный на экзонах.
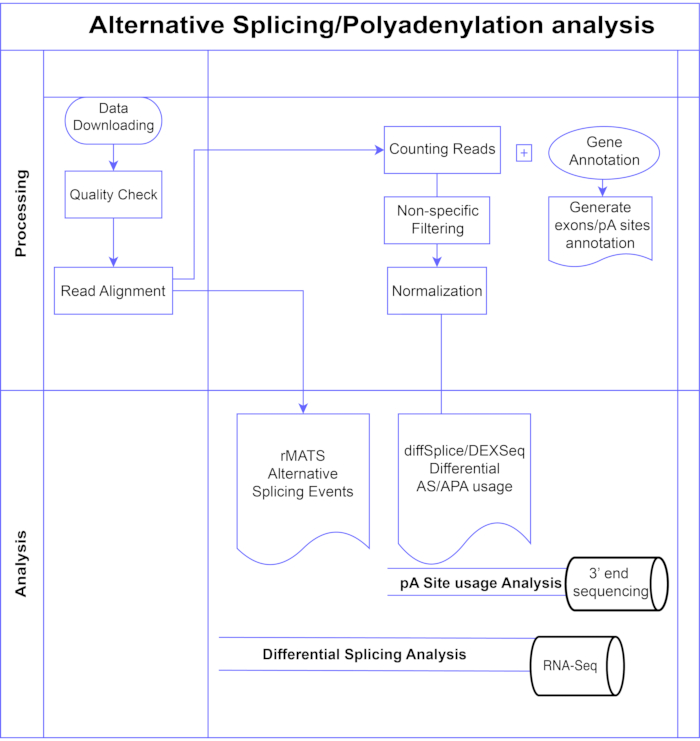
Рисунок 1. Конвейер анализа. Блок-схема шагов, используемых в анализе. Шаги включают в себя: получение данных, выполнение проверок качества и выравнивания считывания с последующим подсчетом считываний с использованием аннотаций для известных экзонов, интронов и участков pA, фильтрацию для удаления низких значений и нормализации. Данные PolyA-seq были проанализированы для альтернативных участков pA с использованием методов diffSplice/DEXSeq, объемная РНК-Seq была проанализирована для альтернативного сплайсинга на уровне экзона методами diffSplice/DEXseq, а события AS анализировались с помощью rMATS. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Данные RNA-seq, использованные в этом исследовании, были получены из Gene Expression Omnibus (GEO) (GSE138691)15. Мы использовали данные RNA-seq мышей из этого исследования с двумя группами состояний: дикий тип (WT) и Muscleblind-подобный нокаут типа 1 (Mbnl1 KO) с тремя репликами в каждой. Чтобы продемонстрировать дифференциальный анализ использования участка полиаденилирования, мы получили данные PolyA-seq о фибробластах эмбриона мыши (MEFs) (GEO Accession GSE60487)16. Данные имеют четыре группы условий: Wild-type (WT), Muscleblind-like type1/type 2 double knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO с Mbnl3 knockdown (KD) и Mbnl1/2 DKO с управлением Mbnl3 (Ctrl). Каждая группа условий состоит из двух реплик.
| Присоединение к ГЭП | Номер запуска SRA | Пример имени | Состояние | Повторять | Ткань | Секвенирование | Длина чтения | |
| РНК-Сек | GSM4116218 | СРР10261601 | Mbnl1KO_Thymus_1 | Нокаут Mbnl1 | Представитель 1 | Тимус | Сопряженный конец | 100 бит/с |
| GSM4116219 | СРР10261602 | Mbnl1KO_Thymus_2 | Нокаут Mbnl1 | Представитель 2 | Тимус | Сопряженный конец | 100 бит/с | |
| GSM4116220 | СРР10261603 | Mbnl1KO_Thymus_3 | Нокаут Mbnl1 | Представитель 3 | Тимус | Сопряженный конец | 100 бит/с | |
| GSM4116221 | СРР10261604 | WT_Thymus_1 | Дикий тип | Представитель 1 | Тимус | Сопряженный конец | 100 бит/с | |
| GSM4116222 | СРР10261605 | WT_Thymus_2 | Дикий тип | Представитель 2 | Тимус | Сопряженный конец | 100 бит/с | |
| GSM4116223 | СРР10261606 | WT_Thymus_3 | Дикий тип | Представитель 3 | Тимус | Сопряженный конец | 100 бит/с | |
| 3П-Сек | GSM1480973 | СРР1553129 | WT_1 | Дикий тип (WT) | Представитель 1 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с |
| GSM1480974 | СРР1553130 | WT_2 | Дикий тип (WT) | Представитель 2 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480975 | СРР1553131 | DKO_1 | Mbnl 1/2 двойной нокаут (DKO) | Представитель 1 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480976 | СРР1553132 | DKO_2 | Mbnl 1/2 двойной нокаут (DKO) | Представитель 2 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480977 | СРР1553133 | DKOsiRNA_1 | Mbnl 1/2 двойной нокаут с Mbnl 3 siRNA (KD) | Представитель 1 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480978 | СРР1553134 | DKOsiRNA_2 | Mbnl 1/2 двойной нокаут с Mbnl 3 siRNA (KD) | Представитель 2 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 36 бит/с | |
| GSM1480979 | СРР1553135 | DKONTsiRNA_1 | Mbnl 1/2 двойной нокаут с нецелевым siRNA (Ctrl) | Представитель 1 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480980 | СРР1553136 | DKONTsiRNA_2 | Mbnl 1/2 двойной нокаут с нецелевым siRNA (Ctrl) | Представитель 2 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с |
Таблица 1. Резюме наборов данных RNA-Seq и PolyA-seq, используемых для анализа.
Protocol
Representative Results
Discussion
В этом исследовании мы оценили основанные на экзонах и событиях подходы к обнаружению AS и APA в объемных данных СЕКВЕНИРОВАНИЯ РНК-Seq и 3′. Подходы AS, основанные на экзонах, дают как список дифференциально экспрессированных экзонов, так и ранжирование на уровне генов, упорядоченное по стат…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Это исследование было поддержано Австралийским исследовательским советом (ARC) Future Fellowship (FT16010043) и ANU Futures Scheme.
References
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

