मोबाइल एकल अणु FRET जांच के स्वचालित दो आयामी Spatiotemporal विश्लेषण
Summary
यह लेख मोबाइल, एकल-अणु Förster अनुनाद ऊर्जा हस्तांतरण (smFRET) के spatiotemporal विश्लेषण के लिए एक विधि प्रस्तुत करता है- वाइडफील्ड प्रतिदीप्ति माइक्रोस्कोपी का उपयोग करके आधारित जांच। नव विकसित सॉफ्टवेयर टूलकिट समय के कार्यों के रूप में सही FRET दक्षता और आणविक पदों सहित चलती जांच के smFRET समय के निशान के निर्धारण की अनुमति देता है।
Abstract
एकल-अणु Förster अनुनाद ऊर्जा हस्तांतरण (smFRET) एक बहुमुखी तकनीक नैनोमीटर रेंज के लिए उप-नैनोमीटर में दूरी पर रिपोर्टिंग है। इसका उपयोग बायोफिजिकल और आणविक जैविक प्रयोगों की एक विस्तृत श्रृंखला में किया गया है, जिसमें आणविक बलों का माप, बायोमोलेक्यूल्स की संरचनात्मक गतिशीलता का लक्षण वर्णन, प्रोटीन के इंट्रासेल्युलर कोलोकलाइजेशन का अवलोकन और रिसेप्टर-लिगैंड इंटरैक्शन समय का निर्धारण शामिल है। एक वाइडफील्ड माइक्रोस्कोपी कॉन्फ़िगरेशन में, प्रयोग आमतौर पर सतह-स्थिर जांच का उपयोग करके किए जाते हैं। यहां, वैकल्पिक उत्तेजना (एलेक्स) smFRET प्रयोगों के साथ एकल-अणु ट्रैकिंग के संयोजन की एक विधि प्रस्तुत की जाती है, जो प्लाज्मा झिल्ली या ग्लास-समर्थित लिपिड बाईलेयर में सतह-बाध्य, फिर भी मोबाइल जांच के smFRET समय के निशान के अधिग्रहण की अनुमति देती है। रिकॉर्ड किए गए डेटा के विश्लेषण के लिए, एक स्वचालित, ओपन-सोर्स सॉफ़्टवेयर संग्रह विकसित किया गया था जो (i) फ्लोरोसेंट संकेतों का स्थानीयकरण, (ii) एकल-कण ट्रैकिंग, (iii) सुधार कारकों सहित FRET-संबंधित मात्राओं का निर्धारण, (iv) smFRET निशान का कठोर सत्यापन, और (v) परिणामों की सहज ज्ञान युक्त प्रस्तुति का समर्थन करता है। उत्पन्न डेटा को विशेष सॉफ़्टवेयर के माध्यम से आगे की खोज के लिए इनपुट के रूप में आसानी से उपयोग किया जा सकता है, उदाहरण के लिए, जांच के प्रसार व्यवहार के मूल्यांकन या FRET संक्रमण की जांच के लिए।
Introduction
Förster अनुनाद ऊर्जा हस्तांतरण (FRET) आणविक जैविक और biophysical अनुसंधान में एक प्रमुख चालक रहा है, क्योंकि यह उप-नैनोमीटर रिज़ॉल्यूशन पर प्रक्रियाओं की जांच की अनुमति देता है। चूंकि दाता और स्वीकर्ता फ्लोरोफोरस के बीच ऊर्जा हस्तांतरण की दक्षता दृढ़ता से नैनोमीटर रेंज के लिए उप-नैनोमीटर में अंतर-डाई दूरी पर निर्भर करती है, इसलिए इसका उपयोग बायोमोलेक्यूल्स 1,2,3,4 की स्थिर और गतिशील संरचना का पता लगाने के लिए एक स्पेक्ट्रोस्कोपिक शासक के रूप में प्रभावी रूप से किया गया है। इसके अतिरिक्त, FRET घटना व्यापक रूप से एक थोक स्तर पर झिल्ली से जुड़े और इंट्रासेल्युलर प्रोटीन के colocalization अध्ययन के लिए इस्तेमाल किया गया है5,6. पिछले दो दशकों में, इस विधि को smFRET events7 की निगरानी के लिए अनुकूलित किया गया था, जिसने अस्थायी और स्थानिक संकल्प को काफी हद तक बढ़ाने में मदद की और विषम नमूनों में दुर्लभ उप-आबादी को भी हल किया। इन तकनीकों से सुसज्जित, अद्वितीय अंतर्दृष्टि को आणविक मशीनरी की गतिशीलता में प्राप्त किया गया था जैसे कि आरएनए पोलीमरेज़ II8 की प्रतिलेख प्रसंस्करण दर, डीएनए पोलीमरेज़ 9,10 की प्रतिकृति गति, न्यूक्लियोसोम ट्रांसलोकेशन दर 11, ट्रांसक्रिप्ट स्प्लिसिंग और असेंबल किए गए स्प्लिसोसोम 12 की स्टालिंग दर, राइबोसोमल सबपॉपुलेशन 13 की गतिविधि, और किनेसिन मोटर्स की चलने की गति 14 , कुछ नाम देने के लिए। रिसेप्टर-लिगैंड इंटरैक्शन durations15 और आणविक बलों 16 की मात्रा निर्धारित की गई है।
तीव्रता-आधारित smFRET अध्ययन आमतौर पर FRET दक्षता को मापने के लिए संवेदनशील उत्सर्जन पर भरोसा करते हैं: उत्सर्जन पथ में एक बीम विभाजक स्थानिक रूप से दाता से उत्पन्न प्रकाश को अलग करता है और दाता उत्तेजना पर स्वीकर्ता फ्लोरोफोरस से उत्पन्न होता है, जिससे व्यक्तिगत प्रतिदीप्ति तीव्रता के परिमाणीकरण की अनुमति मिलती है। दक्षता की गणना बाद में कुल फोटॉन गणना 17 के संबंध में स्वीकर्ता द्वारा उत्सर्जित फोटॉनों के अंश के रूप में की जा सकती है। इसके अलावा, दाता उत्तेजना (एलेक्स) के बाद स्वीकर्ता उत्तेजना FRET घटनाओं के stoichiometry के माप की अनुमति देता है, उत्पन्न होने वाले संकेतों से सही कम FRET संकेतों के बीच भेदभाव में सहायता करता है, उदाहरण के लिए, एक photobleached स्वीकर्ता fluorophore18 की विशेषता जांच से।
एकल-अणु FRET प्रयोग आमतौर पर दो तरीकों में से एक में किए जाते हैं। सबसे पहले, नमूना मात्रा में एक छोटे से क्षेत्र को एक confocal माइक्रोस्कोप का उपयोग करके प्रकाशित किया जाता है। समाधान में एकल जांच अणु उत्साहित होते हैं जब वे फोकल वॉल्यूम के भीतर फैलते हैं। इस तकनीक के साथ, तेजी से फोटॉन-गिनती डिटेक्टरों का उपयोग किया जा सकता है, जिससे उप-माइक्रोसेकंड टाइम रिज़ॉल्यूशन सक्षम हो सकता है। दूसरा, जांच को विशेष रूप से सतहों पर स्थिर किया जाता है और वाइडफील्ड माइक्रोस्कोपी के माध्यम से निगरानी की जाती है, अक्सर पृष्ठभूमि प्रतिदीप्ति को कम करने के लिए कुल आंतरिक प्रतिबिंब (टीआईआर) कॉन्फ़िगरेशन का उपयोग किया जाता है। जांच immobilization पहले दृष्टिकोण का उपयोग करने की तुलना में बहुत लंबे समय तक रिकॉर्डिंग समय के लिए अनुमति देता है। इसके अलावा, दृश्य का बड़ा क्षेत्र समानांतर में कई जांचों की निगरानी की अनुमति देता है। एक कैमरे की आवश्यकता इस विधि को ऊपर वर्णित एक की तुलना में धीमा बनाती है। समय रिज़ॉल्यूशन मिलीसेकंड से दूसरी श्रेणी तक सीमित है.
यदि लंबे समय के निशान की आवश्यकता होती है, उदाहरण के लिए, एक मिलीसेकंड से दूसरी बार के पैमाने पर गतिशील प्रक्रियाओं का अध्ययन करने के लिए, पहली विधि लागू नहीं होती है, क्योंकि प्रतिदीप्ति फटने आमतौर पर बहुत कम होते हैं। दूसरा दृष्टिकोण विफल हो जाता है जब भी स्थिरीकरण संभव नहीं होता है, उदाहरण के लिए, लाइव-सेल प्रयोगों में कोशिका झिल्ली के भीतर जांच की विशेषता होती है। इसके अलावा, यह देखा गया है कि जैविक मॉडल सिस्टम संपर्क की गई सतह 16 की गतिशीलता के आधार पर नाटकीय रूप से अपनी प्रतिक्रिया को बदल सकते हैं।
जबकि संयुक्त smFRET और एकल कण ट्रैकिंग प्रयोगों मोबाइल FRET जांच रिकॉर्डिंग पिछले 19 में प्रदर्शन किया गया है, वहाँ डेटा के मूल्यांकन के लिए कोई सार्वजनिक रूप से उपलब्ध सॉफ्टवेयर है. इसने एक नए विश्लेषण मंच के विकास को प्रेरित किया, जो मोबाइल फ्लोरोसेंट जांच के कई गुणों के निर्धारण के लिए अनुमति देता है, जिसमें smFRET दक्षता और स्टोइकोमेट्री, उप-पिक्सेल सटीकता के साथ स्थिति, और समय के कार्यों के रूप में प्रतिदीप्ति तीव्रता शामिल है। चरणबद्ध ब्लीचिंग व्यवहार, निकटतम-पड़ोसी दूरी, उत्सर्जन तीव्रता, और अन्य लक्षणों की जांच करके परिणामी निशान को फ़िल्टर करने के तरीके विशेष रूप से सही ढंग से संश्लेषित और कार्यात्मक एकल-जांच अणुओं को चुनने के लिए स्थापित किए गए थे। सॉफ्टवेयर भी प्रयोगात्मक और विश्लेषणात्मक तकनीकों का समर्थन करता है हाल ही में विश्वसनीय, मात्रात्मक smFRET data17 का उत्पादन करने के लिए एक multilaboratory अध्ययन में सहमत हुए. विशेष रूप से, कार्यान्वयन FRET दक्षता और stoichiometry की गणना के लिए मान्य प्रक्रियाओं का पालन करता है। दाता उत्सर्जन चैनल आईडीडी और स्वीकर्ता उत्सर्जन चैनल आईडीए में दाता उत्तेजना पर प्रतिदीप्ति तीव्रता का उपयोग Eq (1) का उपयोग करके स्पष्ट FRET दक्षता Eapp की गणना के लिए किया जाता है।
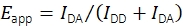 (1)
(1)
स्वीकर्ता उत्तेजना IAA पर स्वीकर्ता उत्सर्जन चैनल में प्रतिदीप्ति तीव्रता की मदद से, स्पष्ट stoichiometry Eq (2) का उपयोग करके गणना की जाती है।
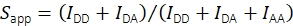 (2)
(2)
FRET दक्षता E और stoichiometry S को चार सुधार कारकों पर विचार करके Eapp और Sapp से प्राप्त किया जा सकता है।
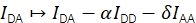
α स्वीकर्ता उत्सर्जन चैनल में दाता प्रतिदीप्ति के रिसाव का वर्णन करता है और केवल दाता फ्लोरोफोरस युक्त नमूने का उपयोग करके या प्रक्षेपवक्र के कुछ हिस्सों का विश्लेषण करके निर्धारित किया जा सकता है जहां स्वीकर्ता को ब्लीच किया गया है। δ दाता उत्तेजना प्रकाश स्रोत द्वारा स्वीकर्ता की प्रत्यक्ष उत्तेजना के लिए सही करता है और केवल स्वीकर्ता fluorophores के साथ एक नमूने का उपयोग करके या प्रक्षेपवक्र के कुछ हिस्सों का विश्लेषण करके मापा जा सकता है जहां दाता को ब्लीच किया गया है।
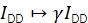 .
.
γ दाता और स्वीकर्ता उत्सर्जन चैनलों और फ्लोरोफोर की विभिन्न क्वांटम दक्षताओं में अलग-अलग पहचान क्षमताओं को सुधारने के लिए आईडीडी को तराजू करता है। कारक उच्च FRET दक्षता 20 के साथ trajectories में स्वीकर्ता विरंजन पर दाता तीव्रता में वृद्धि का विश्लेषण करके या कई असतत FRET राज्यों की विशेषता वाले नमूने का अध्ययन करके गणना की जा सकती है।
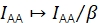
β दाता और स्वीकर्ता उत्तेजना की असमान क्षमताओं के लिए सही करने के लिए आईएए को तराजू करता है। यदि γ स्वीकर्ता ब्लीचिंग विश्लेषण के माध्यम से निर्धारित किया गया था, तो β की गणना ज्ञात दाता-से-स्वीकर्ता अनुपात 21 के नमूने से की जा सकती है। अन्यथा, बहु-राज्य FRET नमूना भी β पैदावार.
साथ में, सुधार Eq (3) का उपयोग करके सही FRET दक्षता की गणना की अनुमति देते हैं।
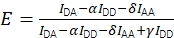 (3)
(3)
और Eq (4) का उपयोग कर सही stoichiometry.
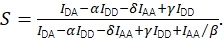 (4)
(4)
आदर्श रूप से, 1: 1 दाता-से-स्वीकर्ता अनुपात के लिए सही स्टोइकोमेट्री एस = 0.5 देता है। व्यवहार में, एक कम सिग्नल-टू-शोर अनुपात एस के मापा मूल्यों का प्रसार पैदा करता है, जो दाता-केवल संकेतों (एस = 1) और स्वीकर्ता-केवल संकेतों (एस = 0) से भेदभाव को बाधित करता है। परिणामी समय के निशान का उपयोग एकल-अणु प्रक्षेपवक्रों के अधिक विस्तृत विश्लेषण के लिए इनपुट के रूप में किया जा सकता है ताकि स्पैटिओटेम्पोरल फोर्स प्रोफाइल 16, एकल-अणु घटनाओं की गतिशीलता 22, या विभिन्न राज्यों के बीच संक्रमण कैनेटीक्स जैसी जानकारी प्राप्त की जा सके।
निम्नलिखित प्रोटोकॉल smFRET ट्रैकिंग प्रयोगों के लिए प्रयोगात्मक मापदंडों और प्रक्रियाओं का वर्णन करता है, साथ ही साथ नए विकसित सॉफ़्टवेयर सूट का उपयोग करके डेटा विश्लेषण के पीछे के कार्य सिद्धांत का वर्णन करता है। प्रयोगात्मक डेटा के अधिग्रहण के लिए, निम्नलिखित आवश्यकताओं को पूरा करने वाले माइक्रोस्कोपी सेटअप का उपयोग करने की सिफारिश की जाती है: i) एकल डाई अणुओं के उत्सर्जन का पता लगाने की क्षमता; ii) वाइडफील्ड रोशनी: विशेष रूप से लाइव-सेल प्रयोगों के लिए, कुल आंतरिक प्रतिबिंब (TIR23,24,25) कॉन्फ़िगरेशन की सिफारिश की जाती है; iii) तरंग दैर्ध्य के अनुसार उत्सर्जन प्रकाश का स्थानिक पृथक्करण जैसे कि दाता और स्वीकर्ता प्रतिदीप्ति को एक ही कैमरा चिप 25 या विभिन्न कैमरों के विभिन्न क्षेत्रों पर प्रक्षेपित किया जाता है; iv) मिलीसेकंड परिशुद्धता के साथ दाता और स्वीकर्ता उत्तेजना के लिए प्रकाश स्रोतों का मॉडुलन, उदाहरण के लिए, एकोस्टो-ऑप्टिक मॉड्यूलेटर के माध्यम से सीधे मॉड्युलेटेबल लेजर या मॉडुलन का उपयोग करना। यह स्ट्रोबोस्कोपिक रोशनी को फ्लोरोफोर के फोटोब्लीचिंग को कम करने के साथ-साथ स्टोइकोमेट्री निर्धारित करने के लिए वैकल्पिक उत्तेजना को कम करने की अनुमति देता है; v) एक प्रारूप है कि PIMS पायथन package26 द्वारा पढ़ा जा सकता है में दर्ज छवि अनुक्रम प्रति एक फ़ाइल का आउटपुट. विशेष रूप से, multipage TIFF फ़ाइलें समर्थित हैं।
Protocol
Representative Results
Discussion
यह लेख स्वचालित रिकॉर्डिंग और smFRET डेटा के मात्रात्मक विश्लेषण के लिए एक पाइपलाइन का विवरण देता है जो मोबाइल अभी तक सतह-टेदर जांच अणुओं से उत्पन्न होता है। यह smFRET प्रयोगों के लिए दो प्रमुख दृष्टिकोणों को पूरक करता है, जिसमें या तो सतह-स्थिर जांच या एक confocal उत्तेजना वॉल्यूम 17 के अंदर और बाहर समाधान में diffusing जांच शामिल है। यह सही FRET दक्षता और समय के एक समारोह के रूप में आणविक पदों प्रदान करता है. इसलिए इसका उपयोग विशेष विश्लेषण कार्यक्रमों के लिए इनपुट के रूप में किया जा सकता है, उदाहरण के लिए, संक्रमण कैनेटीक्स 1, FRET हिस्टोग्राम 39, या दो-आयामी प्रसार 22 को मापने के लिए।
सॉफ्टवेयर को ओपन सोर्स इनिशिएटिव द्वारा अनुमोदित एक मुक्त और ओपन-सोर्स लाइसेंस के तहत जारी किया गया है जो उपयोगकर्ता को मुफ्त उपयोग, संशोधन और पुनर्वितरण का निरंतर अधिकार प्रदान करता है। गिथब को एक विकास और वितरण मंच के रूप में चुना गया था ताकि सॉफ़्टवेयर प्राप्त करने और बग की रिपोर्टिंग या code40 का योगदान करके विकास प्रक्रिया में भाग लेने के लिए जितना संभव हो सके उतना आसान हो सके। पायथन में लिखा गया, सॉफ़्टवेयर मालिकाना घटकों पर निर्भर नहीं करता है। उपयोगकर्ता इंटरफेस के रूप में Jupyter नोटबुक की पसंद हर विश्लेषण चरण में डेटा के निरीक्षण की सुविधा प्रदान करता है और हाथ में प्रयोगात्मक प्रणाली के लिए विशेष रूप से पाइपलाइन को सिलाई और विस्तारित करने की अनुमति देता है। एसडीटी-पायथन लाइब्रेरी 32 नींव के रूप में कार्य करता है और प्रतिदीप्ति माइक्रोस्कोपी डेटा का मूल्यांकन करने के लिए कार्यक्षमता को लागू करता है, जैसे कि एकल-अणु स्थानीयकरण, प्रसार विश्लेषण, प्रतिदीप्ति तीव्रता विश्लेषण, रंग चैनल पंजीकरण, सहस्थानीयकरण विश्लेषण और आरओआई हैंडलिंग।
सिद्धांत रूप में, एकल-कण ट्रैकिंग को एक-, दो- या तीन-आयामी प्रणालियों में किया जा सकता है। यहां, एकल-अणु विश्लेषण पाइपलाइन को 2 डी मोबाइल सिस्टम के अध्ययन के अनुरूप बनाया गया था। यह विकल्प मोबाइल फ्लोरोसेंट जांच पेश करने के लिए सरल प्रणालियों की उपलब्धता को प्रतिबिंबित करता है, जैसे कि प्लानर-समर्थित लिपिड बाईलेयर (एसएलबी)। इस तरह के लिपिड बाईलेयर सिस्टम आमतौर पर दो या दो से अधिक फॉस्फोलिपिड्स moieties से बने होते हैं, जहां थोक अंश एसएलबी (जैसे चरण और चिपचिपाहट) के प्रमुख भौतिक-रासायनिक मापदंडों को निर्धारित करता है, और मामूली अंश बायोमोलेक्यूल्स के लिए अनुलग्नक साइटें प्रदान करता है। इन अनुलग्नक साइटों को एविडिन के लिए बायोटिनिलेटेड फॉस्फोलिपिड्स- या स्ट्रेप्टाविडिन-आधारित प्रोटीन प्लेटफार्मों या हिस्टिडीन टैग 41 के साथ प्रोटीन प्लेटफार्मों के लिए निकल-एनटीए संयुग्मित फॉस्फोलिपिड्स हो सकते हैं। एसएलबी के लिए प्रोटीन को जोड़ने के लिए उपयुक्त मंच का विकल्प वैज्ञानिक प्रश्न पर निर्भर करता है। पाठक सफलतापूर्वक नियोजित रणनीतियों के उदाहरणों के लिए साहित्य 16,38,42 का उल्लेख कर सकते हैं। ओवरलैपिंग बिंदु प्रसार कार्यों से बचने के लिए नमूने में जांच का घनत्व पर्याप्त रूप से कम होना चाहिए; आमतौर पर, प्रति μm2 0.1 अणुओं से कम की सिफारिश की जाती है। एक उपयुक्त जांच घनत्व दिखाने वाले उदाहरण के लिए प्रतिनिधि परिणाम अनुभाग (विशेष रूप से, चित्रा 6) देखें। विश्लेषण विधि एकल फ्लोरोसेंटली लेबल वाले प्रोटीन अणुओं पर भी लागू होती है जो जीवित कोशिकाओं के प्लाज्मा झिल्ली में अलग हो जाती है।
SMFRET प्रयोगों का एक महत्वपूर्ण पहलू FRET जांच का उत्पादन और लक्षण वर्णन स्वयं है। एक FRET जोड़ी के लिए fluorophores का चयन करते समय, उनके Förster त्रिज्या अपेक्षित अंतर डाई दूरी 43 से मेल खाना चाहिए. फोटोब्लीचिंग के लिए प्रतिरोधी रंजक को पसंद किया जाता है क्योंकि वे लंबे समय तक निशान प्राप्त करते हैं। हालांकि, उन्नत ब्लीचिंग दरों के लिए, एक फ्लोरोफोर प्रजातियों का उपयोग स्टेपवाइज फोटोब्लीचिंग विश्लेषण के माध्यम से कोलोकलाइज्ड अणुओं से उत्पन्न होने वाली मल्टीएमिटर घटनाओं को पहचानने के लिए किया जा सकता है; प्रोटोकॉल अनुभाग में चरण 8.1.4 देखें। फ्लोरोफोर जोड़े साइट-विशेष रूप से और सहसंयोजक रूप से ब्याज के अणुओं से जुड़े होने चाहिए, इंट्रा- या इंटरमॉलिक्युलर फ्रेट जोड़े बनाते हैं।
अन्य आसानी से उपलब्ध तकनीकों के साथ smFRET के संयोजन से विवर्तन सीमा (STED44 के माध्यम से) से परे अपने स्थानिक संकल्प को बढ़ाया जा सकता है। यहां प्रस्तुत smFRET ट्रैकिंग एल्गोरिथ्म नए प्रयोगात्मक सेटिंग्स और मॉडल सिस्टम के लिए दृष्टिकोण की प्रयोज्यता को चौड़ा करता है। इसमें (i) मोबाइल बायोमोलेक्यूल्स के स्टोइकोमेट्री में गतिज परिवर्तन, (ii) मोबाइल बायोमोलेक्यूल्स का गतिशील संबंध, (iii) स्वतंत्र रूप से अलग-अलग अभिकारकों की एंजाइमेटिक प्रतिक्रियाओं की दर, और (iv) मोबाइल बायोमोलेक्यूल्स के संरचनात्मक परिवर्तनों के कैनेटीक्स का अध्ययन शामिल है। पहले दो उदाहरणों के लिए मॉडल सिस्टम की आवश्यकता होती है जो इंटरमॉलिक्युलर फ्रेट दिखाते हैं, यानी, दाता और स्वीकर्ता को ब्याज की बायोमोलेक्यूलर संस्थाओं को अलग करने के लिए संयुग्मित किया जाता है। उत्तरार्द्ध उदाहरण एक ही आणविक इकाई (इंट्रामोलेक्यूलर फ्रेट) के भीतर दाता और स्वीकर्ता को ले जाने वाले बायोसेंसर का उपयोग कर सकते हैं।
इंट्रामोलेक्यूलर FRET-आधारित सेंसर बायोमोलेक्यूल्स 1,2,3,4 के आंतरिक संरचनात्मक परिवर्तनों, अंतर्जात या बाहरी बल लोड (आणविक बल सेंसर 16) के कारण होने वाले संरचनात्मक परिवर्तनों, या नैनो-पर्यावरण में आयन सांद्रता जैसे कैल्शियम 45 और पीएच 46 में अंतर्दृष्टि प्रदान कर सकते हैं। . मॉडल प्रणाली और पसंदीदा एंकरिंग प्लेटफ़ॉर्म के आधार पर, इस तरह की smFRET घटनाओं को या तो 2D या 3D में ट्रैक किया जा सकता है: (i) smFRET घटनाओं के प्लानर ट्रैकिंग को प्लाज्मा झिल्ली के भीतर रिसेप्टर-लिगैंड इंटरैक्शन समय के परिमाणीकरण के लिए नियोजित किया जा सकता है, झिल्ली-लंगर वाले सिग्नल प्रवर्धन कैस्केड का संबंध, और सतह रिसेप्टर्स के स्टोइकोमेट्री परिवर्तन; (ii) एसएमएफआरईटी घटनाओं की मात्रा ट्रैकिंग का उपयोग जीवित कोशिकाओं या इन विट्रो पुनर्गठित प्रणालियों में किसी भी इंट्रा- या इंटरमॉलिक्युलर फ्रेट जांच के लिए किया जा सकता है।
smFRET ट्रैकिंग विधि मुख्य रूप से दिमाग में intramolecular FRET जांच के साथ विकसित किया गया था। इन जांचों में फ्लोरोसेंट लेबल की एक निश्चित और अच्छी तरह से ज्ञात संख्या होती है, एक तथ्य जो समूहीकृत और गलत तरीके से संश्लेषित (जैसे, अपूर्ण रूप से लेबल किए गए) अणुओं से डेटा को अस्वीकार करने के लिए शोषण किया गया था, साथ ही साथ जांच से जहां फ्लोरोफोर में से एक को फोटोब्लीच किया गया है। हालांकि, फ़िल्टरिंग चरणों को समायोजित करके, विधि को इंटरमॉलिक्युलर FRET जांच पर भी लागू किया जा सकता है। उदाहरण के लिए, केवल एक ही दाता और एक एकल स्वीकर्ता फ्लोरोफोर की विशेषता वाले अणुओं को स्वीकार करने के बजाय, कोई भी दाता और स्वीकर्ता रंजक के स्थानिक प्रक्षेपवक्रों की जांच कर सकता है और उदाहरण के लिए, दाता-स्वीकर्ता प्रक्षेपवक्रों को सह-अलग करने के लिए चुन सकता है।
चूंकि 3D-DAOSTORM एल्गोरिथ्म में उत्सर्जन बीम पथ में एक बेलनाकार लेंस के कारण अस्थिरता के माध्यम से ऑप्टिकल अक्ष के साथ सिग्नल की स्थिति का निर्धारण करने के लिए समर्थन है, 3 डी प्रयोगों को आसानी से विश्लेषण पाइपलाइन में एकीकृत किया जा सकता है। इस मामले में, स्वीकर्ता उत्तेजना पर स्वीकर्ता संकेत स्टोइकोइमेट्री और अक्षीय स्थिति को निर्धारित करने के लिए काम करेगा। विश्लेषण सॉफ़्टवेयर को स्वचालन और फ़िल्टरिंग योजनाओं की अपनी बड़ी डिग्री का उपयोग करके स्थिर जांच की विशेषता वाले प्रयोगों से डेटा का मूल्यांकन करने के लिए भी नियोजित किया जा सकता है। वास्तव में, जेल-चरण बाईलेयर 38 पर स्थिर होलिडे जंक्शनों से smFRET दक्षता डेटासेट का विश्लेषण सॉफ़्टवेयर के शुरुआती संस्करण का उपयोग करके किया गया था।
Disclosures
The authors have nothing to disclose.
Acknowledgements
इस काम को ऑस्ट्रियाई विज्ञान कोष (एफडब्ल्यूएफ) परियोजनाओं P30214-N36, P32307-B, और वियना विज्ञान और प्रौद्योगिकी कोष (WWTF) LS13-030 द्वारा समर्थित किया गया था।
Materials
| 1,2-dioleoyl-sn-glycero-3-[(N-(5-amino-1-carboxypentyl)iminodiacetic acid)succinyl] (nickel salt) (Ni-NTA-DOGS) | Avanti Polar Lipids | 790404P | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Avanti Polar Lipids | 850375P | |
| 1-palmitoyl-2-oleoyl-glycero-3-phosphocholine (POPC) | Avanti Polar Lipids | 850457P | |
| α Plan-FLUAR 100x/1.45 oil objective | Zeiss | 000000-1084-514 | |
| Axio Observer microscope body | Zeiss | ||
| Bandpass filter | Chroma Technology Corp | ET570/60m | donor emission filter |
| Bandpass filter | Chroma Technology Corp | ET675/50m | acceptor emission filter |
| conda-forge | conda-forge community | community-maintaned Python package repository for Anaconda/miniconda | |
| Coverslips 60 mm x 24 mm #1.5 | MENZEL | ||
| Dichroic mirror | Semrock Inc | FF640-FDi01-25×36 | separation of donor and acceptor emission |
| Dichroic mirror (quad band) | Semrock Inc | Di01-R405/488/532/635-25×36 | separation of excitation and emission light |
| DPBS | Sigma-Aldrich | D8537 | |
| FCS | Sigma-Aldrich | F7524 | for imaging buffer |
| fret-analysis | Schütz group at TU Wien | Python package for smFRET data analysis; version 3 | |
| Fura-2 AM | Thermo Fisher Scientific | 11524766 | |
| HBSS | Sigma-Aldrich | H8264 | for imaging buffer |
| iBeam Smart 405-S 405 nm laser | Toptica Photonics AG | ||
| iXon Ultra 897 EMCCD camera | Andor Technology Ltd | ||
| Lab-Tek chambers (8 wells) | Thermo Fisher Scientific | 177402PK | for sample preparation and imaging |
| Millenia Prime 532 nm laser | Spectra Physics | ||
| miniconda | Anaconda Inc. | Python 3 distribution. Min. version: 3.7 | |
| Monovalent streptavidin (plasmids for bacterial expression) | Addgene | 20860 & 20859 | |
| OBIS 640 nm laser | Coherent Inc | 1185055 | |
| Optosplit II | Cairn Research | ||
| Ovalbumin | Sigma-Aldrich | A5253 | for imaging buffer |
| Plasma cleaner | Harrick Plasma | PDC-002 | |
| sdt-python | Schütz group at TU Wien | Python library for data analysis; version 17 | |
| TetraSpek bead size kit | Thermo Fisher Scientific | T14792 | Randomly distributed, immobilized fiducial markers for image registration |
| USC500TH Ultrasound bath | VWR | for SUV formation |
References
- McKinney, S. A., Déclais, A. -. C., Lilley, D. M. J., Ha, T. Structural dynamics of individual holliday junctions. Nature Structural Biology. 10 (2), 93-97 (2002).
- Wang, S., Vafabakhsh, R., Borschel, W. F., Ha, T., Nichols, C. G. Structural dynamics of potassium-channel gating revealed by single-molecule FRET. Nature Structural & Molecular Biology. 23 (1), 31-36 (2015).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2016).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), 235 (2018).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47 (1), 819-846 (1978).
- Wu, P. G., Brand, L. Resonance energy transfer: Methods and applications. Analytical Biochemistry. 218 (1), 1-13 (1994).
- Qiao, Y., Luo, Y., Long, N., Xing, Y., Tu, J. Single-molecular förster resonance energy transfer measurement on structures and interactions of biomolecules. Micromachines. 12 (5), 492 (2021).
- Malkusch, N., Dörfler, T., Nagy, J., Eilert, T., Michaelis, J. smFRET experiments of the RNA polymerase II transcription initiation complex. Methods. 120, 115-124 (2017).
- Lee, J. -. B., et al. Single-molecule views of MutS on mismatched DNA. DNA repair. 20, 82-93 (2014).
- Phelps, C., Israels, B., Jose, D., Marsh, M. C., von Hippel, P. H., Marcus, A. H. Using microsecond single-molecule FRET to determine the assembly pathways of T4 ssDNA binding protein onto model DNA replication forks. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), E3612-E3621 (2017).
- Deindl, S., Zhuang, X. Monitoring conformational dynamics with single-molecule fluorescence energy transfer: Applications in nucleosome remodeling. Methods in Enzymology. 513, 59-86 (2012).
- Crawford, D. J., Hoskins, A. A., Friedman, L. J., Gelles, J., Moore, M. J. Single-molecule colocalization FRET evidence that spliceosome activation precedes stable approach of 5′ splice site and branch site. Proceedings of the National Academy of Sciences of the United States of America. 110 (17), 6783-6788 (2013).
- Wang, Y., Xiao, M., Li, Y. Heterogeneity of single molecule FRET signals reveals multiple active ribosome subpopulations. Proteins. 82 (1), 1-9 (2014).
- Mori, T., Vale, R. D., Tomishige, M. How kinesin waits between steps. Nature. 450 (7170), 750-754 (2007).
- Huppa, J. B., et al. TCR-peptide-MHC interactions in situ show accelerated kinetics and increased affinity. Nature. 463 (7283), 963-967 (2010).
- Göhring, J., et al. Temporal analysis of T-cell receptor-imposed forces via quantitative single molecule FRET measurements. Nature Communications. 12 (1), 2502 (2021).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669 (2018).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences of the United States of America. 101 (24), 8936-8941 (2004).
- Sakon, J. J., Weninger, K. R. Detecting the conformation of individual proteins in live cells. Nature Methods. 7 (3), 203-205 (2010).
- McCann, J. J., Choi, U. B., Zheng, L., Weninger, K., Bowen, M. E. Optimizing methods to recover absolute FRET efficiency from immobilized single molecules. Biophysical Journal. 99 (3), 961-970 (2010).
- Lee, N. K., et al. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophysical Journal. 88 (4), 2939-2953 (2005).
- Asher, W. B., et al. Single-molecule FRET imaging of GPCR dimers in living cells. Nature Methods. 18 (4), 397-405 (2021).
- Joo, C., Ha, T. . Single-molecule FRET with total internal reflection microscopy. (12), 1223-1237 (2012).
- Joo, C., Ha, T. Objective-type total internal reflection microscopy (excitation) for single-molecule FRET. Cold Spring Harbor Protocols. 11, 1189-1191 (2012).
- Joo, C., Ha, T. Objective-type total internal reflection microscopy (emission) for single-molecule FRET. Cold Spring Harbor Protocols. 11, 1192-1194 (2012).
- Allan, D. B., Caswell, T., van der Wel, C. M., Dimiduk, T. . Soft-matter/pims: PIMS v0.5. , (2020).
- Anaconda Inc. . Miniconda. , (2021).
- conda-forge community. . The conda-forge project: community-based software distribution built on the conda package format and ecosystem. , (2015).
- . . JupyterLab Contributors Notebooks – JupyterLab documentation. , (2021).
- Babcock, H., Sigal, Y. M., Zhuang, X. A high-density 3D localization algorithm for stochastic optical reconstruction microscopy. Optical Nanoscopy. 1 (6), (2012).
- Gao, Y., Kilfoil, M. L. Accurate detection and complete tracking of large populations of features in three dimensions. Optics Express. 17 (6), 4685 (2009).
- Schrangl, L. . sdt-python: Python library for fluorescence microscopy data analysis (v17.1). , (2021).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. Journal of Colloid and Interface Science. 179 (1), 298-310 (1996).
- Preibisch, S., Saalfeld, S., Schindelin, J., Tomancak, P. Software for bead-based registration of selective plane illumination microscopy data. Nature Methods. 7 (6), 418-419 (2010).
- Bradski, G. The OpenCV library. Dr. Dobb’s Journal: Software Tools for the Professional Programmer. 25 (11), 120-123 (2000).
- Allan, D. B., Caswell, T., Keim, N. C., van der Wel, C. M., Verweij, R. W. Soft-matter/trackpy: Trackpy v0.5.0. Zenodo. , 4682814 (2021).
- Killick, R., Fearnhead, P., Eckley, I. A. Optimal detection of changepoints with a linear computational cost. Journal of the American Statistical Association. 107 (500), 1590-1598 (2012).
- Schrangl, L., Göhring, J., Schütz, G. J. Kinetic analysis of single molecule FRET transitions without trajectories. The Journal of Chemical Physics. 148 (12), 123328 (2018).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Schrangl, L. Single-molecule FRET analysis software (3.0). Zenodo. , (2021).
- Nye, J. A., Groves, J. T. Kinetic control of histidine-tagged protein surface density on supported lipid bilayers. Langmuir. 24 (8), 4145-4149 (2008).
- Platzer, R., et al. Unscrambling fluorophore blinking for comprehensive cluster detection via photoactivated localization microscopy. Nature Communications. 11 (1), 4993 (2020).
- Johnson, I., Spence, M. . The molecular probes handbook: A guide to fluorescent probes and labeling technologies. , (2010).
- Szalai, A. M., et al. Super-resolution imaging of energy transfer by intensity-based STED-FRET. Nano Letters. 21 (5), 2296-2303 (2021).
- Miyawaki, A., et al. Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature. 388 (6645), 882-887 (1997).
- Zhai, B., Zhai, S., Hao, R., Xu, J., Liu, Z. A FRET-based two-photon probe for in vivo tracking of pH during a traumatic brain injury process. New Journal of Chemistry. 43 (43), 17018-17022 (2019).

