Geautomatiseerde tweedimensionale spatiotemporale analyse van mobiele fret-sondes met één molecuul
Summary
Dit artikel presenteert een methode voor spatiotemporale analyse van mobiele, single-molecule Förster resonantie energieoverdracht (smFRET)-gebaseerde probes met behulp van widefield fluorescentiemicroscopie. De nieuw ontwikkelde softwaretoolkit maakt het mogelijk om smFRET-tijdsporen van bewegende sondes te bepalen, inclusief de juiste FRET-efficiëntie en de moleculaire posities, als functies van de tijd.
Abstract
Single-molecule Förster resonantie energieoverdracht (smFRET) is een veelzijdige techniek die rapporteert over afstanden in het sub-nanometer tot nanometer bereik. Het is gebruikt in een breed scala van biofysische en moleculair biologische experimenten, waaronder de meting van moleculaire krachten, karakterisering van conformatiedynamiek van biomoleculen, observatie van intracellulaire colocalisatie van eiwitten en bepaling van receptor-ligand interactietijden. In een widefield-microscopieconfiguratie worden experimenten meestal uitgevoerd met behulp van oppervlakte-geïmmobiliseerde sondes. Hier wordt een methode gepresenteerd die single-molecule tracking combineert met alternating excitation (ALEX) smFRET-experimenten, waardoor smFRET-tijdsporen van oppervlaktegebonden, maar mobiele probes in plasmamembranen of door glas ondersteunde lipide bilayers kunnen worden verkregen. Voor de analyse van geregistreerde gegevens werd een geautomatiseerde, open-source softwareverzameling ontwikkeld ter ondersteuning van (i) de lokalisatie van fluorescerende signalen, (ii) single-particle tracking, (iii) bepaling van FRET-gerelateerde grootheden inclusief correctiefactoren, (iv) strenge verificatie van smFRET-sporen en (v) intuïtieve presentatie van de resultaten. De gegenereerde gegevens kunnen gemakkelijk worden gebruikt als input voor verdere verkenning via gespecialiseerde software, bijvoorbeeld voor de beoordeling van het diffusionele gedrag van sondes of het onderzoek van FRET-overgangen.
Introduction
Förster resonantie energieoverdracht (FRET) is een belangrijke drijfveer geweest in moleculair biologisch en biofysisch onderzoek, omdat het het onderzoek van processen met sub-nanometer resolutie mogelijk maakt. Aangezien de efficiëntie van de energieoverdracht tussen donor- en acceptorfluooforen sterk afhangt van de inter-kleurstofafstand in het subnanometer-nanometer-nanometerbereik, is het effectief gebruikt als een spectroscopische liniaal om statische en dynamische conformatie van biomoleculen te onderzoeken1,2,3,4. Bovendien is het FRET-fenomeen op grote schaal gebruikt voor colocalisatiestudies van membraangeassocieerde en intracellulaire eiwitten op bulkniveau5,6. In de afgelopen twee decennia werd de methode aangepast voor het monitoren van smFRET-gebeurtenissen7, wat hielp om de temporele en ruimtelijke resolutie aanzienlijk te verhogen en zelfs zeldzame subpopulaties in heterogene monsters op te lossen. Uitgerust met deze technieken werden unieke inzichten verkregen in de dynamiek van moleculaire machines, zoals de transcriptverwerkingssnelheid van RNA-polymerase II8, replicatiesnelheid van DNA-polymerasen9,10, nucleosoomtranslocatiesnelheid11, transcriptsplitsing en stagnatiesnelheid van geassembleerde spliceosomen12, de activiteit van ribosomale subpopulaties13 en de loopsnelheid van kinesinemotoren14 , om er maar een paar te noemen. Receptor-ligand interactie duur15 en moleculaire krachten16 zijn gekwantificeerd.
Op intensiteit gebaseerde smFRET-studies vertrouwen doorgaans op gesensibiliseerde emissie om de FRET-efficiëntie te meten: een bundelsplitser in het emissiepad scheidt ruimtelijk licht afkomstig van donor- en acceptorfluooforen bij donorexcitatie, waardoor de kwantificering van individuele fluorescentie-intensiteiten mogelijk is. Het rendement kan vervolgens worden berekend als de fractie van fotonen die door de acceptor wordt uitgezonden ten opzichte van het totale aantal fotonen17. Bovendien maakt acceptor excitatie na donor excitatie (ALEX) het mogelijk om de stoichiometrie van de FRET-gebeurtenissen te meten, wat helpt bij het onderscheid tussen echte lage FRET-signalen van signalen die bijvoorbeeld voortkomen uit sondes met een fotogebleekte acceptorfluoofoor18.
Single-molecule FRET-experimenten worden vaak op twee manieren uitgevoerd. Eerst wordt een klein gebied in het monstervolume belicht met behulp van een confocale microscoop. Enkele sondemoleculen in oplossing worden opgewonden wanneer ze toevallig diffunderen binnen het focale volume. Met deze techniek kunnen snelle detectoren voor het tellen van fotonen worden gebruikt, waardoor een tijdresolutie van minder dan een microseconde mogelijk is. Ten tweede worden sondes specifiek geïmmobiliseerd op oppervlakken en bewaakt via widefield-microscopie, vaak met behulp van totale interne reflectie (TIR) -configuratie om achtergrondfluorescentie te minimaliseren. Probe immobilisatie zorgt voor veel langere opnametijden dan het gebruik van de eerste benadering. Bovendien maakt het grotere gezichtsveld het mogelijk om meerdere sondes parallel te bewaken. De behoefte aan een camera maakt deze methode traag in vergelijking met de hierboven beschreven methode. De tijdresolutie is beperkt tot het milliseconde tot tweede bereik.
Als lange tijdsporen nodig zijn, bijvoorbeeld voor het bestuderen van dynamische processen op een milliseconde tot tweede tijdschaal, is de eerste methode niet van toepassing, omdat de fluorescentie-uitbarstingen meestal te kort zijn. De tweede benadering faalt wanneer immobilisatie niet haalbaar is, bijvoorbeeld in live-celexperimenten met sondes die zich in het celmembraan verspreiden. Bovendien is waargenomen dat biologische modelsystemen hun respons dramatisch kunnen variëren, afhankelijk van de mobiliteit van het gecontacteerde oppervlak16.
Hoewel in het verleden gecombineerde smFRET- en single-particle tracking-experimenten met mobiele FRET-sondes zijn uitgevoerd19, is er geen openbaar beschikbare software voor de evaluatie van de gegevens. Dit leidde tot de ontwikkeling van een nieuw analyseplatform, dat de bepaling van meerdere eigenschappen van mobiele fluorescerende sondes mogelijk maakt, waaronder smFRET-efficiëntie en stoichiometrie, posities met subpixelnauwkeurigheid en fluorescentie-intensiteiten als functies van de tijd. Methoden voor het filteren van de resulterende sporen door stapsgewijs bleekgedrag, nearest-neighbor afstanden, emissie-intensiteiten en andere eigenschappen te onderzoeken, werden vastgesteld om uitsluitend correct gesynthetiseerde en functionele single-probe moleculen te kiezen. De software ondersteunt ook experimentele en analytische technieken die onlangs zijn overeengekomen in een multilaboratoriumstudie om betrouwbare, kwantitatieve smFRET-gegevens te produceren17. In het bijzonder houdt de implementatie zich aan de gevalideerde procedures voor de berekening van FRET-efficiëntie en stoichiometrie. Fluorescentie-intensiteiten bij donorexcitatie in het donoremissiekanaal IDD en het acceptoremissiekanaal IDA worden gebruikt voor de berekening van de schijnbare FRET-efficiëntie-Eapp met Eq (1).
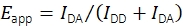 (1)
(1)
Met behulp van de fluorescentie-intensiteit in het acceptoremissiekanaal bij acceptor-excitatie IAA wordt de schijnbare stoichiometrie berekend met behulp van Eq (2).
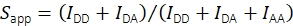 (2)
(2)
De FRET-efficiëntie E en de stoichiometrie S kunnen worden afgeleid van Eapp en Sapp door rekening te houden met vier correctiefactoren.
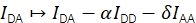
α beschrijft de lekkage van donorfluorescentie in het acceptoremissiekanaal en kan worden bepaald met behulp van een monster dat alleen donorfluoroforen bevat of door delen van trajecten te analyseren waar de acceptor is gebleekt. δ corrigeert voor de directe excitatie van de acceptor door de donor excitatie lichtbron en kan worden gemeten met behulp van een monster met alleen acceptor fluoroforen of door het analyseren van delen van trajecten waar de donor is gebleekt.
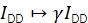 .
.
γ schaalt de ID om divergerende detectie-efficiënties in donor- en acceptoremissiekanalen en verschillende kwantumefficiënties van de fluoroforen te corrigeren. De factor kan worden berekend door de toename van de donorintensiteit te analyseren bij het bleken van de acceptor in trajecten met hoge FRET-efficiëntie20 of door een monster met meerdere discrete FRET-toestanden te bestuderen.
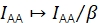
β schaalt IAA om te corrigeren voor ongelijksoortige efficiëntie van donor- en acceptorexcitatie. Als γ werd bepaald via een analyse van het bleken van de acceptor, kon β worden berekend uit een monster van de bekende donor-acceptorverhouding21. Anders levert de multi-state FRET-steekproef ook β op.
Samen maken de correcties het mogelijk om de gecorrigeerde FRET-efficiëntie te berekenen met behulp van Eq (3).
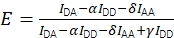 (3)
(3)
en de gecorrigeerde stoichiometrie met Eq (4).
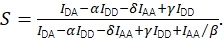 (4)
(4)
Idealiter geeft de gecorrigeerde stoichiometrie voor een 1:1 donor-to-acceptor ratio S = 0,5. In de praktijk zorgt een verminderde signaal-ruisverhouding voor een spreiding van de gemeten waarden van S, waardoor de discriminatie van donor-only signalen (S = 1) en acceptor-only signalen (S = 0) wordt belemmerd. De resulterende tijdsporen kunnen worden gebruikt als input voor een meer gedetailleerde analyse van de trajecten met één molecuul om informatie te verkrijgen zoals spatiotemporale krachtprofielen16, de mobiliteit van de single-molecule events22 of overgangskinetiek tussen verschillende toestanden1.
Het volgende protocol beschrijft experimentele parameters en procedures voor smFRET-trackingexperimenten, evenals het werkingsprincipe achter data-analyse met behulp van de nieuw ontwikkelde softwaresuite. Voor het verzamelen van experimentele gegevens wordt aanbevolen om een microscopie-opstelling te gebruiken die aan de volgende vereisten voldoet: i) het vermogen om de emissie van enkele kleurstofmoleculen te detecteren; ii) widefieldverlichting: met name voor experimenten met levende cellen wordt een configuratie van de totale interne reflectie (TIR23,24,25) aanbevolen; iii) ruimtelijke scheiding van emissielicht volgens golflengte, zodat donor- en acceptorfluorescentie wordt geprojecteerd op verschillende regio’s van dezelfde camerachip25 of verschillende camera’s; iv) modulatie van lichtbronnen voor donor- en acceptorexcitatie met millisecondeprecisie, bijvoorbeeld met behulp van direct modulalabele lasers of modulatie via acousto-optische modulatoren. Dit maakt stroboscopische verlichting mogelijk om fotobleaching van fluoroforen te minimaliseren, evenals afwisselende excitatie om stoichiometrieën te bepalen; v) uitvoer van één bestand per opgenomen beeldreeks in een formaat dat kan worden gelezen door het PIMS Python-pakket26. In het bijzonder worden TIFF-bestanden met meerdere pagina’s ondersteund.
Protocol
Representative Results
Discussion
Dit artikel beschrijft een pijplijn voor de geautomatiseerde registratie en kwantitatieve analyse van smFRET-gegevens afkomstig van mobiele maar aan het oppervlak vastgebonden sondemoleculen. Het vormt een aanvulling op de twee overheersende benaderingen van smFRET-experimenten, waarbij ofwel oppervlakte-geïmmobiliseerde sondes of sondes diffunderen in oplossing in en uit een confocale excitatievolume17. Het biedt de juiste FRET-efficiëntie en de moleculaire posities als functie van de tijd. Het kan daarom worden gebruikt als input voor gespecialiseerde analyseprogramma’s, bijvoorbeeld om overgangskinetiek1, FRET-histogrammen39 of tweedimensionale diffusie22 te kwantificeren.
De software wordt uitgebracht onder een gratis en open-source licentie die is goedgekeurd door het Open Source Initiative en die de gebruiker het eeuwigdurende recht op gratis gebruik, wijziging en herdistributie verleent. Github is gekozen als ontwikkelings- en distributieplatform om het zo gemakkelijk mogelijk te maken om de software te verkrijgen en deel te nemen aan het ontwikkelingsproces door bugs te melden of code40 bij te dragen. Geschreven in Python, is de software niet afhankelijk van propriëtaire componenten. De keuze voor Jupyter-notebooks als gebruikersinterfaces vergemakkelijkt de inspectie van gegevens bij elke analysestap en maakt het mogelijk om de pijplijn specifiek voor het experimentele systeem aan te passen en uit te breiden. De sdt-python library32 dient als basis en implementeert functionaliteit om fluorescentiemicroscopiegegevens te evalueren, zoals lokalisatie van één molecuul, diffusieanalyse, fluorescentie-intensiteitsanalyse, kleurkanaalregistratie, colocalisatieanalyse en ROI-verwerking.
In principe kan single-particle tracking worden uitgevoerd in een-, twee- of driedimensionale systemen. Hier werd de single-molecule analysepijplijn afgestemd op de studie van 2D mobiele systemen. Deze keuze weerspiegelt de beschikbaarheid van eenvoudige systemen, zoals planair ondersteunde lipide bilayers (SLBs), om mobiele fluorescerende sondes te presenteren. Dergelijke lipide dubbellaagssystemen zijn meestal samengesteld uit twee of meer fosfolipiden moieties, waarbij de bulkfractie de belangrijkste fysisch-chemische parameters van de SLB bepaalt (zoals fase en viscositeit), en de kleine fractie zorgt voor hechtingsplaatsen voor biomoleculen. Deze hechtingsplaatsen kunnen gebiotinyleerde fosfolipiden zijn voor op avidine of streptavidin gebaseerde eiwitplatforms of nikkel-NTA geconjugeerde fosfolipiden voor eiwitplatforms met histidinetags41. De keuze van het geschikte platform voor het koppelen van eiwitten aan de SLB hangt af van de wetenschappelijke vraag. Lezers kunnen de literatuur16,38,42 raadplegen voor voorbeelden van succesvol toegepaste strategieën. De dichtheid van sondes in het monster moet voldoende laag zijn om overlappende puntspreidingsfuncties te voorkomen; doorgaans worden minder dan 0,1 moleculen per μm2 aanbevolen. Zie de sectie representatieve resultaten (met name figuur 6) voor een voorbeeld met een geschikte sondedichtheid. De analysemethode is ook toepasbaar op enkele fluorescerend gelabelde eiwitmoleculen die diffunderen in het plasmamembraan van levende cellen.
Een cruciaal aspect van smFRET-experimenten is de productie en karakterisering van de FRET-sondes zelf. Bij het kiezen van fluoroforen voor een FRET-paar moet hun Förster-straal overeenkomen met de verwachte interkleurafstanden43. Kleurstoffen die bestand zijn tegen fotobleaching hebben de voorkeur omdat ze lange tijd sporen opleveren. Voor verhoogde bleeksnelheden kan echter één fluorofoorsoort worden gebruikt om multiemittergebeurtenissen afkomstig van geglokaliseerde moleculen te herkennen via stapsgewijze fotobleachinganalyse; zie stap 8.1.4 in de protocolsectie. Fluorofoorparen moeten plaatsspecifiek en covalent aan de betreffende moleculen worden bevestigd en intra- of intermoleculaire FRET-paren vormen.
Het combineren van smFRET met andere direct beschikbare technieken kan de ruimtelijke resolutie verhogen tot boven de diffractielimiet (via STED44). Het hier gepresenteerde smFRET-trackingalgoritme verbreedt de toepasbaarheid van de aanpak op nieuwe experimentele instellingen en modelsystemen. Dit omvat studies van (i) kinetische veranderingen in de stoichiometrie van mobiele biomoleculen, (ii) dynamische associatie van mobiele biomoleculen, (iii) de snelheid van enzymatische reacties van vrij diffunderende reactanten, en (iv) de kinetiek van conformationele veranderingen van mobiele biomoleculen. De eerste twee voorbeelden vereisen modelsystemen die intermoleculaire FRET laten zien, d.w.z. donor en acceptor worden geconjugeerd om biomoleculaire entiteiten van belang te scheiden. Deze laatste voorbeelden kunnen gebruik maken van biosensoren die donor en acceptor binnen dezelfde moleculaire entiteit dragen (intramoleculaire FRET).
Intramoleculaire FRET-gebaseerde sensoren kunnen inzicht geven in intrinsieke conformatieveranderingen van biomoleculen1,2,3,4, conformationele veranderingen veroorzaakt door endogene of externe krachtbelasting (moleculaire krachtsensoren16) of ionenconcentraties in de nano-omgeving zoals calcium45 en pH46 . Afhankelijk van het modelsysteem en het gewenste verankeringsplatform kunnen dergelijke smFRET-gebeurtenissen in 2D of 3D worden gevolgd: (i) planaire tracking van smFRET-gebeurtenissen kan worden gebruikt voor de kwantificering van receptor-ligandinteractietijden binnen een plasmamembraan, de associatie van membraan-verankerde signaalversterkingscascades en de stoichiometrieveranderingen van oppervlaktereceptoren; ii) volumetracking van smFRET-voorvallen kan worden gebruikt voor intra- of intermoleculaire FRET-sondes in levende cellen of in in vitro gereconstitueerde systemen.
De smFRET-trackingmethode is voornamelijk ontwikkeld met intramoleculaire FRET-sondes in gedachten. Deze sondes hebben een vast en bekend aantal fluorescerende labels, een feit dat werd gebruikt om gegevens van geagglomereerde en onjuist gesynthetiseerde (bijv. Onvolledig gelabelde) moleculen af te wijzen, evenals van sondes waar een van de fluoroforen is gefotobleekt. Door de filterstappen aan te passen, kan de methode echter ook worden toegepast op intermoleculaire FRET-sondes. In plaats van alleen moleculen met een enkele donor en een enkele acceptorfluoofoor te accepteren, zou men bijvoorbeeld de ruimtelijke trajecten van donor- en acceptorkleurstoffen kunnen onderzoeken en bijvoorbeeld kunnen selecteren voor co-diffusie van donor-acceptortrajecten.
Omdat het 3D-DAOSTORM-algoritme ondersteuning biedt voor het bepalen van de positie van een signaal langs de optische as via het astigmatisme als gevolg van een cilindrische lens in het emissiestraalpad, kunnen 3D-experimenten eenvoudig worden geïntegreerd in de analysepijplijn. In dit geval zou het acceptorsignaal bij acceptorexcitatie dienen om de stoichiometrie en de axiale positie te bepalen. De analysesoftware kan ook worden gebruikt om gegevens van experimenten met geïmmobiliseerde sondes te evalueren door gebruik te maken van de grote mate van automatisering en filterschema’s. In feite werden smFRET-efficiëntiedatasets van Holliday-juncties geïmmobiliseerd op gel-fase bilayers38 geanalyseerd met behulp van een vroege versie van de software.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dit werk werd ondersteund door de projecten P30214-N36, P32307-B van het Oostenrijkse Wetenschapsfonds (FWF) en door het Vienna Science and Technology Fund (WWTF) LS13-030.
Materials
| 1,2-dioleoyl-sn-glycero-3-[(N-(5-amino-1-carboxypentyl)iminodiacetic acid)succinyl] (nickel salt) (Ni-NTA-DOGS) | Avanti Polar Lipids | 790404P | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Avanti Polar Lipids | 850375P | |
| 1-palmitoyl-2-oleoyl-glycero-3-phosphocholine (POPC) | Avanti Polar Lipids | 850457P | |
| α Plan-FLUAR 100x/1.45 oil objective | Zeiss | 000000-1084-514 | |
| Axio Observer microscope body | Zeiss | ||
| Bandpass filter | Chroma Technology Corp | ET570/60m | donor emission filter |
| Bandpass filter | Chroma Technology Corp | ET675/50m | acceptor emission filter |
| conda-forge | conda-forge community | community-maintaned Python package repository for Anaconda/miniconda | |
| Coverslips 60 mm x 24 mm #1.5 | MENZEL | ||
| Dichroic mirror | Semrock Inc | FF640-FDi01-25×36 | separation of donor and acceptor emission |
| Dichroic mirror (quad band) | Semrock Inc | Di01-R405/488/532/635-25×36 | separation of excitation and emission light |
| DPBS | Sigma-Aldrich | D8537 | |
| FCS | Sigma-Aldrich | F7524 | for imaging buffer |
| fret-analysis | Schütz group at TU Wien | Python package for smFRET data analysis; version 3 | |
| Fura-2 AM | Thermo Fisher Scientific | 11524766 | |
| HBSS | Sigma-Aldrich | H8264 | for imaging buffer |
| iBeam Smart 405-S 405 nm laser | Toptica Photonics AG | ||
| iXon Ultra 897 EMCCD camera | Andor Technology Ltd | ||
| Lab-Tek chambers (8 wells) | Thermo Fisher Scientific | 177402PK | for sample preparation and imaging |
| Millenia Prime 532 nm laser | Spectra Physics | ||
| miniconda | Anaconda Inc. | Python 3 distribution. Min. version: 3.7 | |
| Monovalent streptavidin (plasmids for bacterial expression) | Addgene | 20860 & 20859 | |
| OBIS 640 nm laser | Coherent Inc | 1185055 | |
| Optosplit II | Cairn Research | ||
| Ovalbumin | Sigma-Aldrich | A5253 | for imaging buffer |
| Plasma cleaner | Harrick Plasma | PDC-002 | |
| sdt-python | Schütz group at TU Wien | Python library for data analysis; version 17 | |
| TetraSpek bead size kit | Thermo Fisher Scientific | T14792 | Randomly distributed, immobilized fiducial markers for image registration |
| USC500TH Ultrasound bath | VWR | for SUV formation |
References
- McKinney, S. A., Déclais, A. -. C., Lilley, D. M. J., Ha, T. Structural dynamics of individual holliday junctions. Nature Structural Biology. 10 (2), 93-97 (2002).
- Wang, S., Vafabakhsh, R., Borschel, W. F., Ha, T., Nichols, C. G. Structural dynamics of potassium-channel gating revealed by single-molecule FRET. Nature Structural & Molecular Biology. 23 (1), 31-36 (2015).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2016).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), 235 (2018).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47 (1), 819-846 (1978).
- Wu, P. G., Brand, L. Resonance energy transfer: Methods and applications. Analytical Biochemistry. 218 (1), 1-13 (1994).
- Qiao, Y., Luo, Y., Long, N., Xing, Y., Tu, J. Single-molecular förster resonance energy transfer measurement on structures and interactions of biomolecules. Micromachines. 12 (5), 492 (2021).
- Malkusch, N., Dörfler, T., Nagy, J., Eilert, T., Michaelis, J. smFRET experiments of the RNA polymerase II transcription initiation complex. Methods. 120, 115-124 (2017).
- Lee, J. -. B., et al. Single-molecule views of MutS on mismatched DNA. DNA repair. 20, 82-93 (2014).
- Phelps, C., Israels, B., Jose, D., Marsh, M. C., von Hippel, P. H., Marcus, A. H. Using microsecond single-molecule FRET to determine the assembly pathways of T4 ssDNA binding protein onto model DNA replication forks. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), E3612-E3621 (2017).
- Deindl, S., Zhuang, X. Monitoring conformational dynamics with single-molecule fluorescence energy transfer: Applications in nucleosome remodeling. Methods in Enzymology. 513, 59-86 (2012).
- Crawford, D. J., Hoskins, A. A., Friedman, L. J., Gelles, J., Moore, M. J. Single-molecule colocalization FRET evidence that spliceosome activation precedes stable approach of 5′ splice site and branch site. Proceedings of the National Academy of Sciences of the United States of America. 110 (17), 6783-6788 (2013).
- Wang, Y., Xiao, M., Li, Y. Heterogeneity of single molecule FRET signals reveals multiple active ribosome subpopulations. Proteins. 82 (1), 1-9 (2014).
- Mori, T., Vale, R. D., Tomishige, M. How kinesin waits between steps. Nature. 450 (7170), 750-754 (2007).
- Huppa, J. B., et al. TCR-peptide-MHC interactions in situ show accelerated kinetics and increased affinity. Nature. 463 (7283), 963-967 (2010).
- Göhring, J., et al. Temporal analysis of T-cell receptor-imposed forces via quantitative single molecule FRET measurements. Nature Communications. 12 (1), 2502 (2021).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669 (2018).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences of the United States of America. 101 (24), 8936-8941 (2004).
- Sakon, J. J., Weninger, K. R. Detecting the conformation of individual proteins in live cells. Nature Methods. 7 (3), 203-205 (2010).
- McCann, J. J., Choi, U. B., Zheng, L., Weninger, K., Bowen, M. E. Optimizing methods to recover absolute FRET efficiency from immobilized single molecules. Biophysical Journal. 99 (3), 961-970 (2010).
- Lee, N. K., et al. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophysical Journal. 88 (4), 2939-2953 (2005).
- Asher, W. B., et al. Single-molecule FRET imaging of GPCR dimers in living cells. Nature Methods. 18 (4), 397-405 (2021).
- Joo, C., Ha, T. . Single-molecule FRET with total internal reflection microscopy. (12), 1223-1237 (2012).
- Joo, C., Ha, T. Objective-type total internal reflection microscopy (excitation) for single-molecule FRET. Cold Spring Harbor Protocols. 11, 1189-1191 (2012).
- Joo, C., Ha, T. Objective-type total internal reflection microscopy (emission) for single-molecule FRET. Cold Spring Harbor Protocols. 11, 1192-1194 (2012).
- Allan, D. B., Caswell, T., van der Wel, C. M., Dimiduk, T. . Soft-matter/pims: PIMS v0.5. , (2020).
- Anaconda Inc. . Miniconda. , (2021).
- conda-forge community. . The conda-forge project: community-based software distribution built on the conda package format and ecosystem. , (2015).
- . . JupyterLab Contributors Notebooks – JupyterLab documentation. , (2021).
- Babcock, H., Sigal, Y. M., Zhuang, X. A high-density 3D localization algorithm for stochastic optical reconstruction microscopy. Optical Nanoscopy. 1 (6), (2012).
- Gao, Y., Kilfoil, M. L. Accurate detection and complete tracking of large populations of features in three dimensions. Optics Express. 17 (6), 4685 (2009).
- Schrangl, L. . sdt-python: Python library for fluorescence microscopy data analysis (v17.1). , (2021).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. Journal of Colloid and Interface Science. 179 (1), 298-310 (1996).
- Preibisch, S., Saalfeld, S., Schindelin, J., Tomancak, P. Software for bead-based registration of selective plane illumination microscopy data. Nature Methods. 7 (6), 418-419 (2010).
- Bradski, G. The OpenCV library. Dr. Dobb’s Journal: Software Tools for the Professional Programmer. 25 (11), 120-123 (2000).
- Allan, D. B., Caswell, T., Keim, N. C., van der Wel, C. M., Verweij, R. W. Soft-matter/trackpy: Trackpy v0.5.0. Zenodo. , 4682814 (2021).
- Killick, R., Fearnhead, P., Eckley, I. A. Optimal detection of changepoints with a linear computational cost. Journal of the American Statistical Association. 107 (500), 1590-1598 (2012).
- Schrangl, L., Göhring, J., Schütz, G. J. Kinetic analysis of single molecule FRET transitions without trajectories. The Journal of Chemical Physics. 148 (12), 123328 (2018).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Schrangl, L. Single-molecule FRET analysis software (3.0). Zenodo. , (2021).
- Nye, J. A., Groves, J. T. Kinetic control of histidine-tagged protein surface density on supported lipid bilayers. Langmuir. 24 (8), 4145-4149 (2008).
- Platzer, R., et al. Unscrambling fluorophore blinking for comprehensive cluster detection via photoactivated localization microscopy. Nature Communications. 11 (1), 4993 (2020).
- Johnson, I., Spence, M. . The molecular probes handbook: A guide to fluorescent probes and labeling technologies. , (2010).
- Szalai, A. M., et al. Super-resolution imaging of energy transfer by intensity-based STED-FRET. Nano Letters. 21 (5), 2296-2303 (2021).
- Miyawaki, A., et al. Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature. 388 (6645), 882-887 (1997).
- Zhai, B., Zhai, S., Hao, R., Xu, J., Liu, Z. A FRET-based two-photon probe for in vivo tracking of pH during a traumatic brain injury process. New Journal of Chemistry. 43 (43), 17018-17022 (2019).

