Lyskontrollerte gjæringer for mikrobiell kjemisk og proteinproduksjon
Summary
Optogenetisk kontroll av mikrobiell metabolisme gir fleksibel dynamisk kontroll over gjæringsprosesser. Protokollen her viser hvordan du setter opp blå lysregulerte gjæringer for kjemisk og proteinproduksjon på forskjellige volumetriske skalaer.
Abstract
Mikrobielle cellefabrikker tilbyr et bærekraftig alternativ for produksjon av kjemikalier og rekombinante proteiner fra fornybare råstoffer. Å overbelaste en mikroorganisme med genetiske modifikasjoner kan imidlertid redusere vertens kondisjon og produktivitet. Dette problemet kan løses ved å bruke dynamisk kontroll: induserbart uttrykk for enzymer og veier, vanligvis ved hjelp av kjemiske eller næringsbaserte tilsetningsstoffer, for å balansere cellulær vekst og produksjon. Optogenetikk tilbyr en ikke-invasiv, svært justerbar og reversibel metode for dynamisk regulering av genuttrykk. Her beskriver vi hvordan du setter opp lyskontrollerte gjæringer av konstruerte Escherichia coli og Saccharomyces cerevisiae for produksjon av kjemikalier eller rekombinante proteiner. Vi diskuterer hvordan man bruker lys på utvalgte tidspunkter og doser for å koble fra mikrobiell vekst og produksjon for forbedret gjæringskontroll og produktivitet, samt de viktigste optimaliseringshensynene for best resultat. I tillegg beskriver vi hvordan man implementerer lyskontroller for laboratorieskala bioreaktorforsøk. Disse protokollene letter innføringen av optogenetiske kontroller i konstruerte mikroorganismer for forbedret gjæringsytelse.
Introduction
Optogenetikk, kontrollen av biologiske prosesser med lysresponsive proteiner, tilbyr en ny strategi for dynamisk styring av mikrobielle gjæringer for kjemisk og proteinproduksjon1,2. Byrden av konstruerte metabolske veier og toksisiteten til noen mellomprodukter og produkter svekker ofte celleveksten3. Slike påkjenninger kan føre til dårlig biomasseakkumulering og redusert produktivitet3. Denne utfordringen kan løses ved å dele gjæringer i en vekst- og produksjonsfase, som vier metabolske ressurser til henholdsvis biomasseakkumulering eller produktsyntese4. Vi viste nylig at overgangen fra vekst til produksjon i denne tofase gjæringen kan induseres med endringer i belysningsforholdene5,6,7. Den høye tunabiliteten, reversibiliteten og ortogonaliteten til lysinnganger8 gir unike fordeler for lyskontrollerte gjæringer som er vanskelige eller umulige å gjenskape med kjemiske indusere som brukes i dynamisk kontroll av konvensjonelle tofasede gjæringer4,9,10,11.
Det blålys responsive EL222-proteinet avledet fra Erythrobacter litoralis har blitt brukt til å utvikle flere optogenetiske kretser for metabolsk engineering i Saccharomyces cerevisiae5,7,12,13. EL222 inneholder et LOV-domene (light-oxygen-voltage sensor) som gjennomgår et konformasjonsskift ved aktivering av blått lys (465 nm), som gjør det mulig å binde seg til sin kojakk-DNA-sekvens (C120)13. Fusing EL222 til det virale VP16-aktiveringsdomenet (VP16-EL222) resulterer i en responsiv transkripsjonsfaktor med blått lys som kan aktivere genuttrykk i S. cerevisiae7 og andre organismer14 fra den syntetiske promotoren PC120. Flere kretser basert på EL222 er utviklet og brukt til kjemisk produksjon i S. cerevisiae, for eksempel det grunnleggende lysaktiverte OptoEXP-systemet7, der genet av interesse uttrykkes direkte fra PC120 (figur 1A). Imidlertid motiverte bekymringer for lysgjennomtrengning ved de høye celletetthetene som vanligvis oppstår i produksjonsfasen av gjæring oss til å utvikle inverterte kretser som er indusert i mørket, for eksempel OptoINVRT- og OptoQ-INVRT-kretsene (figur 1B) 5,7,13. Disse systemene utnytter galaktose (GAL) eller quinic acid (Q) reguloner fra henholdsvis S. cerevisiae og N. crassa, kontrollerer deres tilsvarende undertrykkere (GAL80 og QS) med VP16-EL222, for å undertrykke genuttrykk i lyset og sterkt indusere det i mørket. Ved å kombinere OptoEXP- og OptoINVRT-kretser resulterer det i toveis kontroll av genuttrykk, noe som muliggjør tofasede gjæringer der vekstfasen induseres med blått lys, og produksjonsfasen med mørke (figur 2A)5,7.
Å bruke lys i stedet for mørke for å indusere genuttrykk under produksjonsfasen vil i stor grad utvide evnene til optogenetiske kontroller, men vil også kreve å overvinne lysgjennomtrengningsbegrensningene til de høye celletetthetene som vanligvis oppstår i denne gjæringsfasen. For dette formål har vi utviklet kretser, kjent som OptoAMP og OptoQ-AMP, som forsterker transkripsjonsresponsen på blå lysstimulering. Disse kretsene bruker vilt- eller overfølsomme mutanter av VP16-EL222 for å kontrollere produksjonen av transkripsjonsaktivatorene Gal4p eller QF2 i henholdsvis GAL- eller Q-regulonene, og oppnår forbedret følsomhet og sterkere genuttrykk med lys12,13 (figur 1C). OptoAMP-kretser kan oppnå fullstendig og homogen lysinduksjon i 5 L bioreaktorer med optisk tetthet (målt ved 600 nm; OD600) verdier på minst 40 med bare ~ 0,35% belysning (5% lysdose på bare ~ 7% av bulkoverflaten). Dette viser en høyere grad av følsomhet sammenlignet med OptoEXP, som krever nær 100% belysning12. Evnen til effektivt å indusere genuttrykk med lys ved høye celletettheter åpner nye muligheter for dynamisk kontroll av gjæringer. Dette inkluderer driftsgjæringer i mer enn to tidsfaser, for eksempel trefasede gjæringer, der vekst, induksjon og produksjonsfaser etableres med unike lysplaner for å optimalisere kjemisk produksjon (figur 2B)12.
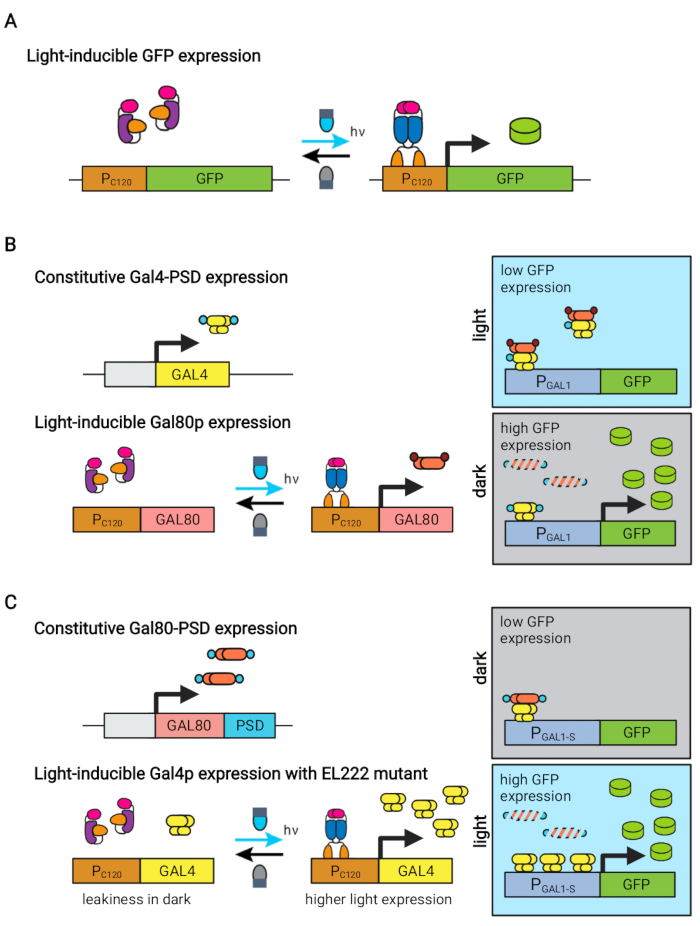
Figur 1: Optogenetiske kretser for dynamisk kontroll av S. cerevisiae. OptoEXP-, OptoINVRT- og OptoAMP-kretsene er basert på det lysfølsomme VP16-EL222-systemet. (A) I OptoEXP-kretsen forårsaker eksponering for blått lys en konformasjonsendring og dimerisering av VP16-EL222, som eksponerer et DNA-bindende domene og tillater transkripsjon fra PC120. Figuren er endret fra Zhao et al.7. (B) OptoINVRT-kretser utnytter GAL(vist) eller Q-reguloner for å indusere uttrykk i mørket. I GAL-baserte kretser uttrykkes VP16-EL222 og GAL4 konstituativt, mens PC120-stasjonsuttrykket til GAL80-repressoren (i Q-baserte kretser erstattes GAL4 og GAL80 av henholdsvis QF2 og QS, og en syntetisk QUAS-inneholdende promotor brukes i stedet for en GAL-promotor). I lys forhindrer Gal80p aktivering av genet av interesse fra PGAL1. I mørket uttrykkes ikke GAL80 og forringes raskt ved å fusjonere det til et konstituerende degrondomene (lite brunt domene), noe som muliggjør aktivering av PGAL1 av Gal4p. Figuren er endret fra Zhao et al.5. (C) OptoAMP-kretser bruker også VP16-EL222 til å kontrollere GAL- (vist) eller Q-reguloner. I disse kretsene er GAL80-repressoren (eller QS) konstituert uttrykt og smeltet sammen til en fotosensitiv degron (lite blått domene) som sikrer tett undertrykkelse i mørket. PC120 og en overfølsom VP16-EL222 mutant kontrolluttrykk av GAL4 (eller QF2) med lys, som sterkt aktiverer PGAL1 (eller en QUAS-inneholdende promotor) i lyset. GAL-avledede kretser kan bruke konstruerte former for PGAL1, for eksempel PGAL1-M eller PGAL1-S, som har økt aktivitet, samt wild-type promotorer kontrollert av GAL-regulonet (PGAL1, PGAL10, PGAL2, PGAL7). Figuren er endret fra Zhao et al.12. Klikk her for å se en større versjon av denne figuren.
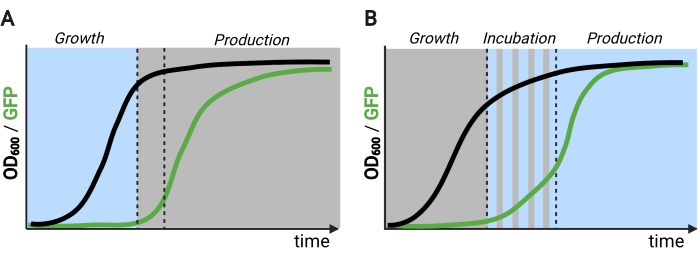
Figur 2: To- og trefasede gjæringer over tid. (A) Tofase gjæringer operert med inverterte kretser består av en lysdrevet vekstfase og en mørk produksjonsfase. I vekstfasen akkumuleres biomasse etter hvert som produksjonsveien forblir undertrykt. Når du når ønsket OD600, blir cellene flyttet til mørket for å justere metabolsk før de blir resuspendert i friske medier for produksjonsfasen. (B) I en trefaset prosess defineres vekst-, inkubasjons- og produksjonsfasene av unike lysplaner, som kan bestå av en mørk vekstperiode, pulsert inkubasjon og fullt opplyst produksjonsfase. Figur opprettet med Biorender. Klikk her for å se en større versjon av denne figuren.
Optogenetiske kretser er også utviklet for dynamisk kontroll av kjemisk og proteinproduksjon i E. coli. OptoLAC-kretser styrer den bakterielle LacI-repressoren ved hjelp av den lysresponsive pDawn-kretsen, som er basert på YF1/FixJ tokomponentsystem6 (figur 3). I likhet med OptoINVRT5 er OptoLAC-kretser designet for å undertrykke genuttrykk i lyset og indusere det i mørket. Uttrykksnivåer ved hjelp av OptoLAC-kretser kan matche eller overskride de som oppnås med standard isopropyl β-d-1-thiogalactopyranoside (IPTG) induksjon, og dermed opprettholde styrken av kjemisk induksjon samtidig som den tilbyr forbedret tunabilitet og reversibilitet6. Derfor muliggjør OptoLAC-kretser effektiv optogenetisk kontroll for metabolsk engineering i E. coli.
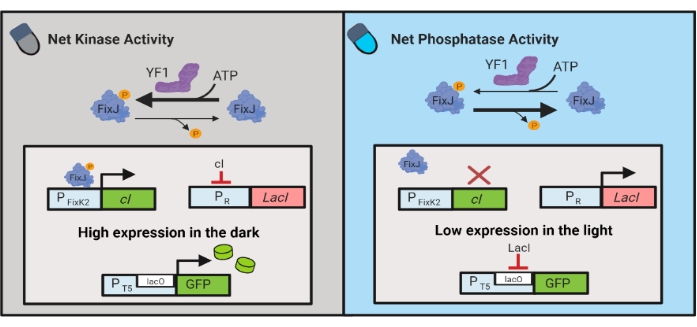
Figur 3: OptoLAC-kretser for dynamisk kontroll av E. coli. OptoLAC-kretsene tilpasser pDawn-systemet og lac operon for å oppnå aktivering i mørket og undertrykkelse i lyset. I mørket, YF1 fosforylater FixJ, som deretter aktiverer PFixK2 promotoren for å uttrykke cI-undertrykkeren . CI-undertrykkeren forhindrer uttrykk for laktI-undertrykkeren fra PR-promotoren , som tillater transkripsjon av genet av interesse fra en laktoholdig promotor. På den annen side reduserer blått lys YF1 netto kinase aktivitet, reversere FixJ fosforylering og dermed cI uttrykk, som derepresser uttrykk for lacI og forhindrer uttrykk fra lacO-inneholdende promotor. Figuren er endret fra Lalwani et al.6. Klikk her for å se en større versjon av denne figuren.
Vi beskriver her de grunnleggende protokollene for lyskontrollerte gjæringer av S. cerevisiae og E. coli for kjemisk eller proteinproduksjon. For både gjær og bakterier fokuserer vi først på gjæringer med en lysdrevet vekstfase og en mørkeindusert produksjonsfase aktivert av OptoINVRT- og OptoLAC-kretser. Deretter beskriver vi en protokoll for en trefaset (vekst, induksjon, produksjon) lyskontrollert gjæring aktivert av OptoAMP-kretser. Videre beskriver vi hvordan man skalerer opp optogenetisk kontrollerte gjæringer fra mikroplater til laboratorieskala bioreaktorer. Med denne protokollen tar vi sikte på å gi en komplett og lett reproduserbar guide for å utføre lyskontrollerte gjæringer for kjemisk eller proteinproduksjon.
Protocol
Representative Results
Discussion
Dynamisk kontroll har lenge vært brukt for å forbedre utbyttet for metabolsk engineering og rekombinant proteinproduksjon4. Endringer i enzymatisk uttrykk implementeres vanligvis ved hjelp av kjemiske indusere som IPTG21, galaktose22 og tetracyklin23, men har også blitt formidlet ved hjelp av prosessforhold som temperatur og pH. Optogenetisk kontroll av genuttrykk eliminerer behovet for endringer i gjæringsparametere eller…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne forskningen ble støttet av U.S. Department of Energy, Office of Science, Office of Biological and Environmental Research Award Number DE-SC0019363, NSF CAREER Award CBET-1751840, The Pew Charitable Trusts og Camille Dreyfus Teacher-Scholar Award.
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
References
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. 생체공학. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

