Autofluorescensbilleddannelse til evaluering af cellulær metabolisme
Summary
Denne protokol beskriver fluorescensbilleddannelse og analyse af de endogene metaboliske coenzymer, reduceret nicotinamid adenin (phosphat) dinukleotid (NAD (P) H) og oxideret flavin adenin dinukleotid (FAD). Autofluorescensbilleddannelse af NAD (P) H og FAD giver en etiketfri, ikke-destruktiv metode til vurdering af cellulær metabolisme.
Abstract
Cellulær metabolisme er den proces, hvormed celler genererer energi, og mange sygdomme, herunder kræft, er karakteriseret ved unormal metabolisme. Reduceret nicotinamid adenin (phosphat) dinukleotid (NAD (P) H) og oxideret flavin adenin dinukleotid (FAD) er coenzymer af metaboliske reaktioner. NAD (P) H og FAD udviser autofluorescens og kan spektralt isoleres ved excitation og emissionsbølgelængder. Begge coenzymer, NAD (P) H og FAD, kan eksistere i enten en fri eller proteinbundet konfiguration, som hver især har en særskilt fluorescenslevetid – den tid, hvor fluoroforen forbliver i ophidset tilstand. Fluorescenslevetidsbilleddannelse (FLIM) muliggør kvantificering af fluorescensintensiteten og levetiden for NAD (P) H og FAD til etiketfri analyse af cellulær metabolisme. Fluorescensintensitet og levetidsmikroskoper kan optimeres til billeddannelse af NAD (P) H og FAD ved at vælge de passende excitations- og emissionsbølgelængder. Metaboliske forstyrrelser af cyanid verificerer autofluorescensbilleddannelsesprotokoller for at detektere metaboliske ændringer i celler. Denne artikel vil demonstrere teknikken til autofluorescensbilleddannelse af NAD (P) H og FAD til måling af cellulær metabolisme.
Introduction
Metabolisme er den cellulære proces til produktion af energi. Cellulær metabolisme omfatter flere veje, herunder glykolyse, oxidativ fosforylering og glutaminolyse. Sunde celler bruger disse metaboliske veje til at generere energi til spredning og funktion, såsom produktion af cytokiner af immunceller. Mange sygdomme, herunder stofskifteforstyrrelser, kræft og neurodegeneration, er karakteriseret ved ændret cellulær metabolisme1. For eksempel har nogle kræftcelletyper forhøjede glykolysehastigheder, selv i nærvær af ilt, for at generere molekyler til syntese af nukleinsyrer, proteiner og lipider2,3. Dette fænomen, kendt som Warburg-effekten, er et kendetegn for mange kræfttyper, herunder brystkræft, lungekræft og glioblastomer4. På grund af ændringerne i cellulær metabolisme forbundet med kræftprogression kan cellulær metabolisme være en surrogatbiomarkør for lægemiddelrespons5,6. Desuden er forståelse af lægemiddeleffektivitet på celleniveau afgørende, da celleheterogenitet kan føre til forskellige lægemiddelresponser hos individer7,8.
Teknologier, der identificerer og kvantificerer ændringer i cellulær metabolisme, er afgørende for undersøgelser af kræft og lægemiddelrespons. Kemiske og proteinanalyser bruges til at evaluere metabolismen af celler eller væv, men mangler enkeltcelleopløsning og rumlig information. Metaboliske pladelæserbaserede assays kan måle pH og iltforbrug i prøven over tid og den efterfølgende metaboliske forstyrrelse af kemikalier. pH kan bruges til at beregne den ekstracellulære forsuringshastighed (ECAR), som giver et indblik i cellernes glykolytiske aktivitet9. Helkropsbilleddannelsesmetoder, herunder 2-[fluor-18] fluor-D-glucose positronemissionstomografi (FDG PET) og magnetisk resonansspektroskopi (MRS), er ikke-invasive billeddannelsesmetoder, der anvendes klinisk til at identificere tumorgentagelse og lægemiddeleffektivitet gennem metaboliske målinger10,11,12,13,14.
FDG-PET billeder vævsoptagelsen af FDG, en radioaktivt mærket glukoseanalog. Øget optagelse af FDG-PET af tumorer i forhold til omgivende væv skyldes Warburg-effekten12,13. MRS-billeder af almindelige kerner af molekyler, der anvendes til metabolisme, såsom 13C og 31P, og kan få dynamisk information om, hvordan stofskiftet ændres som reaktion på stimuli, såsom motion eller spisning14. Selvom FDG-PET og MRS kan bruges klinisk, mangler disse teknologier den rumlige opløsning til at løse intratumoral heterogenitet. Ligeledes foretages iltforbrugsmålinger på en bulkpopulation af celler. Autofluorescensbilleddannelse overvinder den rumlige opløsningshindring for disse teknologier og giver en ikke-invasiv metode til kvantificering af cellulær metabolisme.
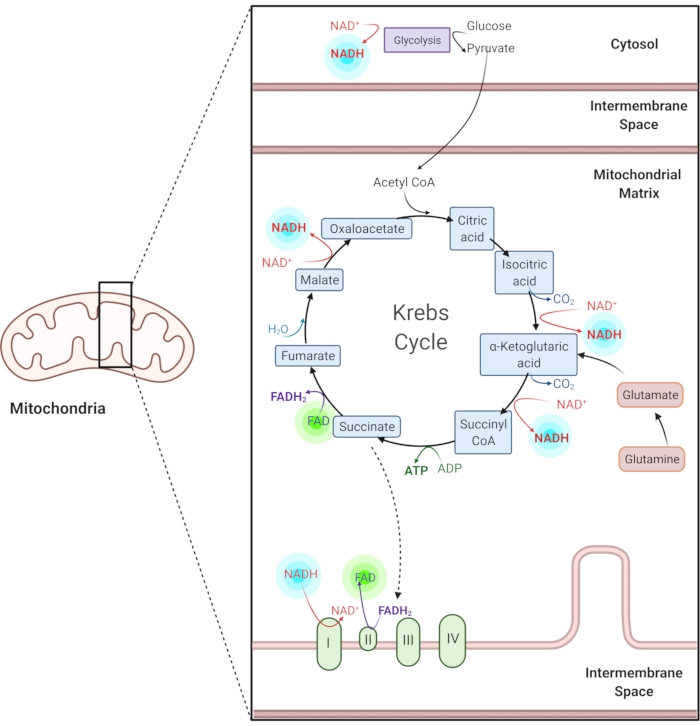
Figur 1: NADH og FAD i fælles metaboliske veje. NADH og FAD er coenzymer, der anvendes i glykolyse, Krebs-cyklussen og elektrontransportkæden. Autofluorescensbilleddannelse af disse molekyler giver information om cellulær metabolisme. Klik her for at se en større version af denne figur.
Reduceret nicotinamid adenin (phosphat) dinukleotid (NAD (P) H) og oxideret flavin adenin dinukleotid (FAD) er coenzymer af metaboliske reaktioner, herunder glycolyse, oxidativ phosphorylering og glutaminolyse (figur 1). Både NAD(P)H og FAD er autofluorescerende og giver endogen kontrast til fluorescensbilleddannelse1,15. NADPH har lignende fluorescerende egenskaber som NADH. På grund af dette bruges NAD (P) H ofte til at repræsentere det kombinerede signal fra NADH og NADPH2,16.
Fluorescens levetidsbilleddannelse (FLIM) kvantificerer fluorescensens levetid eller den tid, hvor en fluorofor er i ophidset tilstand. Fluorescensens levetid reagerer på fluoroforernes mikromiljø og giver information om cellulær metabolisme17. NAD(P)H og FAD kan eksistere i celler i enten proteinbundne eller frie konformationer, som hver især har en anden levetid. Fri NAD(P)H har en kortere levetid end proteinbundet NAD(P)H; Omvendt har fri FAD en længere levetid end bundet FAD18,19. Levetiden og levetidskomponentvægtene kan kvantificeres ud fra fluorescensens levetidshenfaldsdata gennem Eq. (1)20:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) repræsenterer den normaliserede fluorescensintensitet som funktion af tiden. De α 1 og α 2 i denne ligning repræsenterer de proportionale komponenter i korte og lange levetider (α 1+ α 2=1), henholdsvis τ1 og τ2 repræsenterer henholdsvis den korte og lange levetid, og C tegner sig for baggrundslys7,20. Den amplitudevægtede levetid, her repræsenteret som τm, beregnes ved hjælp af Eq. (2).
τm= α 1τ1+ α 2τ2 (2)
En gennemsnitlig levetid kan beregnes ved at beregne gennemsnittet af “t” over fluoroforens intensitetshenfald, som for et to-eksponentielt henfald er vist ved Eq. (3)17,21.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Et fluorescensintensitetsbillede kan beregnes ud fra livstidsbilledet ved at integrere fluorescensens levetidshenfald. Autofluorescensbilleddannelse er en ikke-destruktiv og etiketfri metode, der kan bruges til at karakterisere metabolismen af levende celler ved en subcellulær opløsning. Det optiske redoxforhold tilvejebringer en optisk analog metrik af cellens kemiske redoxtilstand og beregnes som forholdet mellem NAD (P) H og FAD intensiteter. Selvom formlen til beregning af det optiske redoxforhold ikke er standardiseret22,23,24,25, defineres den her som intensiteten af FAD over de kombinerede intensiteter af NAD (P) H og FAD. Denne definition anvendes, fordi den opsummerede intensitet i nævneren normaliserer metrikken mellem 0 og 1, og det forventede resultat af cyanidhæmningen er et fald i redoxforholdet. Fluorescensleveterne for fri NAD(P)H og FAD giver indsigt i ændringer i det metaboliske opløsningsmiddelmikromiljø, herunder pH, temperatur, nærhed til ilt og osmolaritet17.
Ændringer i fluorescenslevetid for de bundne fraktioner af NAD(P)H og FAD kan indikere udnyttelse af metabolisk vej og substratspecifik metabolisme26. Komponentvægte kan fortolkes for ændringer i den frie til den bundne brøkdel af coenzymerne18,19. Alt i alt tillader disse kvantitative autofluorescenslevetidsmålinger analyse af cellulær metabolisme, og autofluorescensbilleddannelse er blevet anvendt til at identificere neoplasmer fra normalt væv27,28, karakterisere stamceller29,30, evaluere immuncellefunktion31,32,33,34,35, måle neurologisk aktivitet36, 37,38 og forståelse af lægemiddeleffektivitet i kræfttyper som brystkræft og hoved- og halskræft21,39,40,41,42. Autofluorescensbilleddannelse i høj opløsning kan kombineres med billedsegmentering til enkeltcelleanalyse og kvantificering af intrapopulationsheterogenitet43,44,45,46,47.
NAD(P)H og FAD kan afbildes på enkeltfoton- eller multifotonfluorescensmikroskoper, der er konfigureret til intensitets- eller levetidsbilleddannelse. For enkeltfotonmikroskoper er NAD(P)H og FAD typisk spændte ved bølgelængder på henholdsvis 375-405 nm og 488 nm på grund af almindelige laserkilder ved disse bølgelængder48. I to-fotonfluorescens excitation vil NAD (P) H og FAD ophidse ved bølgelængder på henholdsvis ca. 700 til 750 nm og 700 til 900 nm15,49. Når fluoroforerne er spændte, udsender NAD (P) H og FAD fotoner ved bølgelængder mellem ~ 410 nm til ~ 490 nm og ~ 510 nm til ~ 640 nm, henholdsvis15. NAD(P)H- og FAD maxima-emissionsbølgelængderne er henholdsvis ca. 450 nm og 535 nm48.
På grund af deres forskellige excitations- og emissionsbølgelængder kan fluorescensen af de to metaboliske coenzymer isoleres spektralt. En forståelse af de spektrale egenskaber ved NAD (P) H og FAD er nødvendig for design og optimering af autofluorescensbilleddannelsesprotokoller. Cyanid er en elektrontransportkæde (ETC) kompleks IV-hæmmer. Virkningerne af cyanid på cellulær metabolisme og autofluorescensintensiteterne og levetiderne for NAD (P) H og FAD i celler er godt karakteriseret27,40. Derfor er et cyanidforstyrrelseseksperiment et effektivt middel til validering af NAD (P) H- og FAD-billeddannelsesprotokoller. Et vellykket cyanideeksperiment giver tillid til, at NAD (P) H og FAD-billeddannelsesprotokollen kan bruges til at vurdere metabolismen af ukendte grupper eller forstyrrelser.
Protocol
Representative Results
Discussion
Autofluorescensintensitet og levetidsbilleddannelse er blevet brugt i vid udstrækning til at vurdere metabolisme i celler21,55. FLIM er høj opløsning og løser derfor enkeltceller, hvilket er vigtigt for kræftundersøgelser, fordi cellulær heterogenitet bidrager til tumoraggression og lægemiddelresistens7,39,41,44,45,46,58.</…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Finansieringskilder omfatter Cancer Prevention and Research Institute of Texas (CPRIT RP200668) og Texas A & M University. Figur 1 blev oprettet med BioRender.com.
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
References
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. 암 연구학. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. 암 연구학. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. 암 연구학. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

