Autofluorescentie beeldvorming om cellulair metabolisme te evalueren
Summary
Dit protocol beschrijft fluorescentiebeeldvorming en analyse van de endogene metabole co-enzymen, gereduceerd nicotinamide-adenine (fosfaat) dinucleotide (NAD (P) H) en geoxideerd flavine-adenine dinucleotide (FAD). Autofluorescentie beeldvorming van NAD(P)H en FAD biedt een labelvrije, niet-destructieve methode om het cellulaire metabolisme te beoordelen.
Abstract
Cellulair metabolisme is het proces waarbij cellen energie genereren en veel ziekten, waaronder kanker, worden gekenmerkt door een abnormaal metabolisme. Gereduceerd nicotinamide-adenine (fosfaat) dinucleotide (NAD(P)H) en geoxideerd flavine adenine dinucleotide (FAD) zijn co-enzymen van metabole reacties. NAD(P)H en FAD vertonen autofluorescentie en kunnen spectraal worden geïsoleerd door excitatie- en emissiegolflengten. Beide co-enzymen, NAD(P)H en FAD, kunnen bestaan in een vrije of eiwitgebonden configuratie, die elk een verschillende fluorescentielevensduur hebben – de tijd gedurende welke de fluorofoor in de aangeslagen toestand blijft. Fluorescentie lifetime imaging (FLIM) maakt kwantificering van de fluorescentie-intensiteit en levensduur van NAD(P)H en FAD mogelijk voor labelvrije analyse van cellulair metabolisme. Fluorescentie-intensiteit en levensduurmicroscopen kunnen worden geoptimaliseerd voor beeldvorming van NAD (P) H en FAD door de juiste excitatie- en emissiegolflengten te selecteren. Metabole verstoringen door cyanide verifiëren autofluorescentie beeldvormingsprotocollen om metabole veranderingen in cellen te detecteren. Dit artikel demonstreert de techniek van autofluorescentie beeldvorming van NAD(P)H en FAD voor het meten van cellulair metabolisme.
Introduction
Metabolisme is het cellulaire proces van het produceren van energie. Cellulair metabolisme omvat meerdere routes, waaronder glycolyse, oxidatieve fosforylering en glutaminolyse. Gezonde cellen gebruiken deze metabole routes om energie te genereren voor proliferatie en functie, zoals de productie van cytokines door immuuncellen. Veel ziekten, waaronder metabole stoornissen, kanker en neurodegeneratie, worden gekenmerkt door een veranderd cellulair metabolisme1. Sommige soorten kankercellen hebben bijvoorbeeld verhoogde glycolysesnelheden, zelfs in de aanwezigheid van zuurstof, om moleculen te genereren voor de synthese van nucleïnezuren, eiwitten en lipiden2,3. Dit fenomeen, bekend als het Warburg-effect, is een kenmerk van vele soorten kanker, waaronder borstkanker, longkanker en glioblastomen4. Vanwege de veranderingen van het cellulaire metabolisme geassocieerd met kankerprogressie, kan cellulair metabolisme een surrogaatbiomarker zijn voor medicijnrespons5,6. Bovendien is het begrijpen van de werkzaamheid van geneesmiddelen op cellulair niveau cruciaal, omdat celheterogeniteit kan leiden tot verschillende geneesmiddelresponsen bij individuen7,8.
Technologieën die veranderingen in het cellulaire metabolisme identificeren en kwantificeren, zijn essentieel voor studies naar kanker en medicijnrespons. Chemische en eiwitanalyses worden gebruikt om het metabolisme van cellen of weefsels te evalueren, maar missen eencellige resolutie en ruimtelijke informatie. Metabole plaatlezer-gebaseerde assays kunnen de pH en het zuurstofverbruik in het monster in de loop van de tijd en de daaropvolgende metabole verstoring door chemicaliën meten. De pH kan worden gebruikt om de extracellulaire verzuringssnelheid (ECAR) te berekenen, wat inzicht geeft in de glycolytische activiteit van de cellen9. Beeldvormingsmethoden voor het hele lichaam, waaronder 2-[fluor-18] fluor-D-glucose positronemissietomografie (FDG PET) en magnetische resonantiespectroscopie (MRS), zijn niet-invasieve beeldvormingsmodaliteiten die klinisch worden gebruikt om tumorrecidief en werkzaamheid van geneesmiddelen te identificeren door middel van metabole metingen10,11,12,13,14.
FDG-PET brengt de weefselopname van FDG in beeld, een radioactief gelabeld glucose-analoog. Verhoogde opname van FDG-PET door tumoren ten opzichte van het omliggende weefsel is te wijten aan het Warburg-effect12,13. MRS toont gemeenschappelijke kernen van moleculen die worden gebruikt voor het metabolisme, zoals 13C en 31P, en kan dynamische informatie verkrijgen over hoe het metabolisme verandert als reactie op stimuli, zoals lichaamsbeweging of eten14. Hoewel FDG-PET en MRS klinisch kunnen worden gebruikt, missen deze technologieën de ruimtelijke resolutie om intratumorale heterogeniteit op te lossen. Evenzo worden metingen van het zuurstofverbruik uitgevoerd op een bulkpopulatie van cellen. Autofluorescentie beeldvorming overwint het ruimtelijke resolutie obstakel van deze technologieën en biedt een niet-invasieve methode voor het kwantificeren van cellulair metabolisme.
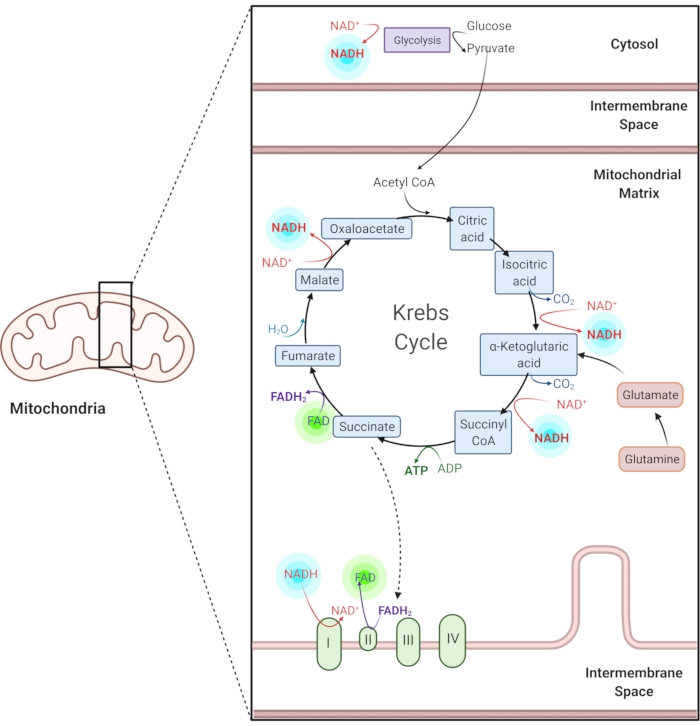
Figuur 1: NADH en FAD in gemeenschappelijke metabole routes. NADH en FAD zijn co-enzymen die worden gebruikt in glycolyse, de Krebs-cyclus en de elektronentransportketen. Autofluorescentie beeldvorming van deze moleculen geeft informatie over cellulair metabolisme. Klik hier om een grotere versie van deze figuur te bekijken.
Gereduceerd nicotinamide-adenine (fosfaat) dinucleotide (NAD (P)H) en geoxideerd flavine adenine dinucleotide (FAD) zijn co-enzymen van metabole reacties, waaronder glycolyse, oxidatieve fosforylering en glutaminolyse (figuur 1). Zowel NAD(P)H als FAD zijn autofluorescent en bieden endogene contrast voor fluorescentiebeeldvorming1,15. NADPH heeft vergelijkbare fluorescerende eigenschappen als NADH. Daarom wordt NAD(P)H vaak gebruikt om het gecombineerde signaal van NADH en NADPH2,16 weer te geven.
Fluorescence lifetime imaging (FLIM) kwantificeert de fluorescentielevensduur of de tijd waarvoor een fluorofoor zich in de aangeslagen toestand bevindt. Fluorescentielevensduur reageert op de micro-omgeving van de fluoroforen en geeft informatie over cellulair metabolisme17. NAD(P)H en FAD kunnen in cellen bestaan in eiwitgebonden of vrije conformaties, die elk een andere levensduur hebben. Vrije NAD(P)H heeft een kortere levensduur dan eiwitgebonden NAD(P)H; omgekeerd heeft vrije FAD een langere levensduur dan gebonden FAD18,19. De levensduur en het gewicht van de levensduurcomponenten kunnen worden gekwantificeerd op basis van gegevens over het levensduurverval van fluorescentie via Eq. (1)20:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) vertegenwoordigt de genormaliseerde fluorescentie-intensiteit als functie van de tijd. De α 1 en α 2 in deze vergelijking vertegenwoordigen de proportionele componenten van korte en lange levensduur (respectievelijk α 1+ α 2=1), respectievelijk τ1 en τ2 vertegenwoordigen respectievelijk de korte en lange levensduur, en C is goed voor achtergrondlicht7,20. De amplitudegewogen levensduur, hier weergegeven als τm, wordt berekend met eq. (2).
τm= α 1τ1+ α 2τ2 (2)
Een gemiddelde levensduur kan worden berekend door het gemiddelde van “t” over het intensiteitsverval van de fluorofoor, wat voor een twee-exponentieel verval wordt aangetoond door Eq. (3)17,21.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Een fluorescentie-intensiteitsbeeld kan worden berekend uit het levensduurbeeld door het fluorescentielevensverval te integreren. Autofluorescentie beeldvorming is een niet-destructieve en labelvrije methode die kan worden gebruikt om het metabolisme van levende cellen met een subcellulaire resolutie te karakteriseren. De optische redoxverhouding biedt een optische analoge metriek van de chemische redoxtoestand van de cel en wordt berekend als de verhouding van NAD(P)H- en FAD-intensiteiten. Hoewel de formule voor het berekenen van de optische redoxverhouding niet gestandaardiseerd is22,23,24,25, wordt deze hier gedefinieerd als de intensiteit van FAD over de gecombineerde intensiteiten van NAD(P)H en FAD. Deze definitie wordt gebruikt omdat de opgetelde intensiteit in de noemer de metriek tussen 0 en 1 normaliseert, en het verwachte resultaat van de cyanideremming een afname van de redoxverhouding is. De fluorescentielevensduur van vrije NAD(P)H en FAD geeft inzicht in veranderingen in de micro-omgeving van het metabole oplosmiddel, waaronder pH, temperatuur, nabijheid van zuurstof en osmolariteit17.
Veranderingen in de fluorescentielevensduur van de gebonden fracties van NAD(P)H en FAD kunnen wijzen op het gebruik van metabole routes en substraatspecifiek metabolisme26. Componentgewichten kunnen worden geïnterpreteerd voor veranderingen in de vrije tot de gebonden fractie van de co-enzymen18,19. Al met al maken deze kwantitatieve autofluorescentielevensduurmetingen de analyse van cellulair metabolisme mogelijk, en autofluorescentiebeeldvorming is gebruikt voor het identificeren van neoplasmata uit normale weefsels27,28, het karakteriseren van stamcellen29,30, het evalueren van de immuuncelfunctie31,32,33,34,35, het meten van neurologische activiteit36, 37,38, en inzicht in de werkzaamheid van geneesmiddelen bij kankersoorten zoals borstkanker en hoofd-halskanker21,39,40,41,42. Hoge-resolutie autofluorescentie beeldvorming kan worden gecombineerd met beeldsegmentatie voor eencellige analyse en kwantificering van intrapopulatie heterogeniteit43,44,45,46,47.
NAD(P)H en FAD kunnen worden afgebeeld op fluorescentiemicroscopen met één foton of multifotonen die zijn geconfigureerd voor intensiteits- of levenslange beeldvorming. Voor microscopen met één foton worden NAD(P)H en FAD meestal geëxciteerd op golflengten van respectievelijk 375-405 nm en 488 nm, als gevolg van gemeenschappelijke laserbronnen op deze golflengten48. Bij fluorescentie-excitatie van twee fotonen zullen NAD(P)H en FAD exciteren op golflengten van respectievelijk ongeveer 700 tot 750 nm en 700 tot 900 nm, 15,49. Zodra de fluoroforen zijn geëxciteerd, zenden NAD(P)H en FAD fotonen uit op golflengten tussen respectievelijk ~410 nm tot ~490 nm en ~510 nm tot ~640 nm15. De NAD(P)H en FAD maxima emissie golflengten zijn respectievelijk ongeveer 450 nm en 535 nm48.
Vanwege hun verschillende excitatie- en emissiegolflengten kan de fluorescentie van de twee metabole co-enzymen spectraal worden geïsoleerd. Een goed begrip van de spectrale kenmerken van NAD(P)H en FAD is noodzakelijk voor het ontwerp en de optimalisatie van autofluorescentie beeldvormingsprotocollen. Cyanide is een elektronentransportketen (ETC) complexe IV-remmer. De effecten van cyanide op het cellulair metabolisme en de autofluorescentie-intensiteiten en levensduur van NAD(P)H en FAD in cellen worden goed gekarakteriseerd27,40. Daarom is een cyanide perturbatie-experiment een effectief middel om NAD(P)H- en FAD-beeldvormingsprotocollen te valideren. Een succesvol cyanide-experiment geeft vertrouwen dat het NAD(P)H- en FAD-beeldvormingsprotocol kan worden gebruikt om het metabolisme van onbekende groepen of verstoringen te beoordelen.
Protocol
Representative Results
Discussion
Autofluorescentie-intensiteit en levenslange beeldvorming zijn op grote schaal gebruikt om het metabolisme in cellen te beoordelen21,55. FLIM heeft een hoge resolutie en lost daarom afzonderlijke cellen op, wat belangrijk is voor kankerstudies omdat cellulaire heterogeniteit bijdraagt aan tumoragressie en medicijnresistentie7,39,41,44,45,46,58.<sup class="xref…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Financieringsbronnen zijn onder meer het Cancer Prevention and Research Institute of Texas (CPRIT RP200668) en Texas A & M University. Figuur 1 is gemaakt met BioRender.com.
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
References
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. 암 연구학. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. 암 연구학. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. 암 연구학. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

