細胞代謝を評価するための自己蛍光イメージング
Summary
このプロトコルは、内因性代謝補酵素、還元型ニコチンアミドアデニン(リン酸)ジヌクレオチド(NAD(P)H)、および酸化フラビンアデニンジヌクレオチド(FAD)の蛍光イメージングおよび分析について記載しています。NAD(P)HおよびFADの自己蛍光イメージングは、細胞代謝を評価するための標識フリーの非破壊的方法を提供します。
Abstract
細胞代謝は細胞がエネルギーを生成するプロセスであり、癌を含む多くの疾患は異常な代謝によって特徴付けられる。還元型ニコチンアミドアデニン(リン酸)ジヌクレオチド(NAD(P)H)および酸化型フラビンアデニンジヌクレオチド(FAD)は、代謝反応の補酵素である。NAD(P)HおよびFADは自己蛍光を示し、励起および発光波長によってスペクトル的に単離することができる。両方の補酵素、NAD(P)HおよびFADは、遊離またはタンパク質結合配置のいずれかで存在することができ、それぞれが異なる蛍光寿命(蛍光色素分子が励起状態にとどまる時間)を有する。蛍光寿命イメージング(FLIM)は、細胞代謝の標識のない分析のために、NAD(P)HおよびFADの蛍光強度および寿命の定量化を可能にする。蛍光強度と寿命顕微鏡は、適切な励起波長と発光波長を選択することで、NAD(P)HとFADのイメージングに最適化できます。シアン化物による代謝摂動は、細胞内の代謝変化を検出するための自己蛍光イメージングプロトコルを検証します。本稿では、細胞代謝を測定するためのNAD(P)HおよびFADの自己蛍光イメージング技術を実証する。
Introduction
代謝は、エネルギーを生産する細胞プロセスです。細胞代謝は、解糖系、酸化的リン酸化、およびグルタミン分解を含む複数の経路を包含する。健康な細胞は、これらの代謝経路を使用して、免疫細胞によるサイトカインの産生などの増殖および機能のためのエネルギーを生成する。代謝障害、癌、神経変性を含む多くの疾患は、細胞代謝の変化によって特徴付けられます1。例えば、一部のがん細胞型は、酸素の存在下でも解糖系の割合が上昇し、核酸、タンパク質、および脂質を合成するための分子を生成します2,3。ウォーバーグ効果として知られるこの現象は、乳がん、肺がん、膠芽腫など、多くのがんタイプの特徴です4。がんの進行に伴う細胞代謝の変化のため、細胞代謝は薬物応答の代理バイオマーカーとなり得る5,6。さらに、細胞の不均一性は個人において異なる薬物応答につながる可能性があるため、細胞レベルで薬物の有効性を理解することは極めて重要です7,8。
細胞代謝の変化を同定および定量化する技術は、がんおよび薬物応答の研究に不可欠です。化学分析およびタンパク質分析は、細胞または組織の代謝を評価するために使用されますが、単一細胞分解能および空間情報が不足しています。代謝プレートリーダーベースのアッセイは、経時的にサンプル中のpHおよび酸素消費量、およびその後の化学物質による代謝摂動を測定することができる。pHを使用して細胞外酸性化率(ECAR)を計算することができ、細胞の解糖系活性に関する洞察が得られます9。2-[フッ素-18]フルオロ-D-グルコース陽電子放射断層撮影法(FDG PET)および磁気共鳴分光法(MRS)を含む全身イメージング法は、代謝測定を通じて腫瘍の再発および薬物有効性を同定するために臨床的に使用される非侵襲的イメージングモダリティである10,11,12,13,14。
FDG-PETは、放射性標識グルコース類似体であるFDGの組織取り込みを画像化する。周囲の組織に対する腫瘍によるFDG-PETの取り込みの増加は、ウォーバーグ効果によるものである12,13。MRSは、13Cや31Pなどの代謝に使用される分子の一般的な核を画像化し、運動や食事などの刺激に応答して代謝がどのように変化するかについての動的情報を得ることができます14。FDG-PETおよびMRSは臨床的に使用することができるが、これらの技術は腫瘍内の不均一性を解決するための空間分解能を欠いている。同様に、酸素消費量の測定は、大量の細胞集団に対して行われます。自己蛍光イメージングは、これらの技術の空間分解能の障害を克服し、細胞代謝を定量化する非侵襲的な方法を提供します。
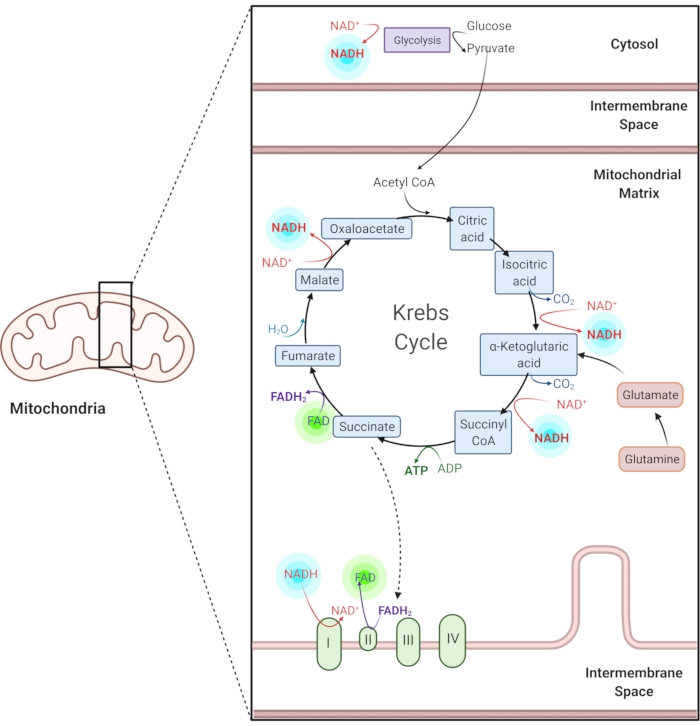
図1:一般的な代謝経路におけるNADHおよびFAD。 NADHおよびFADは、解糖系、クレブスサイクル、および電子輸送鎖に使用される補酵素である。これらの分子の自己蛍光イメージングは、細胞代謝に関する情報を提供する。 この図の拡大版を表示するには、ここをクリックしてください。
還元型ニコチンアミドアデニン(リン酸)ジヌクレオチド(NAD(P)H)および酸化型フラビンアデニンジヌクレオチド(FAD)は、解糖系、酸化的リン酸化系、グルタミン分解系などの代謝反応の補酵素です(図1)。NAD(P)HとFADはどちらも自己蛍光性であり、蛍光イメージングに内因性のコントラストを提供します1,15。NADPHはNADHと同様の蛍光特性を有する。このため、NAD(P)H は、NADH と NADPH2,16 の結合信号を表すためによく使用されます。
蛍光寿命イメージング(FLIM)は、蛍光寿命または蛍光色素分子が励起状態にある時間を定量化します。蛍光の寿命は蛍光色素分子の微小環境に反応し、細胞代謝に関する情報を提供します17。NAD(P)HおよびFADは、タンパク質結合または遊離立体配座のいずれかで細胞内に存在することができ、それぞれが異なる寿命を有する。遊離NAD(P)Hは、タンパク質結合NAD(P)Hよりも寿命が短い。逆に、遊離FADは束縛されたFADよりも寿命が長い18,19。寿命および寿命成分の重みは、蛍光寿命減衰データから式(1)20を通して定量化することができる。
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
式(1)は、時間の関数として規格化された蛍光強度を表す。この式のα 1とα 2は、それぞれ短寿命と長寿命(α 1+α 2=1)の比例成分を表し、τ1とτ2はそれぞれ短寿命と長寿命を表し、Cは背景光7,20を表します。振幅加重寿命は、ここではτmとして表され、式(2)を使用して計算されます。
τm= α 1τ1+ α 2τ2 (2)
平均寿命は、蛍光色素分子の強度減衰に対して「t」を平均することによって計算することができ、2指数関数的な減衰の場合、式(3)17,21で示されます。
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
蛍光強度画像は、蛍光寿命減衰を積分することにより、寿命画像から計算することができる。自己蛍光イメージングは、生細胞の代謝を細胞内分解能で特徴付けるために使用できる非破壊的で標識のない方法です。光酸化還元比は、セルの化学的酸化還元状態の光学アナログ測定を提供し、NAD(P)HとFAD強度の比として計算されます。光学酸化還元比の計算式は標準化されていませんが22,23,24,25,ここではNAD(P)HとFADを合わせた強度に対するFADの強度と定義しています。この定義が使用されるのは、分母の強度の合計がメトリックを 0 ~ 1 の間で正規化し、シアン化物阻害の予想される結果が酸化還元比の減少であるためです。遊離NAD(P)HおよびFADの蛍光寿命は、pH、温度、酸素への近接性、浸透圧など、代謝溶媒微小環境の変化に関する洞察を提供します17。
NAD(P)HとFADの結合画分の蛍光寿命の変化は、代謝経路の利用と基質特異的代謝を示す可能性があります26。成分重量は、補酵素の遊離から結合画分への変化について解釈することができる18、19。全体として、これらの定量的自己蛍光寿命測定基準は、細胞代謝の分析を可能にし、自己蛍光イメージングは、正常組織からの新生物の同定27,28、幹細胞の特性評価29,30、免疫細胞機能の評価31,32,33,34,35、神経学的活性の測定36、 37、38、および乳癌および頭頸部癌などの癌タイプにおける薬物有効性を理解する21、39、40、41、42。高解像度の自己蛍光イメージングは、単一細胞分析および集団内不均一性の定量化のための画像セグメンテーションと組み合わせることができます43,44,45,46,47。
NAD(P)HおよびFADは、強度または寿命イメージング用に構成された単一光子または多光子蛍光顕微鏡でイメージングできます。単一光子顕微鏡の場合、NAD(P)HとFADは、これらの波長で共通のレーザー光源のために、それぞれ375-405nmと488nmの波長で励起されるのが一般的です48。2光子蛍光励起では、NAD(P)HとFADはそれぞれ約700~750nmと700~900nmの波長で励起します15,49。蛍光色素分子が励起されると、NAD(P)HおよびFADは、それぞれ~410 nm~~490 nmおよび~510 nm~~640 nmの波長で光子を放出します15。NAD(P)HおよびFADの最大発光波長は、それぞれ約450nmおよび535nmである48。
励起波長と発光波長が異なるため、2つの代謝補酵素の蛍光をスペクトル的に単離することができます。NAD(P)HおよびFADのスペクトル特性を理解することは、自己蛍光イメージングプロトコルの設計および最適化に必要です。シアン化物は電子輸送鎖(ETC)錯体IV阻害剤である。細胞代謝に対するシアン化物の影響、ならびに細胞内のNAD(P)HおよびFADの自己蛍光強度および寿命は、よく特徴付けられる27,40。したがって、シアン化物摂動実験は、NAD(P)HおよびFADイメージングプロトコルを検証するための有効な手段である。シアン化物実験が成功すれば、NAD(P)HおよびFADイメージングプロトコルを使用して未知のグループまたは摂動の代謝を評価できるという確信が得られます。
Protocol
Representative Results
Discussion
自己蛍光強度および寿命イメージングは、細胞における代謝を評価するために広く使用されている21,55。FLIMは高分解能であり、したがって単一細胞を解決し、細胞の不均一性が腫瘍の攻撃性および薬剤耐性に寄与するため、癌研究にとって重要である7,39,41,44,45,46,58。<sup…
Disclosures
The authors have nothing to disclose.
Acknowledgements
資金源には、Cancer Prevention and Research Institute of Texas (CPRIT RP200668) とTexas A&M Universityが含まれる。 図 1 は、BioRender.com を使用して作成されました。
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
References
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. 암 연구학. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. 암 연구학. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. 암 연구학. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

