세포 대사를 평가하기 위한 자가형광 이미징
Summary
이 프로토콜은 내인성 대사 조효소, 환원된 니코틴아미드 아데닌(인산염) 디뉴클레오타이드(NAD(P)H) 및 산화된 플라빈 아데닌 디뉴클레오타이드(FAD)의 형광 이미징 및 분석을 기술한다. NAD(P)H 및 FAD의 자가형광 이미징은 세포 대사를 평가하기 위한 라벨이 없는 비파괴적인 방법을 제공한다.
Abstract
세포 대사는 세포가 에너지를 생성하는 과정이며, 암을 포함한 많은 질병은 비정상적인 신진 대사를 특징으로합니다. 환원된 니코틴아미드 아데닌(포스페이트) 디뉴클레오타이드(NAD(P)H) 및 산화된 플라빈 아데닌 디뉴클레오타이드(FAD)는 대사 반응의 조효소이다. NAD(P)H 및 FAD는 자가형광을 나타내며 여기 및 방출 파장에 의해 분광적으로 분리될 수 있다. 조효소인 NAD(P)H 및 FAD는 모두 유리 또는 단백질 결합 구성으로 존재할 수 있으며, 이들 각각은 형광단이 여기된 상태로 유지되는 시간인 뚜렷한 형광 수명을 갖는다. 형광 수명 이미징 (FLIM)은 세포 대사의 라벨없는 분석을 위해 NAD (P) H 및 FAD의 형광 강도와 수명을 정량화 할 수 있습니다. 형광 강도 및 수명 현미경은 적절한 여기 및 방출 파장을 선택하여 NAD(P)H 및 FAD 이미징에 최적화할 수 있습니다. 시안화물에 의한 대사 교란은 세포 내의 대사 변화를 검출하기 위해 자기 형광 이미징 프로토콜을 검증합니다. 이 기사에서는 세포 신진 대사를 측정하기위한 NAD (P) H 및 FAD의 자기 형광 이미징 기술을 시연합니다.
Introduction
신진 대사는 에너지를 생산하는 세포 과정입니다. 세포 대사는 글리코 분해, 산화 인산화 및 글루타미노분해를 포함한 여러 경로를 포함합니다. 건강한 세포는 면역 세포에 의한 사이토카인의 생산과 같은 증식 및 기능을위한 에너지를 생성하기 위해 이러한 대사 경로를 사용합니다. 대사 장애, 암 및 신경 변성을 포함한 많은 질병은 변경된 세포 대사를 특징으로합니다1. 예를 들어, 일부 암 세포 유형은 산소의 존재 하에서도 핵산, 단백질 및 지질의 합성을 위한 분자를 생성하기 위해 상승된 당분해 속도를 갖는다2,3. 바르부르크 효과로 알려진 이 현상은 유방암, 폐암, 교모세포종4을 포함한 많은 암 유형의 특징이다. 암 진행과 관련된 세포 대사의 변화로 인해, 세포 대사는 약물 반응에 대한 대리 바이오마커가 될 수 있다5,6. 더욱이, 세포 수준에서 약물 효능을 이해하는 것은 세포 이질성이 개인에서 상이한 약물 반응을 초래할 수 있기 때문에 중요하다7,8.
세포 신진 대사의 변화를 확인하고 정량화하는 기술은 암 및 약물 반응 연구에 필수적입니다. 화학 및 단백질 분석은 세포 또는 조직의 신진 대사를 평가하는 데 사용되지만 단일 세포 해상도 및 공간 정보가 부족합니다. 대사 플레이트 리더 기반 분석은 시간에 따른 샘플의 pH 및 산소 소비와 화학 물질에 의한 후속 대사 교란을 측정 할 수 있습니다. pH는 세포외 산성화 속도(ECAR)를 계산하는데 사용될 수 있으며, 이는 세포의 글리콜 활성에 대한 통찰력을 제공한다9. 2-[불소-18] 플루오로-D-글루코스 양전자 방출 단층 촬영 (FDG PET) 및 자기 공명 분광법 (MRS)을 포함한 전신 영상 방법은 대사 측정을 통해 종양 재발 및 약물 효능을 확인하기 위해 임상적으로 사용되는 비침습적 영상 방식입니다10,11,12,13,14.
FDG-PET는 방사성 표지된 글루코스 유사체인 FDG의 조직 흡수를 이미지화한다. 주변 조직에 비해 종양에 의한 FDG-PET의 흡수 증가는 바르부르크 효과12,13에 기인한다. MRS 이미지는 13C 및 31P와 같은 신진 대사에 사용되는 분자의 일반적인 핵을 이미지화하고 운동이나 식사와 같은 자극에 반응하여 신진 대사가 어떻게 변하는지에 대한 동적 정보를 얻을 수 있습니다14. FDG-PET 및 MRS가 임상적으로 사용될 수 있지만, 이들 기술은 종양내 이질성을 해결하기 위한 공간 분해능이 부족하다. 마찬가지로, 산소 소비량 측정은 세포의 벌크 집단에 대해 이루어진다. 자가 형광 이미징은 이러한 기술의 공간 분해능 장애물을 극복하고 세포 대사를 정량화하는 비침습적 방법을 제공합니다.
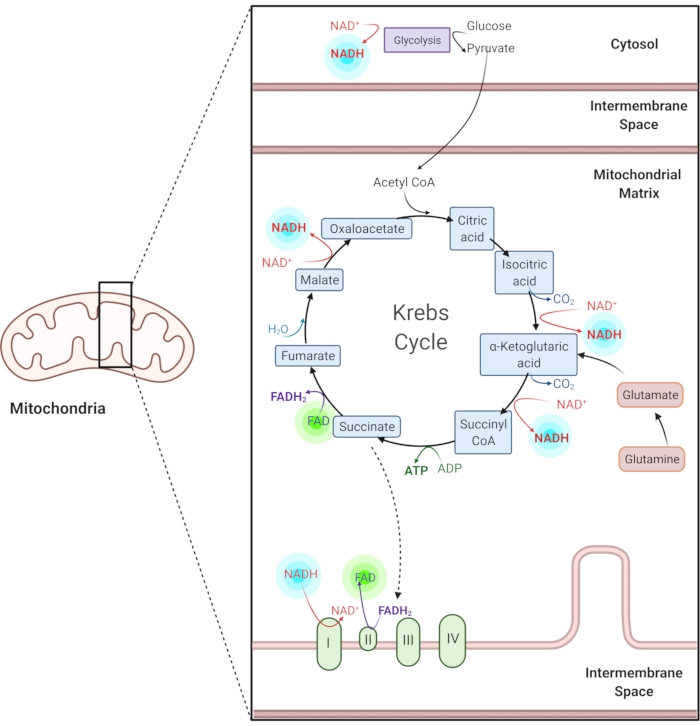
도 1: 일반적인 대사 경로에서의 NADH 및 FAD. NADH 및 FAD는 당분해, 크렙스 사이클 및 전자 수송 사슬에 사용되는 조효소이다. 이들 분자의 자가형광 이미징은 세포 대사에 대한 정보를 제공한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
환원된 니코틴아미드 아데닌(phosphate) 디뉴클레오타이드(NAD(P)H) 및 산화된 플라빈 아데닌 디뉴클레오타이드(FAD)는 당분해, 산화적 인산화 및 글루타미노분해를 포함하는 대사 반응의 조효소이다(도 1). NAD(P)H 및 FAD는 모두 자기형광성이며 형광 영상1,15에 내인성 대비를 제공합니다. NADPH는 NADH와 유사한 형광 특성을 갖는다. 이 때문에 NAD(P)H는 NADH와 NADPH2,16의 결합 신호를 나타내는 데 자주 사용됩니다.
형광 수명 이미징 (FLIM)은 형광 수명 또는 형광단이 여기된 상태에 있는 시간을 정량화합니다. 형광 수명은 형광단의 미세 환경에 반응하고 세포 대사에 대한 정보를 제공합니다17. NAD(P)H 및 FAD는 단백질 결합 또는 자유 형태에서 세포 내에 존재할 수 있으며, 이들 각각은 상이한 수명을 갖는다. 유리 NAD(P)H는 단백질 결합 NAD(P)H보다 짧은 수명을 갖는다; 반대로, 무료 FAD는 바운드 FAD18,19보다 수명이 길다. 수명 및 수명 성분 가중치는 Eq. (1)20을 통해 형광 수명 감쇠 데이터로부터 정량화될 수 있다:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq(1)은 시간의 함수로서 정규화된 형광 강도를 나타낸다. 이 방정식의 α 1과 α 2는 각각 짧은 수명과 긴 수명(α 1+ α 2=1)의 비례 성분을 나타내고, τ1과 τ2는 각각 짧은 수명과 긴 수명을 나타내며, C는 배경광7,20을 설명합니다. 여기서 τm으로 표현되는 진폭 가중 수명은 Eq. (2)를 사용하여 계산됩니다.
τm= α 1τ1+ α 2τ2 (2)
평균 수명은 형광단의 강도 붕괴에 대한 평균 “t”를 계산함으로써 계산될 수 있으며, 이는 2-지수 붕괴에 대해 Eq. (3)17,21에 의해 도시된다.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
형광 강도 이미지는 형광 수명 붕괴를 통합함으로써 수명 이미지로부터 계산될 수 있다. 자가 형광 이미징은 세포 내 해상도에서 살아있는 세포의 신진 대사를 특성화하는 데 사용할 수있는 비파괴적이고 라벨이없는 방법입니다. 광학 산화 환원 비율은 셀의 화학적 산화 환원 상태에 대한 광학 아날로그 메트릭을 제공하며 NAD (P) H 및 FAD 강도의 비율로 계산됩니다. 광학 산화 환원 비율을 계산하는 공식은 표준화되지 않았지만22,23,24,25, NAD (P) H 및 FAD의 결합 된 강도에 대한 FAD의 강도로 정의됩니다. 이 정의는 분모의 합산 강도가 0과 1 사이의 메트릭을 정규화하고 시안화물 억제의 예상 결과가 산화 환원 비율의 감소이기 때문에 사용됩니다. 유리 NAD(P)H 및 FAD의 형광 수명은 pH, 온도, 산소에 대한 근접성 및 삼투압을 포함한 대사 용매 미세환경의 변화에 대한 통찰력을 제공합니다17.
NAD(P)H 및 FAD의 결합된 분획의 형광 수명의 변화는 대사 경로 이용률 및 기질-특이적 대사26을 나타낼 수 있다. 성분 중량은 조효소18,19의 결합 분획에 대한 자유의 변화에 대해 해석될 수 있다. 전체적으로, 이러한 정량적 자가형광 수명 측정은 세포 대사의 분석을 허용하고, 자가형광 이미징은 정상 조직으로부터 신생물을 확인하는데 사용되어왔다.27,28, 줄기 세포의 특성화29,30, 면역 세포 기능 평가31,32,33,34,35, 신경 활성 측정36, 37,38, 유방암 및 두경부암과 같은 암 유형에서의 약물 효능 이해21,39,40,41,42. 고해상도 자가형광 이미징은 단일 세포 분석 및 집단내 이질성의 정량화를 위한 이미지 세분화와 결합될 수 있다43,44,45,46,47.
NAD(P)H 및 FAD는 강도 또는 수명 이미징을 위해 구성된 단일 광자 또는 다중 광자 형광 현미경으로 이미징할 수 있습니다. 단일 광자 현미경의 경우, NAD(P)H 및 FAD는 일반적으로 이러한 파장에서 일반적인 레이저 소스로 인해 각각 375-405nm 및 488nm의 파장에서 여기됩니다48. 이광자 형광 여기에서, NAD(P)H 및 FAD는 각각 대략 700 내지 750 nm 및 700 내지 900 nm의 파장에서 여기할 것이다(15,49). 일단 형광단이 여기되면, NAD(P)H 및 FAD는 각각 ~410nm에서 ~490nm, ~510nm에서 ~640nm 사이의 파장에서 광자를 방출한다15. NAD(P)H 및 FAD 최대 방출 파장은 각각 약 450nm 및 535nm(48)이다.
그들의 상이한 여기 및 방출 파장 때문에, 두 대사 조효소의 형광은 분광적으로 단리될 수 있다. NAD(P)H 및 FAD의 스펙트럼 특성에 대한 이해는 자가형광 이미징 프로토콜의 설계 및 최적화에 필요합니다. 시안화물은 전자 수송 사슬 (ETC) 착물 IV 억제제이다. 시안화물이 세포 대사에 미치는 영향과 세포 내 NAD(P)H 및 FAD의 자가형광 강도 및 수명은 잘 특성화되어 있습니다27,40. 따라서 시안화물 섭동 실험은 NAD(P)H 및 FAD 이미징 프로토콜을 검증하는 효과적인 수단입니다. 성공적인 시안화물 실험은 NAD(P)H 및 FAD 이미징 프로토콜이 알려지지 않은 그룹이나 교란의 신진대사를 평가하는 데 사용될 수 있다는 확신을 제공합니다.
Protocol
Representative Results
Discussion
자가 형광 강도 및 평생 이미징은 세포의 신진 대사를 평가하기 위해 널리 사용되어 왔습니다.21,55. FLIM은 고분해능이므로 단일 세포를 해결하는데, 이는 세포 이질성이 종양 공격성 및 약물 내성에 기여하기 때문에 암 연구에 중요합니다.7,39,41,44,45,46,58.<sup…
Disclosures
The authors have nothing to disclose.
Acknowledgements
자금 출처에는 텍사스 암 예방 및 연구소 (CPRIT RP200668)와 텍사스 A & M 대학이 포함됩니다. 그림 1은 BioRender.com 로 작성되었습니다.
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
References
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. 암 연구학. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. 암 연구학. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. 암 연구학. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

