Imagerie par autofluorescence pour évaluer le métabolisme cellulaire
Summary
Ce protocole décrit l’imagerie par fluorescence et l’analyse des coenzymes métaboliques endogènes, du dinucléotide réduit en nicotinamide adénine (phosphate) (NAD(P)H) et du dinucléotide de flavine adénine oxydé (FAD). L’imagerie par autofluorescence du NAD(P)H et du FAD fournit une méthode non destructive sans étiquette pour évaluer le métabolisme cellulaire.
Abstract
Le métabolisme cellulaire est le processus par lequel les cellules génèrent de l’énergie, et de nombreuses maladies, y compris le cancer, sont caractérisées par un métabolisme anormal. La réduction de la nicotinamide adénine (phosphate), du dinucléotide (NAD(P)H) et de la flavine adénine dinucléotide (FAD) oxydée sont des coenzymes de réactions métaboliques. Le NAD(P)H et le FAD présentent une autofluorescence et peuvent être isolés spectralement par des longueurs d’onde d’excitation et d’émission. Les deux coenzymes, NAD(P)H et FAD, peuvent exister dans une configuration libre ou liée aux protéines, chacune ayant une durée de vie de fluorescence distincte – le temps pendant lequel le fluorophore reste à l’état excité. L’imagerie par durée de vie de fluorescence (FLIM) permet de quantifier l’intensité de fluorescence et la durée de vie du NAD(P)H et du FAD pour une analyse sans étiquette du métabolisme cellulaire. L’intensité de fluorescence et la durée de vie des microscopes peuvent être optimisés pour l’imagerie NAD(P)H et FAD en sélectionnant les longueurs d’onde d’excitation et d’émission appropriées. Les perturbations métaboliques par le cyanure vérifient les protocoles d’imagerie par autofluorescence pour détecter les changements métaboliques dans les cellules. Cet article démontrera la technique d’imagerie par autofluorescence du NAD(P)H et du FAD pour mesurer le métabolisme cellulaire.
Introduction
Le métabolisme est le processus cellulaire de production d’énergie. Le métabolisme cellulaire englobe de multiples voies, y compris la glycolyse, la phosphorylation oxydative et la glutaminolyse. Les cellules saines utilisent ces voies métaboliques pour générer de l’énergie pour la prolifération et la fonction, comme la production de cytokines par les cellules immunitaires. De nombreuses maladies, y compris les troubles métaboliques, le cancer et la neurodégénérescence, sont caractérisées par une altération du métabolisme cellulaire1. Par exemple, certains types de cellules cancéreuses ont des taux élevés de glycolyse, même en présence d’oxygène, pour générer des molécules pour la synthèse d’acides nucléiques, de protéines et de lipides2,3. Ce phénomène, connu sous le nom d’effet Warburg, est une caractéristique de nombreux types de cancer, y compris le cancer du sein, le cancer du poumon et les glioblastomes4. En raison des altérations du métabolisme cellulaire associées à la progression du cancer, le métabolisme cellulaire peut être un biomarqueur de substitution de la réponse aux médicaments5,6. De plus, la compréhension de l’efficacité des médicaments au niveau cellulaire est cruciale, car l’hétérogénéité cellulaire peut entraîner des réponses différentes aux médicaments chez les individus7,8.
Les technologies qui identifient et quantifient les changements dans le métabolisme cellulaire sont essentielles pour les études sur le cancer et la réponse aux médicaments. Les analyses chimiques et protéiques sont utilisées pour évaluer le métabolisme des cellules ou des tissus, mais manquent de résolution unicellulaire et d’informations spatiales. Les tests basés sur des lecteurs de plaques métaboliques peuvent mesurer le pH et la consommation d’oxygène dans l’échantillon au fil du temps et la perturbation métabolique subséquente par les produits chimiques. Le pH peut être utilisé pour calculer le taux d’acidification extracellulaire (ECAR), ce qui donne un aperçu de l’activité glycolytique des cellules9. Les méthodes d’imagerie du corps entier, y compris la tomographie par émission de positons 2-[fluor-18] fluoro-D-glucose (FDG PET) et la spectroscopie par résonance magnétique (MRS), sont des modalités d’imagerie non invasives utilisées cliniquement pour identifier la récidive tumorale et l’efficacité des médicaments par des mesures métaboliques10,11,12,13,14.
FDG-PET image l’absorption tissulaire de FDG, un analogue du glucose radiomarqué. L’absorption accrue de FDG-PET par les tumeurs par rapport aux tissus environnants est due à l’effet Warburg12,13. MRS image les noyaux communs des molécules utilisées pour le métabolisme, telles que le 13C et le 31P, et peut obtenir des informations dynamiques sur la façon dont le métabolisme change en réponse à des stimuli, tels que l’exercice ou l’alimentation14. Bien que fdG-PET et MRS puissent être utilisés cliniquement, ces technologies n’ont pas la résolution spatiale nécessaire pour résoudre l’hétérogénéité intratumorale. De même, les mesures de consommation d’oxygène sont effectuées sur une population en vrac de cellules. L’imagerie par autofluorescence surmonte l’obstacle de résolution spatiale de ces technologies et fournit une méthode non invasive de quantification du métabolisme cellulaire.
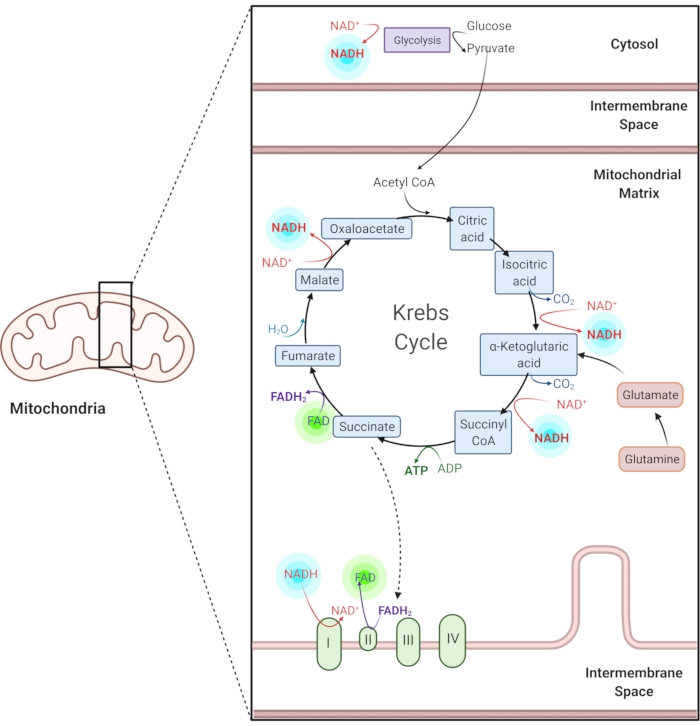
Figure 1 : NADH et FAD dans les voies métaboliques courantes. Le NADH et le FAD sont des coenzymes utilisées dans la glycolyse, le cycle de Krebs et la chaîne de transport d’électrons. L’imagerie par autofluorescence de ces molécules fournit des informations sur le métabolisme cellulaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La nicotinamide adénine (phosphate) dinucléotide (NAD(P)H) réduite et la flavine adénine dinucléotide (FAD) oxydée sont des coenzymes de réactions métaboliques, y compris la glycolyse, la phosphorylation oxydative et la glutaminolyse (Figure 1). Le NAD(P)H et le FAD sont tous deux autofluorescents et fournissent un contraste endogène pour l’imagerie par fluorescence1,15. Le NADPH a des propriétés fluorescentes similaires à celles du NADH. Pour cette raison, nad(P)H est souvent utilisé pour représenter le signal combiné du NADH et du NADPH2,16.
L’imagerie de la durée de vie de la fluorescence (FLIM) quantifie la durée de vie de la fluorescence ou le temps pendant lequel un fluorophore est à l’état excité. Les durées de vie de la fluorescence sont sensibles au microenvironnement des fluorophores et fournissent des informations sur le métabolisme cellulaire17. Le NAD(P)H et le FAD peuvent exister dans les cellules dans des conformations liées aux protéines ou libres, chacune ayant une durée de vie différente. Le NAD(P)H libre a une durée de vie plus courte que le NAD(P)H lié aux protéines; à l’inverse, le FAD libre a une durée de vie plus longue que le FAD lié18,19. Les durées de vie et les poids des composants à vie peuvent être quantifiés à partir des données de désintégration de la durée de vie de la fluorescence via Eq. (1)20:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) représente l’intensité de fluorescence normalisée en fonction du temps. Les α 1 et α 2 de cette équation représentent les composantes proportionnelles des durées de vie courtes et longues (α 1+ α 2=1), respectivement, τ1 et τ2 représentent les durées de vie courtes et longues, respectivement, et C représente la lumière de fond7,20. La durée de vie pondérée en fonction de l’amplitude, représentée ici par τm, est calculée à l’aide de Eq. (2).
τm= α 1τ1+ α 2τ2 (2)
Une durée de vie moyenne peut être calculée en faisant la moyenne de « t » sur la désintégration d’intensité du fluorophore, qui pour une désintégration à deux exponentielles est montrée par Eq. (3)17,21.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Une image d’intensité de fluorescence peut être calculée à partir de l’image de durée de vie en intégrant la désintégration de la durée de vie de fluorescence. L’imagerie par autofluorescence est une méthode non destructive et sans étiquette qui peut être utilisée pour caractériser le métabolisme des cellules vivantes à une résolution subcellulaire. Le rapport redox optique fournit une métrique analogique optique de l’état redox chimique de la cellule et est calculé comme le rapport des intensités NAD(P)H et FAD. Bien que la formule de calcul du rapport redox optique ne soit pas normalisée22,23,24,25, elle est définie ici comme l’intensité de la DCP par rapport aux intensités combinées de NAD(P)H et de FAD. Cette définition est utilisée parce que l’intensité totale dans le dénominateur normalise la métrique entre 0 et 1, et le résultat attendu de l’inhibition du cyanure est une diminution du rapport redox. Les durées de vie en fluorescence du NAD(P)H libre et du FAD donnent un aperçu des changements dans le microenvironnement du solvant métabolique, y compris le pH, la température, la proximité de l’oxygène et l’osmolarité17.
Les changements dans la durée de vie de fluorescence des fractions liées de NAD(P)H et de FAD peuvent indiquer l’utilisation de la voie métabolique et le métabolisme spécifique du substrat26. Les poids des composants peuvent être interprétés pour les changements dans la fraction libre à la fraction liée des coenzymes18,19. Dans l’ensemble, ces mesures quantitatives de la durée de vie de l’autofluorescence permettent l’analyse du métabolisme cellulaire, et l’imagerie par autofluorescence a été utilisée pour identifier les néoplasmes des tissus normaux27,28, caractériser les cellules souches29,30, évaluer la fonction des cellules immunitaires31,32,33,34,35, évaluer l’activité neurologique36, 37,38, et comprendre l’efficacité des médicaments dans les types de cancer tels que le cancer du sein et le cancer de la tête et du cou21,39,40,41,42. L’imagerie par autofluorescence à haute résolution peut être combinée à la segmentation d’images pour l’analyse unicellulaire et la quantification de l’hétérogénéité intrapopulation43,44,45,46,47.
Le NAD(P)H et le FAD peuvent être imagés sur des microscopes à fluorescence monophotonique ou multiphotonique configurés pour l’imagerie d’intensité ou de durée de vie. Pour les microscopes monophotoniques, le NAD(P)H et le FAD sont généralement excités à des longueurs d’onde de 375-405 nm et 488 nm, respectivement, en raison de sources laser courantes à ces longueurs d’onde48. Dans l’excitation par fluorescence à deux photons, le NAD(P)H et le FAD s’exciteront à des longueurs d’onde d’environ 700 à 750 nm et de 700 à 900 nm, respectivement15,49. Une fois les fluorophores excités, le NAD(P)H et le FAD émettent des photons à des longueurs d’onde comprises respectivement entre ~410 nm et ~510 nm à ~640 nm15. Les longueurs d’onde d’émission maximales NAD(P)H et FAD sont respectivement d’environ 450 nm et 535 nm48.
En raison de leurs différentes longueurs d’onde d’excitation et d’émission, la fluorescence des deux coenzymes métaboliques peut être isolée spectralement. Une compréhension des caractéristiques spectrales du NAD(P)H et du FAD est nécessaire pour la conception et l’optimisation des protocoles d’imagerie par autofluorescence. Le cyanure est un inhibiteur IV du complexe IV de la chaîne de transport d’électrons (ETC). Les effets du cyanure sur le métabolisme cellulaire et les intensités d’autofluorescence et la durée de vie du NAD(P)H et du FAD dans les cellules sont bien caractérisés27,40. Par conséquent, une expérience de perturbation du cyanure est un moyen efficace de valider les protocoles d’imagerie NAD(P)H et FAD. Une expérience réussie sur le cyanure permet de s’assurer que le protocole d’imagerie NAD(P)H et FAD peut être utilisé pour évaluer le métabolisme de groupes inconnus ou de perturbations.
Protocol
Representative Results
Discussion
L’intensité de l’autofluorescence et l’imagerie à vie ont été largement utilisées pour évaluer le métabolisme dans les cellules21,55. FLIM est à haute résolution et résout donc les cellules individuelles, ce qui est important pour les études sur le cancer car l’hétérogénéité cellulaire contribue à l’agression tumorale et à la résistance aux médicaments7,39,41,44,45,46,58.<sup class="xref…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Les sources de financement comprennent le Cancer Prevention and Research Institute of Texas (CPRIT RP200668) et la Texas A & M University. La figure 1 a été créée avec BioRender.com.
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
References
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. 암 연구학. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. 암 연구학. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. 암 연구학. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. 암 연구학. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. 암 연구학. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

