Untersuchung des Membranproteintransports in Drosophila-Photorezeptorzellen unter Verwendung von eGFP-markierten Proteinen
Summary
Hier werden nicht-invasive Methoden zur Lokalisation von Photorezeptormembranproteinen und zur Beurteilung der Netzhautdegeneration im Drosophila-Facettenauge mittels eGFP-Fluoreszenz beschrieben.
Abstract
Der Membranproteintransport reguliert den Einbau und die Entfernung von Rezeptoren und Ionenkanälen in die Plasmamembran. Dieser Prozess ist grundlegend wichtig für die Zellfunktion und die Zellintegrität von Neuronen. Drosophila-Photorezeptorzellen sind zu einem Modell für die Untersuchung des Membranproteintransports geworden. Neben Rhodopsin, das bei Beleuchtung aus der Photorezeptormembran internalisiert und abgebaut wird, weist der transiente Rezeptorpotential-ähnliche (TRPL) Ionenkanal in Drosophila eine lichtabhängige Translokation zwischen der rhabdomeralen Photorezeptormembran (wo es sich im Dunkeln befindet) und dem Photorezeptorzellkörper (zu dem es bei Beleuchtung transportiert wird) auf. Dieser intrazelluläre Transport von TRPL kann auf einfache und nicht-invasive Weise untersucht werden, indem eGFP-markiertes TRPL in Photorezeptorzellen exprimiert wird. Die eGFP-Fluoreszenz kann dann entweder in der tiefen Pseudopupille oder mittels Wasserimmersionsmikroskopie beobachtet werden. Diese Methoden ermöglichen den Nachweis der Fluoreszenz im intakten Auge und sind daher nützlich für Hochdurchsatz-Assays und genetische Screens für Drosophila-Mutanten , die bei der TRPL-Translokation defekt sind. Hier werden die Herstellung von Fliegen, die mikroskopischen Techniken sowie Quantifizierungsmethoden, mit denen diese lichtgetriggerte Translokation von TRPL untersucht wird, ausführlich erläutert. Diese Methoden können auch für Handelsstudien an anderen Drosophila-Photorezeptorproteinen , zum Beispiel Rhodopsin, angewendet werden. Darüber hinaus können diese Methoden durch die Verwendung von eGFP-markierten rhabdomeralen Proteinen verwendet werden, um die Degeneration von Photorezeptorzellen zu beurteilen.
Introduction
Durch die Abgabe und Entfernung von Proteinen zur und von der Plasmamembran steuert der Membranproteintransport in Neuronen die Plasmamembranausrüstung mit Rezeptoren sowie Ionenkanälen und reguliert dadurch die neuronale Funktion. Fehlregulationen oder Defekte im Proteintransport haben typischerweise schädliche Auswirkungen auf Zellen und führen zu neuronaler Degeneration. Beim Menschen kann dies neurodegenerative Erkrankungen wie Alzheimer und Parkinson oder Retinitis pigmentosa1 verursachen. Photorezeptoren im Facettenauge von Drosophila melanogaster sind zu einem In-vivo-Modellsystem für die Untersuchung des Membranproteintransportsgeworden 2. Dies liegt nicht nur an der genetischen Vielseitigkeit von Drosophila , die effektive genetische Screens ermöglicht, sondern auch daran, dass alle wesentlichen Bestandteile der lichtabsorbierenden Photorezeptormembran sehr detailliert charakterisiert sind und effiziente mikroskopische Techniken zur Verfügung stehen, die auf das Fliegenauge angewendet werden können. Diese Techniken stehen im Mittelpunkt dieses Artikels.
In Drosophila-Photorezeptorzellen bildet die apikale Plasmamembran einen dicht gepackten Stapel von Mikrovilli entlang einer Seite der Zelle, der als Rhabdomere bezeichnet wird. Die Rhabdomere der Photorezeptorzellen R1-6 sind in einem charakteristischen trapezförmigen Muster angeordnet, während die Photorezeptorzellen R7 und R8 ein einzelnes Rhabdomere in der Mitte diesesTrapezes 3 bilden. Der Membranproteintransport wird für einen regulierten Umsatz von rhabdomeralen Membranproteinen wie Rhodopsin und den lichtaktivierten Ionenkanälen TRP (transient receptor potential) und TRPL (TRP-like) benötigt, um die richtige Menge dieser Phototransduktionsproteine im Rhabdomere sicherzustellen. Photorezeptormembranproteine werden im endoplasmatischen Retikulum synthetisiert und über den Golgi-Apparat zum Rhabdomere transportiert. Nach der Aktivierung von Rhodopsin durch Licht kann ein Rhodopsinmolekül entweder durch Absorption eines zweiten Photons inaktiviert oder durch Clathrin-vermittelte Endozytose aus dem Rhabdomere entfernt werden. Endozytosiertes Rhodopsin wird entweder im Lysosom abgebaut oder in den Rhabdomerezurückgeführt 4,5. Der Ionenkanal TRPL wird nach Aktivierung der Phototransduktionskaskade ebenfalls internalisiert und durchläuft eine lichtabhängige Translokation zwischen dem Rhabdomere (wo er sich befindet, wenn Fliegen im Dunkeln gehalten werden) und einem ER-angereicherten Staufach im Zellkörper (zu dem er bei Beleuchtung innerhalb weniger Stunden transportiert wird)6,7,8,9,10 . Im Gegensatz zu endozytosiertem Rhodopsin werden nur geringe Mengen TRPL über den endolysosomalen Weg abgebaut, und der Großteil wird stattdessen intrazellulär gespeichert und bei dunkler Anpassung wieder in den Rhabdomer zurückgeführt6. TRPL kann somit zur Analyse des lichtgetriggerten Transports von Plasmamembranproteinen verwendet werden. Drosophila-Photorezeptorzellen werden auch zur Untersuchung der neuronalen Degeneration eingesetzt. Die Degeneration von Photorezeptorzellen wird häufig durch die Beurteilung der Struktur von Rhabdomeren bestimmt, die infolge degenerativer Prozesse zerfallen5.
Um die subzelluläre Lokalisation von TRPL und Rhodopsin in Photorezeptorzellen oder die Degeneration von Photorezeptorzellen zu untersuchen, wurden hier zwei Fluoreszenzmikroskopie-Methoden angewendet, die sich hinsichtlich Analysegeschwindigkeit und Auflösung unterscheiden. Eine sehr schnelle, nicht-invasive Methode, die für genetische Screens verwendet werden kann, jedoch mit einer begrenzten räumlichen Auflösung, ist der Nachweis der Fluoreszenz in der tiefen Pseudopupille (DPP). Das DPP ist ein optisches Phänomen von Arthropoden-Facettenaugen, dessen geometrischer Ursprung1971 von Franceschini und Kirschfeld ausführlich erklärt wurde 11. Kurz gesagt, auf mehreren optischen Ebenen unterhalb der Netzhautüberlagerung können Bilder von Rhabdomeren aus benachbarten Ommatidien beobachtet werden. Auf einer Fokusebene durch die Mitte der Augenkrümmung bilden diese überlagerten Projektionen ein Bild, das dem trapezförmigen Layout von Rhabdomeren in einem einzigen Ommatidium ähnelt, das nur um Größenordnungen größer ist. Dieses Phänomen kann auch unabhängig von der exogenen Expression von Fluoreszenzproteinen (z.B. TRPL::EGFP 8) beobachtet werden, was den Nachweis des DPP jedoch erleichtert (Abbildung 1A-A‘‘)12. Eine zweite nicht-invasive Methode ist die Wasserimmersionsmikroskopie, die auf der Abbildung fluoreszierend markierter Proteine beruht, nachdem der Dioptrienapparat der Augen optisch mit Wasser neutralisiert wurde (Abbildung 1B-C‘‘)12. Mit der Wasserimmersionsmethode kann die relative Menge an TRPL::eGFP in den Rhabdomeren oder im Zellkörper für einzelne Photorezeptorzellen quantitativ beurteilt werden. Darüber hinaus können nicht-translozierende fluoreszenzmarkierte Proteine verwendet werden, um die rhabdomerale Integrität zu bewerten und den zeitlichen Verlauf einer möglichen Degeneration quantitativ zu bestimmen, wie hier beschrieben.
Während Aufzeichnungen des DPP bei weitem die einfachste und schnellste dieser Methoden sind, ist die räumliche Auflösung der von ihnen generierten Daten begrenzt. Darüber hinaus gibt es zahlreiche Gründe, warum ein DPP fehlen kann, die für die DPP-Bildgebung selbst nicht unbedingt erkennbar sind. Da der DPP eine Summe mehrerer Ommatidien darstellt, gehen Informationen über einzelne Zellen verloren. Daher erfüllt die DPP-Bildgebung mit niedriger Auflösung eine wichtige Funktion beim Screening einer großen Anzahl von Fliegen, sollte jedoch im Allgemeinen von Aufnahmen mit höherer Auflösung mittels Wasserimmersionsmikroskopie gefolgt werden. Wasserimmersionsmikroskope erlauben Interpretationen über einzelne Zellen, Entwicklungsdefekte, Augenmorphologie, Proteinfehllokalisation oder Netzhautdegeneration sowie die Quantifizierung dieser Effekte. Dieses Protokoll beschreibt diese beiden Techniken im Detail.
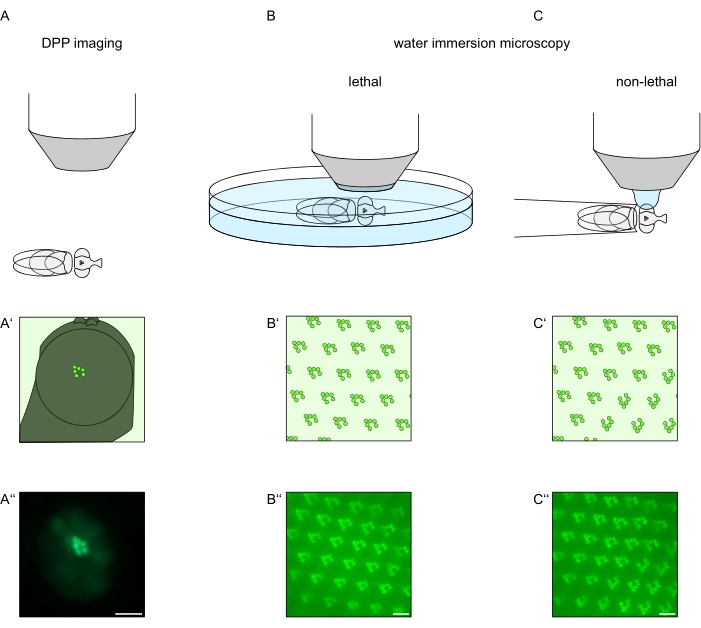
Abbildung 1: Übersicht über die in diesem Protokoll dargestellten Mikroskopievariationen für das Drosophila-Auge. Schematische Darstellungen und exemplarische Mikroaufnahmen der (A-A”) fluoreszierenden tiefen Pseudopupillenbildgebung (DPP), (B-B”) der tödlichen Wasserimmersionsmikroskopie von fluoreszierenden Rhabdomeren und (C-C‘‘) der nicht-tödlichen Wassertropfenmikroskopie von fluoreszierenden Rhabdomeren. Maßstabsleiste (A”): 100 μm. Maßstabsstäbe (B”–C‘‘): 10 μm Die Abbildung wurde gegenüber Referenz13 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protocol
Representative Results
Discussion
Die Anwendbarkeit von Fluoreszenzproteinen und die Einfachheit des Screenings durch DPP-Bildgebung und retinale Wasserimmersionsmikroskopie haben sich in vielen Gruppen12 als erfolgreich erwiesen. Ähnliche Strategien wie die hier vorgestellten wurden in mehreren genetischen Screens verwendet, um Defekte in der Rhodopsinexpression, Homöostase, Netzhautorganisation oder zellulären Integrität mit Hilfe von Rh1::eGFP17,18,19,20,21</su…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir danken unseren studentischen Forschern im Laufe der Jahre. Insbesondere Nina Meyer, Sibylle Mayer, Juliane Kaim und Laura Jaggy, deren Daten in diesem Protokoll als repräsentative Ergebnisse verwendet wurden. Die Forschung unserer hier vorgestellten Gruppe wurde durch Zuschüsse der Deutschen Forschungsgemeinschaft (Hu 839/2-4, Hu 839/7-1) an Armin Huber gefördert.
Materials
| 15 mL centrifuge tube | Greiner Bio-One | 188271 | |
| CO2 anaesthesia fly pad | Flystuff | 59-172 | |
| Cold light lamp (KL 1500 LCD) | Zeiss | ||
| Fiji/ImageJ | NIH | ||
| Fluorescence microscope with UV lamp, camera, filter set and software (AxioImager.Z1m, Axiocam 530 mono, 38 HE, ZEN2 blue edition) | Zeiss | ||
| Fluorescent tube (Lumilux T8, L 30W/840, 4000 K, G13) [1750 Lux, Ee470nm = 298 µW cm-2, Ee590nm = 215 µW cm-2] and [760 Lux, Ee470nm = 173 µW cm-2, Ee590nm = 147 µW cm-2] |
Osram | 4050300518039 | |
| Laboratory pipette (20-200 µL) | Eppendorf | ||
| Object slide | Roth | 0656.1 | |
| Petri dish (94 mm) | Greiner Bio-One | 633102 | |
| Pipette tips (200 µL) | Labsolute | 7695844 | |
| Plasticine (Blu-Tack) | Bostik | 30811745 | |
| Stereo microscope (SMZ445) | Nikon | ||
| Stereo microscope with UV lamp, camera, filer set and software (MZ16F, MC170 HD, GFP3, LAS 4.12) | Leica |
References
- Wang, X., Huang, T., Bu, G., Xu, H. Dysregulation of protein trafficking in neurodegeneration. Molecular Neurodegeneration. 9. 9, 31 (2014).
- Schopf, K., Huber, A. Membrane protein trafficking in Drosophila photoreceptor cells. European Journal of Cell Biology. 96 (5), 391-401 (2017).
- Hardie, R. C. The photoreceptor array of the dipteran retina. Trends in Neurosciences. 9, 419-423 (1986).
- Wang, T., Montell, C. Phototransduction and retinal degeneration in Drosophila. Pflügers Archiv – European Journal of Physiology. 454 (5), 821-847 (2007).
- Xiong, B., Bellen, H. J. Rhodopsin homeostasis and retinal degeneration: lessons from the fly. Trends in Neurosciences. 36 (11), 652-660 (2013).
- Bähner, M., et al. Light-regulated subcellular translocation of Drosophila TRPL channels induces long-term adaptation and modifies the light-induced current. Neuron. 34 (1), 83-93 (2002).
- Cronin, M. A., Lieu, M. -. H., Tsunoda, S. Two stages of light-dependent TRPL-channel translocation in Drosophila photoreceptors. Journal of Cell Science. 119, 2935-2944 (2006).
- Meyer, N., Joel-Almagor, T., Frechter, S., Minke, B., Huber, A. Subcellular translocation of the eGFP-tagged TRPL channel in Drosophila photoreceptors requires activation of the phototransduction cascade. Journal of Cell Science. 119, 2592-2603 (2006).
- Oberegelsbacher, C., Schneidler, C., Voolstra, O., Cerny, A., Huber, A. The Drosophila TRPL ion channel shares a Rab-dependent translocation pathway with rhodopsin. European Journal of Cell Biology. 90 (8), 620-630 (2011).
- Wagner, K., Smylla, T. K., Lampe, M., Krieg, J., Huber, A. Phospholipase D and retromer promote recycling of TRPL ion channel via the endoplasmic reticulum. Traffic. , (2021).
- Franceschini, N., Kirschfeld, K. Les phénoménes de pseudopupille dans l’oeil compose de Drosophila. Kybernetik. 9 (5), 159-182 (1971).
- Pichaud, F., Desplan, C. A new visualization approach for identifying mutations that affect differentiation and organization of the Drosophila ommatidia. Development. 128 (6), 815-826 (2001).
- Smylla, T. K., Wagner, K., Huber, A. Application of fluorescent proteins for functional dissection of the Drosophila visual system. International Journal of Molecular Sciences. 22 (16), (2021).
- Meyer, N. Mechanisms of the light-dependent translocation of the ion channel TRPL in the photoreceptors of Drosophila melanogaster: Dissertation for obtaining the academic degree Dr. rer. nat. University of Karlsruhe (TH). , (2005).
- Meyer, N., Oberegelsbacher, C., Dürr, T. D., Schäfer, A., Huber, A. An eGFP-based genetic screen for defects in light-triggered subcelluar translocation of the Drosophila photoreceptor channel TRPL. Fly. 2 (1), 36-46 (2008).
- Zheng, L., Carthew, R. W. Lola regulates cell fate by antagonizing Notch induction in the Drosophila eye. Mechanisms of Development. 125 (1-2), 18-29 (2008).
- Hibbard, K. L., O’Tousa, J. E. A role for the cytoplasmic DEAD box helicase Dbp21E2 in rhodopsin maturation and photoreceptor viability. Journal of Neurogenetics. 26 (2), 177-188 (2012).
- Huang, Y., Xie, J., Wang, T. A Fluorescence-based genetic screen to study retinal degeneration in Drosophila. PloS One. 10 (12), 0144925 (2015).
- Zhao, H., Wang, J., Wang, T. The V-ATPase V1 subunit A1 is required for rhodopsin anterograde trafficking in Drosophila. Molecular Biology of the Cell. 29 (13), 1640-1651 (2018).
- Xiong, L., et al. ER complex proteins are required for rhodopsin biosynthesis and photoreceptor survival in Drosophila and mice. Cell Death and Differentiation. 27 (2), 646-661 (2020).
- Zhao, H., Wang, T. PE homeostasis rebalanced through mitochondria-ER lipid exchange prevents retinal degeneration in Drosophila. PLoS Genetics. 16 (10), 1009070 (2020).
- Cerny, A. C., et al. The GTP- and phospholipid-binding protein TTD14 regulates trafficking of the TRPL ion channel in Drosophila photoreceptor cells. PLoS Genetics. 11 (10), 1005578 (2015).
- Richter, D. Structural and functional analyses of TRP ion channels in the photoreceptor cells of Drosophila melanogaster: Dissertation for obtaining the academic degree Dr. rer, nat. University of Karlsruhe (TH). , (2007).
- Gambis, A., Dourlen, P., Steller, H., Mollereau, B. Two-color in vivo imaging of photoreceptor apoptosis and development in Drosophila. 발생학. 351 (1), 128-134 (2011).
- Hardie, R. C., Liu, C. -. H., Randall, A. S., Sengupta, S. In vivo tracking of phosphoinositides in Drosophila photoreceptors. Journal of Cell Science. 128 (23), 4328-4340 (2015).
- Chakrabarti, P., et al. A dPIP5K dependent pool of phosphatidylinositol 4,5 bisphosphate (PIP2) is required for G-protein coupled signal transduction in Drosophila photoreceptors. PLoS Genetics. 11 (1), 1004948 (2015).

