आणविक चैपरोन Hsp90 और इसके ग्राहक प्रोटीन किनेज Cdc37 के बीच बायोमोलेक्यूलर इंटरैक्शन की खोज फील्ड-इफेक्ट बायोसेंसिंग तकनीक का उपयोग करके
Summary
फील्ड-इफेक्ट बायोसेंसिंग (फेब) बायोमोलेक्यूलर इंटरैक्शन का पता लगाने के लिए एक लेबल-मुक्त तकनीक है। यह graphene biosensor के माध्यम से विद्युत प्रवाह को मापता है जिसके लिए बाध्यकारी लक्ष्य ों को स्थिर किया जाता है। फेब तकनीक का उपयोग एचएसपी 90 और सीडीसी 37 के बीच बायोमोलेक्यूलर इंटरैक्शन का मूल्यांकन करने के लिए किया गया था और दो प्रोटीनों के बीच एक मजबूत बातचीत का पता लगाया गया था।
Abstract
बायोमोलेक्यूलर इंटरैक्शन कार्यात्मक रूप से प्रासंगिक जैविक घटनाओं को विनियमित और समन्वयित करके कई सेलुलर प्रक्रियाओं में बहुमुखी भूमिका निभाते हैं। प्रोटीन, कार्बोहाइड्रेट, विटामिन, फैटी एसिड, न्यूक्लिक एसिड और एंजाइम जैसे बायोमोलेक्यूल्स जीवित प्राणियों के मौलिक निर्माण खंड हैं; वे जीवन की घटनाओं के असंख्य सिंक्रनाइज़ करने के लिए biosystems में जटिल नेटवर्क में इकट्ठा. प्रोटीन आमतौर पर अपने कार्यों को पूरा करने के लिए जटिल इंटरैक्टोम नेटवर्क का उपयोग करते हैं; इसलिए सेलुलर और जीव दोनों स्तरों पर कोशिकाओं में उनके महत्व को उजागर करने के लिए इस तरह की बातचीत का मूल्यांकन करना अनिवार्य है। इस लक्ष्य की ओर, हम विशिष्ट बायोमोलेक्यूलर इंटरैक्शन निर्धारित करने के लिए एक तेजी से उभरती हुई तकनीक, फील्ड-इफेक्ट बायोसेंसिंग (एफईबी) पेश करते हैं। FEB एक बेंचटॉप, लेबल-मुक्त, और विश्वसनीय बायोमोलेक्यूलर डिटेक्शन तकनीक है जो विशिष्ट इंटरैक्शन को निर्धारित करने के लिए है और उच्च गुणवत्ता वाले इलेक्ट्रॉनिक-आधारित बायोसेंसर का उपयोग करता है। FEB तकनीक अपने बायोसेंसर सतह पर उपयोग किए जाने वाले बायोकंपैटिबल नैनोमैटेरियल्स के कारण नैनोमोलर रेंज में इंटरैक्शन की निगरानी कर सकती है। अवधारणा के प्रमाण के रूप में, गर्मी सदमे प्रोटीन 90 (Hsp90) और सेल डिवीजन चक्र 37 (Cdc37) के बीच प्रोटीन-प्रोटीन इंटरैक्शन (पीपीआई) को स्पष्ट किया गया था। Hsp90 एक एटीपी-निर्भर आणविक चैपरोन है जो कई प्रोटीनों के तह, स्थिरता, परिपक्वता और गुणवत्ता नियंत्रण में एक आवश्यक भूमिका निभाता है, जिससे कई महत्वपूर्ण सेलुलर कार्यों को विनियमित किया जाता है। Cdc37 को प्रोटीन किनेज-विशिष्ट आणविक चैपरोन के रूप में माना जाता है, क्योंकि यह विशेष रूप से अपने डाउनस्ट्रीम सिग्नल ट्रांसडक्शन मार्गों को विनियमित करने के लिए Hsp90 में प्रोटीन किनेसेस को पहचानता है और भर्ती करता है। इस प्रकार, Cdc37 को Hsp90 का एक सह-चैपरोन माना जाता है। चैपरोन-किनेज मार्ग (Hsp90/ Cdc37 कॉम्प्लेक्स) सेलुलर विकास को बढ़ावा देने वाले कई दुर्दमताओं में हाइपर-सक्रिय है; इसलिए, यह कैंसर चिकित्सा के लिए एक संभावित लक्ष्य है। वर्तमान अध्ययन Hsp90 / Cdc37 मॉडल प्रणाली का उपयोग करके FEB प्रौद्योगिकी की दक्षता को दर्शाता है। FEB ने दो प्रोटीनों के बीच एक मजबूत PPI का पता लगाया (तीन स्वतंत्र प्रयोगों में 0.014 μM, 0.053 μM, और 0.072 μM केK D मान)। संक्षेप में, FEB एक लेबल-मुक्त और लागत प्रभावी पीपीआई डिटेक्शन प्लेटफ़ॉर्म है, जो तेज़ और सटीक माप प्रदान करता है।
Introduction
Biomolecular इंटरैक्शन:
प्रोटीन जीवों के आवश्यक हिस्से हैं और सेल चयापचय, सेल संरचना, सेल सिग्नलिंग, प्रतिरक्षा प्रतिक्रियाओं, सेल आसंजन, और अधिक जैसे कई आणविक मार्गों में भाग लेते हैं। जबकि कुछ प्रोटीन स्वतंत्र रूप से अपने कार्य (ओं) को निष्पादित करते हैं, अधिकांश प्रोटीन उचित जैविक गतिविधि के समन्वय के लिए एक बाध्यकारी इंटरफ़ेस का उपयोग करके अन्यप्रोटीनों के साथ बातचीत करते हैं।
बायोमोलेक्यूलर इंटरैक्शन को मुख्य रूप से2 में शामिल प्रोटीन की विशिष्ट संरचनात्मक और कार्यात्मक विशेषताओं के आधार पर वर्गीकृत किया जा सकता है, उदाहरण के लिए, प्रोटीन सतहों, जटिल स्थिरता, या इंटरैक्शन की दृढ़ता के आधार पर3। बायोमोलेक्यूलर इंटरैक्शन में आवश्यक प्रोटीन और उनकी भूमिकाओं की पहचान करना आणविक स्तर4 पर जैव रासायनिक तंत्र को समझने के लिए महत्वपूर्ण है। वर्तमान में, इन इंटरैक्शन5 का पता लगाने के लिए विभिन्न दृष्टिकोण हैं: इन विट्रो6 में, सिलिको7 में, लाइव कोशिकाओं में8, पूर्व वीवो9, और विवो10 में प्रत्येक की अपनी ताकत और कमजोरियां हैं।
विवो assays में एक प्रयोगात्मक उपकरण11 के रूप में पूरे जानवर का उपयोग कर प्रदर्शन कर रहे हैं, और टीवह पूर्व vivo assays ऊतक अर्क या पूरे अंगों (जैसे, दिल, मस्तिष्क, जिगर) पर एक नियंत्रित बाहरी वातावरण में प्राकृतिक परिस्थितियों में न्यूनतम परिवर्तन प्रदान करके प्रदर्शन कर रहे हैं. विवो और पूर्व विवो अध्ययनों का सबसे आम अनुप्रयोग उनकी समग्र सुरक्षाऔर प्रभावकारिता सुनिश्चित करके मानव परीक्षणों से पहले संभावित औषधीय एजेंटों के फार्माकोकाइनेटिक्स, फार्माकोडायनेमिक्स और विषाक्तता प्रभावों का मूल्यांकन करना है।
जीवित कोशिकाओं के भीतर बायोमोलेक्यूलर इंटरैक्शन का भी पता लगाया जा सकता है। इमेजिंग लाइव कोशिकाएं हमें गतिशील इंटरैक्शन का निरीक्षण करने की अनुमति देती हैं क्योंकि वे एक विशेष जैव रासायनिक मार्ग13 की प्रतिक्रियाओं को निष्पादित करते हैं। इसके अलावा, पता लगाने की तकनीक, जैसे कि बायोल्यूमिनेसेंस या प्रतिदीप्ति अनुनाद ऊर्जा हस्तांतरण, इस बारे में जानकारी प्रदान कर सकती है कि ये इंटरैक्शन सेल14 के भीतर कहां और कब होते हैं। यद्यपि लाइव कोशिकाओं में पता लगाने से महत्वपूर्ण विवरण मिलते हैं, ये पहचान के तरीके प्रकाशिकी और लेबल पर भरोसा करते हैं, जो देशी जीव विज्ञान को प्रतिबिंबित नहीं कर सकते हैं; वे इन विट्रो विधियों की तुलना में भी कम नियंत्रित होते हैं और15 प्रदर्शन करने के लिए विशेष विशेषज्ञता की आवश्यकता होती है।
सिलिको कम्प्यूटेशनल विधियों में मुख्य रूप से इन विट्रो प्रयोगों से पहले लक्ष्य अणुओं की बड़े पैमाने पर स्क्रीनिंग के लिए उपयोग किया जाता है। कम्प्यूटेशनल भविष्यवाणी के तरीके, कंप्यूटर-आधारित डेटाबेस, आणविक डॉकिंग, मात्रात्मक संरचना-गतिविधि संबंध, और अन्य आणविक गतिशीलता सिमुलेशन दृष्टिकोण सिलिको टूल16 में अच्छी तरह से स्थापित हैं। श्रमसाध्य प्रयोगात्मक तकनीकों की तुलना में, सिलिको टूल्स में आसानी से उच्च संवेदनशीलता के साथ भविष्यवाणियां कर सकते हैं, लेकिन भविष्यवाणी प्रदर्शन में कम सटीकताके साथ 17।
इन विट्रो assays उनके मानक जैविक संदर्भ के बाहर सूक्ष्मजीवों या जैविक अणुओं के साथ प्रदर्शन कर रहे हैं। इन विट्रो विधियों के माध्यम से बायोमोलेक्यूलर इंटरैक्शन को चित्रित करना प्रोटीन कार्यों और सेल कामकाज के जटिल नेटवर्क के पीछे जीव विज्ञान को समझने के लिए महत्वपूर्ण है। पसंदीदा परख पद्धति प्रोटीन के आंतरिक गुणों, गतिज मूल्यों, और मोड और इंटरैक्शनकी तीव्रता 18,19 के अनुसार चुना जाता है।
Hsp90 / Cdc37 इंटरैक्शन:
चैपरोन-किनेज मार्ग, एचएसपी 90 और सीडीसी 37 को जोड़ता है, ट्यूमर जीव विज्ञान20 में एक आशाजनक चिकित्सीय लक्ष्य है। Hsp90 सेल चक्र नियंत्रण, प्रोटीन असेंबली, सेल अस्तित्व, और सिग्नलिंग मार्गों में एक केंद्रीय भूमिका निभाता है। प्रोटीन जो अपने कार्यों के लिए Hsp90 पर भरोसा करते हैं, उन्हें एक सह-चैपरोन के माध्यम से जटिलता के लिए Hsp90 तक पहुंचाया जाता है, जैसे कि Cdc37। Hsp90 / Cdc37 कॉम्प्लेक्स अधिकांश प्रोटीन किनेसेस की तह को नियंत्रित करता है और इंट्रासेल्युलर सिग्नलिंग नेटवर्क21 की भीड़ के लिए एक केंद्र के रूप में कार्य करता है। यह तीव्र मायलोब्लास्टिक ल्यूकेमिया, मल्टीपल मायलोमा और हेपेटोसेलुलर कार्सिनोमा22,23 सहित विभिन्न दुर्दमताओं में अपनी उन्नत अभिव्यक्ति के कारण एक आशाजनक एंटी-ट्यूमर लक्ष्य है।
आमतौर पर विट्रो बायोमोलेक्यूलर इंटरैक्शन डिटेक्शन तकनीकों में उपयोग किया जाता है
Co-immunoprecipitation (co-IP) एक ऐसी तकनीक है जो जैविक रूप से प्रासंगिक इंटरैक्शन की पहचान करने के लिए एंटीजन-एंटीबॉडी विशिष्टता पर निर्भरकरती है। इस विधि का प्राथमिक नुकसान कम-आत्मीयता इंटरैक्शन और गतिज मूल्यों का पता लगाने में असमर्थताहै। बायोफिजिकल तरीकों जैसे समतापी अनुमापन कैलोरीमेट्री (आईटीसी), सतह प्लास्मोन अनुनाद (एसपीआर), बायोलेयर इंटरफेरोमेट्री (बीएलआई), और फेब तकनीक को गतिज मूल्यों को निर्धारित करने के लिए प्राथमिकता दी जाती है।
आईटीसी एक बायोफिजिकल डिटेक्शन विधि है जो बायोमोलेक्यूलर इंटरैक्शन को चिह्नित करने के लिए एक पूर्ण थर्मोडायनामिक्स विश्लेषण के साथ बाध्यकारी ऊर्जा के निर्धारण पर आधारितहै। आईटीसी का प्राथमिक लाभ यह है कि इसे लक्ष्य प्रोटीन के किसी भी लेबलिंग या निर्धारण की आवश्यकता नहीं होती है। आईटीसी द्वारा सामना की जाने वाली मुख्य कठिनाइयां एक प्रयोग के लिए आवश्यक लक्ष्य प्रोटीन की उच्च सांद्रता और छोटे बाध्यकारी एंथैल्पी26 के कारण गैर-सहसंयोजक परिसरों का विश्लेषण करने में कठिनाई हैं। एसपीआर और बीएलआई दोनों लेबल-मुक्त बायोफिजिकल तकनीकें हैं जो सेंसर सतह पर लक्ष्य अणु के स्थिरीकरण पर भरोसा करती हैं, इसके बाद स्थिर लक्ष्य27,28 पर विश्लेषक के बाद के इंजेक्शन होते हैं। एसपीआर में, बायोमोलेक्यूलर इंटरैक्शन के दौरान अपवर्तक सूचकांक में परिवर्तनको मापा जाता है 27; बीएलआई में, परावर्तित प्रकाश में हस्तक्षेप को वास्तविक समय में समय28 के एक समारोह के रूप में तरंग दैर्ध्य में परिवर्तन के रूप में दर्ज किया जाता है। एसपीआर और बीएलआई दोनों उच्च विशिष्टता, संवेदनशीलता और पहचान क्षमताओं की पेशकश करने के सामान्य लाभसाझा करते हैं। दोनों तरीकों में, लक्ष्य प्रोटीन को बायोसेंसर सतहों पर स्थिर किया जाता है, और इसलिए, लक्ष्य की मूल संरचना का कुछ नुकसान हो सकता है, जिससे विशिष्ट बनाम गैर-विशिष्ट इंटरैक्शन30 के बीच भेदभाव करना मुश्किल हो जाता है। बीएलआई लक्ष्य को स्थिर करने के लिए महंगे डिस्पोजेबल फाइबर-ऑप्टिक बायोसेंसर का उपयोग करता है, और इसलिए, एक महंगी तकनीकहै। इन अच्छी तरह से स्थापित biomolecular पता लगाने के उपकरणों की तुलना में, FEB प्रौद्योगिकी गतिज लक्षण वर्णन के साथ वास्तविक समय में biomolecular पता लगाने के लिए कम nanomolar सांद्रता का उपयोग करके एक विश्वसनीय और लेबल मुक्त मंच प्रदान करता है। FEB प्रौद्योगिकी भी आईटीसी में सामना की जाने वाली बुदबुदाती चुनौतियों को दूर करती है और एसपीआर या बीएलआई की तुलना में अधिक लागत प्रभावी है।
फील्ड-इफेक्ट ट्रांजिस्टर (एफईटी) आधारित बायोसेंसर विभिन्न बायोमेडिकल अनुप्रयोगों की पेशकश करके बायोमोलेक्यूलर इंटरैक्शन का पता लगाने के लिए एक उभरता हुआ क्षेत्र है। एफईटी प्रणाली में, लक्ष्यों को बायोसेंसर चिप्स के लिए स्थिर किया जाता है और चालकता32 में परिवर्तन द्वारा इंटरैक्शन का पता लगाया जाता है। एक कुशल इलेक्ट्रॉनिक बायोसेंसर के विकास में विचार की जाने वाली अद्वितीय विशेषता भौतिक-रासायनिक गुण हैं जैसे कि अर्ध-प्रवाहकीय प्रकृति और सेंसर सतह33 को बनाने के लिए उपयोग की जाने वाली कोटिंग सामग्री की रासायनिक स्थिरता। एफईटी के लिए उपयोग की जाने वाली सिलिकॉन जैसी पारंपरिक सामग्रियों ने सेंसर की संवेदनशीलता को सीमित कर दिया है क्योंकि इसके लिए ट्रांजिस्टर चैनल और उचित कामकाज के लिए एक विशिष्ट वातावरण के बीच सैंडविच की गई ऑक्साइड परतों की आवश्यकता होतीहै। इसके अलावा, सिलिकॉन ट्रांजिस्टर उच्च नमक वातावरण के प्रति संवेदनशील होते हैं, इस प्रकार उनके प्राकृतिक वातावरण में जैविक बातचीत को मापना मुश्किल हो जाता है। Graphene-आधारित बायोसेंसर को एक विकल्प के रूप में प्रस्तुत किया जाता है क्योंकि यह उत्कृष्ट रासायनिक स्थिरता और विद्युत क्षेत्र प्रदान करता है। चूंकि ग्राफीन कार्बन की एक एकल परमाणु परत है, इसलिए यह अर्ध-कंडक्टर के रूप में बेहद संवेदनशील है और जैविक समाधानों के साथ रासायनिक रूप से संगत है; इन गुणों के दोनों संगत इलेक्ट्रॉनिक biosensors35 उत्पन्न करने के लिए वांछनीय हैं. graphene-लेपित biosensors द्वारा की पेशकश biomolecules की उल्लेखनीय ultrahigh लोडिंग क्षमता graphene आधारित biosensors FEB प्रौद्योगिकी के विकास के लिए नेतृत्व करते हैं।
FEB प्रौद्योगिकी का सिद्धांत: FEB एक लेबल-मुक्त बायोमोलेक्यूलर डिटेक्शन तकनीक है जो ग्राफीन बायोसेंसर के माध्यम से विद्युत प्रवाह को मापता है जिसके लिए बाध्यकारी लक्ष्य स्थिर होते हैं। immobilized प्रोटीन और विश्लेषक के बीच बातचीत वर्तमान है कि वास्तविक समय में निगरानी कर रहे हैं, सटीक गतिज मापसक्षम 36 में परिवर्तन में परिणाम.
इंस्ट्रूमेंटेशन: फेब सिस्टम में एक ग्राफीन फील्ड-इफेक्ट ट्रांजिस्टर (जीएफईटी) सेंसर चिप और एक इलेक्ट्रॉनिक रीडर शामिल है जो पूरे प्रयोग में एक निरंतर वोल्टेज लागू करता है (चित्रा 1)। विश्लेषक को बायोसेंसर सतह पर स्थिर किए गए लक्ष्य प्रोटीन के समाधान में लागू किया जाता है। जब कोई इंटरैक्शन होता है, तो वर्तमान में एक परिवर्तन मापा जाता है और वास्तविक समय में दर्ज किया जाता है। जैसे-जैसे विश्लेषक एकाग्रता बढ़ती है, बाध्य विश्लेषक का अंश भी बढ़ेगा, जिससे वर्तमान में उच्च परिवर्तन होंगे। उपकरण (सामग्री की तालिका) के साथ प्रदान किए गए स्वचालित विश्लेषण सॉफ़्टवेयर का उपयोग करके, आई-रिस्पांस को मापा जाता है और बायोसेंसिंग इकाइयों (बीयू) 37 के संदर्भ में दर्ज किया जाता है। I-Response को विश्लेषण के साथ immobilized लक्ष्य की बातचीत पर वास्तविक समय में मापा गया बायोसेंसर चिप के माध्यम से वर्तमान (I) में परिवर्तन के रूप में परिभाषित किया गया है। FEB स्वचालित विश्लेषण सॉफ़्टवेयर गतिशील इंटरैक्शन घटनाओं के लिए I-Response और C-Response दोनों का विश्लेषण कर सकता है, जहां C-Response धारिता (C) में परिवर्तन रिकॉर्ड करता है। आई-रिस्पांस और सी-रिस्पांस दोनों में भिन्नताएं सीधे बाध्य विश्लेषक के अंश के अनुरूप हैं और केडी मान उत्पन्न करने के लिए आगे का विश्लेषण किया जा सकता है। स्वचालित विश्लेषण सॉफ़्टवेयर की डिफ़ॉल्ट वरीयता I-Response है.
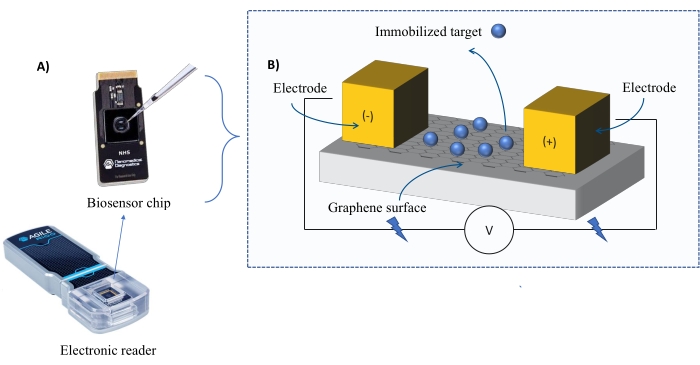
चित्रा 1: प्रयोगात्मक सेटअप का अवलोकन. (ए) Graphene-आधारित चिप और एक इलेक्ट्रॉनिक रीडर. (बी) चिप घटकों का एक सिंहावलोकन। चिप दो इलेक्ट्रोड से जुड़ी होती है जो सिस्टम को वर्तमान की आपूर्ति करती है। चिप की सतह को ग्राफीन के साथ कवर किया गया है, जो सक्रिय होने पर लक्ष्य को बांध सकता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
पद्धति:
प्रारंभ में, सक्रिय बायोसेंसर चिप को FEB डिवाइस (चित्रा 1) में डाला जाता है, जिसके बाद नीचे उल्लिखित चरणों का निष्पादन होता है: (1) अंशांकन: प्रयोग बेसलाइन संतुलन प्रतिक्रिया बनाने के लिए 1x फॉस्फेट-बफ़र्ड खारा (PBS; pH = 7.4) का उपयोग करके सिस्टम अंशांकन के साथ शुरू होता है। (2) एसोसिएशन: चिप में विश्लेषक पेश किया जाता है, और बाध्यकारी संतृप्ति तक पहुंचने तक आई-रिस्पांस की निगरानी की जाती है। (3) पृथक्करण: विश्लेषक को 1x PBS का उपयोग करके अलग किया जाता है। (4) पुनर्जनन: 1x PBS का उपयोग करके विश्लेषक के अवशेष ों को हटा दिया जाता है। (5) धुलाई: चिप से बाध्य और अनबाउंड एनालिस्ट को पूरी तरह से हटाने के लिए 1x पीबीएस का उपयोग करके कुल पांच धोने का प्रदर्शन किया जाता है।
विश्लेषण:
डेटा विश्लेषण उपकरण के साथ प्रदान किए गए पूरी तरह से स्वचालित सॉफ़्टवेयर का उपयोग करके किया जाता है। स्वचालित विश्लेषण सॉफ़्टवेयर एक KD मान के साथ एक हिल फिट प्लॉट उत्पन्न करता है। हिल फिट प्लॉट विश्लेषक सांद्रता के एक समारोह के रूप में लक्ष्य प्रोटीन के लिए एक विश्लेषक के संबंध का वर्णन करता है। वह सांद्रता जिस पर एक अर्ध-अधिकतम प्रतिक्रिया प्राप्त की जाती है, KD मान के लिए आनुपातिक होती है। एक कम KD मान उच्च बाध्यकारी आत्मीयता और इसके विपरीत का प्रतिनिधित्व करता है।
FEB प्रयोग से प्राप्त डेटा को मान्य करने के लिए, I-Responses डेटा समीक्षा / निर्यात सॉफ़्टवेयर का उपयोग करके प्रत्येक विश्लेषक एकाग्रता के लिए प्रत्येक रीडआउट बिंदु से निकाले जाते हैं और अन्य सांख्यिकीय विश्लेषण सॉफ़्टवेयर ( सामग्री की तालिका देखें) को निर्यात किया जा सकता है जैसा कि नीचे बताया गया है।
Protocol
Representative Results
Discussion
इस अध्ययन में, एचएसपी 90 और सीडीसी 37 के बीच बायोमोलेक्यूलर इंटरैक्शन को निर्धारित करने के लिए फेब तकनीक (एक वास्तविक समय गतिज लक्षण वर्णन दृष्टिकोण) का उपयोग करने की व्यवहार्यता का मूल्यांकन किया गया था?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
इस शोध को द्विराष्ट्रीय विज्ञान फाउंडेशन (BSF) से S.K.S. और N.Q. को अनुदान द्वारा समर्थित किया गया था।
Materials
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA |
Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA |
T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 |
White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA |
|
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 |
White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA |
Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 |
Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA |
Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA |
Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA |
Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA |
|
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA |
|
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA |
|
| Statistical software | GraphPad, Prism | NA CAS number: NA |
Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 |
White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA |
Tip 1-200 µl, in bulk, 1,000 pcs |
References
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -. A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083 (2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -. H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. 암 연구학. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438 (2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213 (2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882 (2014).
- Vu, C. -. A., Chen, W. -. Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214 (2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434 (2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era…? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).

