Caenorhabditis elegans 건강에 영향을 미치는 미생물 분리물의 고처리량 스크리닝
Summary
장내 미생물은 특정 또는 보존 메커니즘을 통해 숙주의 건강에 긍정적 또는 부정적 영향을 미칠 수 있습니다. Caenorhabditis elegans 는 그러한 미생물을 스크리닝하는 편리한 플랫폼입니다. 본 프로토콜은 웜 건강의 대용으로 사용되는 선충 스트레스 저항성에 미치는 영향에 대한 48개의 박테리아 분리주의 고처리량 스크리닝을 설명합니다.
Abstract
작은 크기, 짧은 수명 및 쉬운 유전학을 갖춘 Caenorhabditis elegans 는 미생물 분리가 숙주 생리학에 미치는 영향을 연구할 수 있는 편리한 플랫폼을 제공합니다. 또한 죽을 때 파란색으로 형광을 발하여 죽음을 정확히 찾아내는 편리한 수단을 제공합니다. 이 특성은 고처리량 무표지 C. 엘레간스 생존 분석법(LFASS)을 개발하는 데 활용되었습니다. 여기에는 멀티웰 플레이트에 설정된 웜 개체군의 시간 경과 형광 기록이 포함되며, 이로부터 개체군 사망 중앙값을 도출할 수 있습니다. 본 연구는 심한 열 및 산화 스트레스에 대한 C. elegans 감수성에 대한 영향에 대해 한 번에 여러 미생물 분리 물을 스크리닝하기 위해 LFASS 접근법을 채택합니다. 이러한 미생물 스크리닝 파이프라인은 특히 숙주 건강에 대한 대용으로 심각한 스트레스 저항성을 사용하여 프로바이오틱스를 사전 스크리닝하는 데 사용될 수 있습니다. 이 프로토콜은 분석을 위해 결합하기 전에 멀티웰 어레이에서 C. elegans 장내 미생물 분리 컬렉션과 동기 웜 개체군을 모두 성장시키는 방법을 설명합니다. 제공된 예에서는 2개의 웜 균주에 대한 47개의 박테리아 분리주와 1개의 대조 균주를 두 개의 스트레스 분석에서 병렬로 테스트하는 방법을 다룹니다. 그러나 접근 파이프라인은 쉽게 확장할 수 있으며 다른 많은 양식의 스크리닝에 적용할 수 있습니다. 따라서 C. elegans 건강에 영향을 미치는 생물학적 및 생화학적 조건의 다중 매개변수 환경을 신속하게 조사할 수 있는 다목적 설정을 제공합니다.
Introduction
인체에는 주로 장, 피부 및점막 환경에서 발견되는 약 10-100조 개의 살아있는 미생물 세포(박테리아, 고세균 균류)가 있습니다1. 건강한 상태에서 이들은 비타민 생산, 면역 체계의 성숙, 병원체에 대한 선천성 및 적응성 면역 반응의 자극, 지방 대사 조절, 스트레스 반응의 조절 등을 포함하여 숙주에게 이점을 제공하며 성장 및 발달, 질병 발병 및 노화에 영향을 미칩니다 2,3,4,5 . 장내 미생물은 또한 평생 동안 상당히 진화합니다. 가장 급격한 진화는 유아기와 유아기에 발생하지만6, 비피도박테리움 풍부도의 감소와 클로스트리디움, 락토바실러스, 장내세균 및 엔테로코커스 종7의 증가를 포함하여 연령에 따라 상당한 변화도 발생합니다. 생활 습관은 dysbiosis(유익한 박테리아의 손실, 기회 박테리아의 과증식)로 이어지는 장내 미생물 구성을 더욱 변화시켜 염증성 장 질환, 당뇨병 및 비만5과 같은 다양한 병리를 유발할 수 있지만 알츠하이머 및 파킨슨병 8,9,10,11에도 기여할 수 있습니다.
이러한 실현은 장 생리학(현재 그 안에 있는 미생물 포함)과 신경계 간의 상호 작용이 동물의 대사 및 생리 기능의 주요 조절자로 간주되는 장-뇌 축(GBA)의 개념을 개선하는 데 결정적으로 기여했습니다12. 그러나 장-뇌 신호 전달에서 미생물총의 정확한 역할과 관련 작용 메커니즘은 완전히 이해되지 않았습니다13. 장내 미생물총이 건강한 노화의 핵심 결정 요인이기 때문에 박테리아가 노화 과정을 조절하는 방법은 강렬한 연구와 논쟁의 대상이 되었습니다 6,14,15.
회충 Caenorhabditis elegans가 박테로이데테스, 피르미쿠테스 및 방선균 16,17,18,19,20에 의해 다른 종에서와 마찬가지로 지배하는 보나피드 장내 미생물총을 숙주한다는 입증과 함께, 숙주-장 공생 상호 작용을 연구하기 위한 실험 플랫폼으로서의 급속한 부상21,22,23,24 ,25,26은 우리의 수사 무기고26,27,28,29를 크게 확장했습니다. 특히, C. elegans가 유전자-식단, 유전자-약물, 유전자-병원체 등의 상호 작용을 연구하는 데 사용할 수 있는 고처리량 실험 접근 방식을 적용하여 박테리아 분리 및 칵테일이 C. elegans의 건강과 노화에 미치는 영향을 빠르게 탐색할 수 있습니다.
본 프로토콜은 프로바이오틱스를 식별하는 데 사용할 수 있는 건강의 대용물로서 C. elegans 스트레스 저항성에 미치는 영향을 위해 멀티웰 플레이트에 설정된 박테리아 분리 또는 혼합물의 어레이를 한 번에 스크리닝하는 실험 파이프라인을 설명합니다. 형광 플레이트 판독기를 사용하여 자동화된 응력 저항 분석을 위해 웜을 처리하기 전에 큰 벌레 개체군을 늘리고 96웰 및 384웰 플레이트 형식으로 박테리아 어레이를 처리하는 방법을 자세히 설명합니다(그림 1). 이 접근법은 사멸 형광 31 현상을 이용하는 무표지 자동 생존 분석법(LFASS)30을 기반으로 하며, 이를 통해 죽어가는 벌레는 사망 시간을 정확히 찾아내는 데 사용할 수 있는 청색 형광을 생성합니다. 청색 형광은 C. elegans 장 과립(리소좀 관련 소기관의 일종)에 저장된 안트라닐산의 글루코실 에스테르에 의해 방출되며, 사망 시 벌레 장에서 괴사 캐스케이드가 촉발될 때 파열됩니다.31.
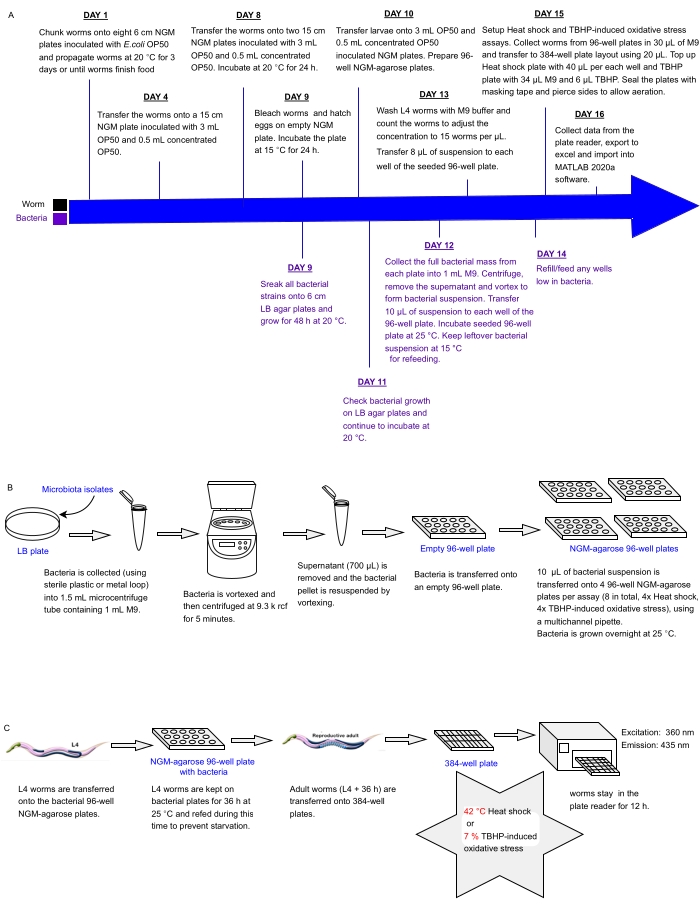
그림 1: 스트레스에 대한 C. elegans 저항성에 영향을 미치는 박테리아 분리주의 고처리량 스크리닝을 위한 실험 워크플로 . (A) 웜 및 박테리아 유지 관리 및 분석 설정 일정. (B) 96웰 박테리아 플레이트 어레이 설정 및 취급. (C) 384웰 웜 플레이트 설정. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
C. elegans는 작은 크기, 투명성, 빠른 개발, 짧은 수명, 저렴한 가격 및 취급 용이성으로 인해 한 번에 여러 실험 매개 변수를 신속하게 스크리닝하는 데 많은 이점을 제공합니다. 게놈, 신체 계획, 신경계, 장 및 미생물군집이 상당히 단순하지만 인간과 충분히 복잡하고 유사하기 때문에 생체 활성 효능 또는 독성을 테스트하는 동안 기계론적 통찰력을 얻을 수 있는 강력한 전임상 모델입니다….
Disclosures
The authors have nothing to disclose.
Acknowledgements
웜 균주를 제공한 CGC 미네소타(미국 매디슨, NIH – P40 OD010440)와 여기에 묘사된 모든 환경 미생물 분리물을 제공한 OP50 및 Pr. Hinrich Schulenburg(CAU, Kiel, 독일)에 감사드립니다. 이 작업은 AB에 대한 UKRI-BBSRC 보조금(BB/S017127/1)으로 자금을 지원받았습니다. JM은 랭커스터 대학교 FHM PhD 장학금으로 자금을 지원합니다.
Materials
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
References
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood – Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer’s disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson’s disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio – The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer’s disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson’s disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).

