Circuitos digitales genéticos basados en sistemas CRISPR-Cas y proteínas anti-CRISPR
Summary
Los sistemas CRISPR-Cas y las proteínas anti-CRISPR se integraron en el esquema de puertas booleanas en Saccharomyces cerevisiae. Los nuevos circuitos lógicos pequeños mostraron un buen rendimiento y profundizaron la comprensión de los factores de transcripción basados en dCas9 / dCas12a y las propiedades de las proteínas anti-CRISPR.
Abstract
Las puertas booleanas de genes sintéticos y los circuitos digitales tienen una amplia gama de aplicaciones, desde el diagnóstico médico hasta el cuidado del medio ambiente. El descubrimiento de los sistemas CRISPR-Cas y sus inhibidores naturales, las proteínas anti-CRISPR (Acrs), proporciona una nueva herramienta para diseñar e implementar circuitos digitales de genes in vivo . Aquí, describimos un protocolo que sigue la idea del ciclo de ingeniería biológica “Diseñar-Construir-Probar-Aprender” y hace uso de dCas9/dCas12a junto con sus correspondientes Acrs para establecer pequeñas redes transcripcionales, algunas de las cuales se comportan como puertas booleanas, en Saccharomyces cerevisiae. Estos resultados señalan las propiedades de dCas9/dCas12a como factores de transcripción. En particular, para lograr la activación máxima de la expresión génica, dSpCas9 necesita interactuar con un ARN de andamio diseñado que recolecta múltiples copias del dominio de activación VP64 (AD). Por el contrario, dCas12a se fusionará, en el extremo C, con el fuerte VP64-p65-Rta (VPR) AD. Además, la actividad de ambas proteínas Cas no se ve reforzada por el aumento de la cantidad de sgRNA/crRNA en la célula. Este artículo también explica cómo construir puertas booleanas basadas en la interacción CRISPR-dCas-Acr. El dominio de unión a hormonas fusionado AcrIIA4 del receptor de estrógeno humano es el núcleo de una puerta NOT que responde al β-estradiol, mientras que los AcrVA sintetizados por el promotor GAL1 inducible permiten imitar las puertas SÍ y NO con galactosa como entrada. En estos últimos circuitos, AcrVA5, junto con dLbCas12a, mostró el mejor comportamiento lógico.
Introduction
En 2011, los investigadores propusieron un método computacional y desarrollaron una pieza correspondiente de software para el diseño automático de circuitos genéticos sintéticos digitales1. Un usuario tenía que especificar el número de entradas (tres o cuatro) y rellenar la tabla de verdad del circuito; Esto proporcionó toda la información necesaria para derivar la estructura del circuito utilizando técnicas de la electrónica. La tabla de verdad se tradujo a dos fórmulas booleanas a través del método 2 del mapa de Karnaugh. Cada fórmula booleana está hecha de cláusulas que describen operaciones lógicas (suma o multiplicación) entre (parte de) las entradas del circuito y sus negaciones (los literales). Las cláusulas, a su vez, se suman (OR) o se multiplican (AND) para calcular la salida del circuito. Cada circuito se puede realizar de acuerdo con cualquiera de sus dos fórmulas correspondientes: una escrita en forma POS (producto de sumas) y la otra en representación SOP (suma de productos). El primero consiste en una multiplicación de cláusulas (es decir, puertas booleanas) que contienen una suma lógica de los literales. Este último, en cambio, es una suma de cláusulas donde los literales se multiplican.
Los circuitos eléctricos se pueden realizar, en una placa de pruebas, conectando físicamente diferentes puertas juntas. La corriente eléctrica permite el intercambio de señales entre puertas, lo que conduce al cálculo de la salida.
En biología, la situación es más compleja. Una puerta booleana se puede realizar como una unidad de transcripción (TU; es decir, la secuencia “promotor-codificante de la región-terminador” dentro de las células eucariotas), donde la transcripción o la traducción (o ambas) están reguladas. Así, al menos dos tipos de moléculas establecen un cableado biológico: las proteínas del factor de transcripción y los ARN antisentido no codificantes1.
Un circuito digital de genes está organizado en dos o tres capas de puertas, a saber: 1) la capa de entrada, que está hecha de puertas SÍ (tampón) y NO y convierte los productos químicos de entrada en moléculas de cableado; 2) la capa interna, que consiste en tantas TU como cláusulas hay en la fórmula booleana correspondiente. Si el circuito está diseñado de acuerdo con la fórmula SOP, cada cláusula en la capa interna producirá la salida del circuito (por ejemplo, fluorescencia) en una llamada arquitectura de salida distribuida. Si se utiliza la fórmula del producto de suma (POS), entonces se requiere una capa final 3), que contendrá una sola puerta multiplicativa que recoge las moléculas de cableado de la capa interna.
En general, en biología sintética, se pueden diseñar muchos esquemas diferentes para el mismo circuito. Difieren en el número y el tipo de TU y moléculas de cableado. Para elegir la solución más fácil de implementar en celdas de levadura, cada diseño de circuito se asocia con una puntuación de complejidad S, definida como
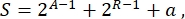
donde A representa el número de activadores, R representa el número de represores y a es la cantidad de moléculas de ARN antisentido. Si los activadores o represores están ausentes del circuito, su contribución a S es cero. Por lo tanto, es más difícil realizar un esquema de circuito en el laboratorio (S alta) cuando requiere un alto número de factores de transcripción ortogonales. Esto significa que los nuevos activadores y represores deben diseñarse de novo para realizar el cableado completo dentro de los circuitos digitales. En principio, las nuevas proteínas de unión al ADN se pueden ensamblar utilizando las proteínas Zinc Finger3 y los efectores TAL4 como plantillas. Sin embargo, esta opción parece demasiado ardua y lenta; por lo tanto, uno debe confiar principalmente en pequeños ARN y regulación de traducción para finalizar circuitos genéticos complejos.
Originalmente, este método fue desarrollado para fabricar circuitos digitales en bacterias. De hecho, en las células eucariotas, en lugar de ARN antisentido, es más adecuado hablar de microARN (miARN) o pequeños ARN interferentes (siRNAs)5. Sin embargo, la vía del ARNi no está presente en la levadura S. cerevisiae. Por lo tanto, uno debe optar por redes completamente transcripcionales. Supongamos que un circuito necesita cinco activadores y cinco represores; su puntuación de complejidad sería S = 32. La complejidad del circuito puede reducirse reemplazando los 10 factores de transcripción con un solo dCas96 (Cas9 deficiente en nucleasa) fusionado a un dominio de activación (AD). Como se muestra en7, dCas9-AD funciona como un represor en la levadura cuando se une a un promotor entre la caja TATA y el TSS (sitio de inicio de la transcripción) y como un activador cuando se une bien aguas arriba de la caja TATA. Por lo tanto, se pueden reemplazar 10 factores de transcripción con una sola proteína de fusión dCas9-AD y 10 sgRNAs (ARN guía únicos) para una puntuación de complejidad total de S = 11. Es rápido y fácil sintetizar diez sgRNAs, mientras que, como se comentó anteriormente, el ensamblaje de 10 proteínas exigiría un trabajo mucho más largo y complicado.
Alternativamente, se podrían usar dos proteínas dCas ortogonales (por ejemplo, dCas9 y dCas12a): una para fusionarse con un AD y la otra desnuda o en combinación con un dominio de represión. La puntuación de complejidad aumentaría en una sola unidad (S = 12). Por lo tanto, los sistemas CRISPR-dCas son la clave para la construcción de circuitos digitales de genes muy intrincados en S. cerevisiae.
Este documento caracteriza profundamente la eficiencia de los represores y activadores basados en dCas9 y dCas12a en levadura. Los resultados muestran que no demandan una gran cantidad de sgRNA para optimizar su actividad, por lo que los plásmidos episomales se evitan preferentemente. Además, los activadores basados en dCas9 son mucho más efectivos cuando se usa un ARN de andamio (scRNA) que recluta copias del VP64 AD. Por el contrario, dCas12a funciona bien cuando se fusiona directamente con el fuerte VPR AD. Además, un promotor activado sintético exige un número variable de sitios diana, dependiendo de la configuración del activador (por ejemplo, tres cuando se utiliza dCas12a-VPR, seis para dCas9-VP64 y solo uno con dCas9 y un scRNA). Como represor, dCas12a parece más incisivo cuando se une a la región codificante que al promotor.
Sin embargo, como inconveniente, CRISPR-dCas9 / dCas12a no interactúan directamente con los productos químicos. Por lo tanto, es posible que no sean útiles en la capa de entrada. Por esta razón, se han investigado diseños alternativos de puertas booleanas que contienen proteínas anti-CRISPR (Acrs). Acrs actúan sobre las proteínas (d)Cas e inhiben su funcionamiento8. Por lo tanto, son un medio para modular la actividad de los sistemas CRISPR-(d)Cas. Este artículo analiza a fondo las interacciones entre Acrs tipo II y (d)Cas9, así como Acrs tipo V y (d)Cas12a en S. cerevisiae. Dado que los Acr son mucho más pequeños que las proteínas Cas, se construyó una puerta NOT que responde al estrógeno β-estradiol fusionando el dominio de unión hormonal del receptor de estrógeno humano9-HBD (hER) con AcrIIA4. Además, se realizaron un puñado de puertas SÍ y NO que expresaban dCas12a (-AD) constitutivamente y AcrVA en la inducción con galactosa. En la actualidad, estas puertas sirven solo como prueba de concepto. Sin embargo, también representan el primer paso hacia un replanteamiento profundo del algoritmo para llevar a cabo el diseño automático computacional de circuitos digitales de genes sintéticos en células de levadura.
Protocol
Representative Results
Discussion
El protocolo mostró un posible flujo de trabajo completo para los circuitos digitales de genes sintéticos, siguiendo el ciclo de ingeniería biológica “Diseñar-Construir-Probar-Aprender” (DBTL) y con respecto a los experimentos de laboratorio seco y laboratorio húmedo. Aquí, nos centramos en el sistema CRISPR-Cas, principalmente dSpCas9, denAsCas12a, dLbCas12a y las proteínas anti-CRISPR correspondientes, diseñando y construyendo en S. cerevisiae pequeñas redes transcripcionales. Algunos de ellos imitab…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Queremos agradecer a todos los estudiantes del laboratorio de Biología Sintética -SPST, TJU- por su ayuda general, junto con Zhi Li y Xiangyang Zhang por su asistencia en los experimentos de FACS.
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
References
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

