CRISPR-Cas Sistemlerine ve Anti-CRISPR Proteinlerine Dayalı Gen Dijital Devreleri
Summary
CRISPR-Cas sistemleri ve anti-CRISPR proteinleri, Saccharomyces cerevisiae’deki Boole kapılarının şemasına entegre edildi. Yeni küçük mantık devreleri iyi performans gösterdi ve hem dCas9 / dCas12a tabanlı transkripsiyon faktörlerinin hem de anti-CRISPR proteinlerinin özelliklerinin anlaşılmasını derinleştirdi.
Abstract
Sentetik gen Boole kapıları ve dijital devreler, tıbbi teşhisten çevresel bakıma kadar geniş bir uygulama alanına sahiptir. CRISPR-Cas sistemlerinin ve doğal inhibitörlerinin (anti-CRISPR proteinleri (Acrs)) keşfi, in vivo gen dijital devrelerini tasarlamak ve uygulamak için yeni bir araç sağlar. Burada, “Tasarla-İnşa Et-Test Et-Öğren” biyolojik mühendislik döngüsü fikrini takip eden ve Saccharomyces cerevisiae’de bazıları Boole kapıları gibi davranan küçük transkripsiyonel ağlar oluşturmak için dCas9 / dCas12a’yı karşılık gelen Acrs ile birlikte kullanan bir protokolü açıklıyoruz. Bu sonuçlar, transkripsiyon faktörleri olarak dCas9 / dCas12a’nın özelliklerine işaret etmektedir. Özellikle, gen ekspresyonunun maksimum aktivasyonunu elde etmek için, dSpCas9’un VP64 aktivasyon alanının (AD) çoklu kopyalarını toplayan tasarlanmış bir iskele RNA’sı ile etkileşime girmesi gerekir. Buna karşılık, dCas12a, C terminusta, güçlü VP64-p65-Rta (VPR) AD ile kaynaştırılmalıdır. Ayrıca, her iki Cas proteininin aktivitesi, hücredeki sgRNA / crRNA miktarını artırarak arttırılmaz. Bu makalede ayrıca CRISPR-dCas-Acr etkileşimine dayalı Boole kapılarının nasıl oluşturulacağı açıklanmaktadır. İnsan östrojen reseptörünün AcrIIA4 kaynaşmış hormon bağlama alanı, β-östradiol’e yanıt veren bir NOT kapısının çekirdeğidir, oysa indüklenebilir GAL1 promotörü tarafından sentezlenen AcrVA’lar, hem EVET hem de NOT kapılarını bir girdi olarak galaktoz ile taklit etmeye izin verir. İkinci devrelerde, AcrVA5, dLbCas12a ile birlikte en iyi mantık davranışını gösterdi.
Introduction
2011 yılında, araştırmacılar hesaplamalı bir yöntem önerdiler ve dijital sentetik gen devrelerinin otomatik tasarımı için karşılık gelen bir yazılım parçası geliştirdiler1. Bir kullanıcının giriş sayısını (üç veya dört) belirtmesi ve devre doğruluk tablosunu doldurması gerekiyordu; Bu, elektronikten teknikler kullanarak devre yapısını türetmek için gerekli tüm bilgileri sağladı. Doğruluk tablosu, Karnaugh harita yöntemi2 aracılığıyla iki Boole formülüne çevrildi. Her Boole formülü, devre girişleri ve bunların olumsuzlamaları (değişmez değerler) arasındaki (bir kısmı) mantık işlemlerini (toplama veya çarpma) tanımlayan yan tümcelerden oluşur. Yan tümceler, sırasıyla, devre çıkışını hesaplamak için ya toplanır (OR) ya da çarpılır (AND). Her devre, karşılık gelen iki formülden herhangi birine göre gerçekleştirilebilir: biri POS (toplamların ürünü) formunda, diğeri SÇP (ürünlerin toplamı) temsilinde yazılmıştır. İlki, değişmez değerlerin mantıksal bir toplamını içeren cümleciklerin (yani, Boole kapıları) çarpımından oluşur. İkincisi, aksine, değişmez değerlerin çarpıldığı bir cümle toplamıdır.
Elektrik devreleri, bir breadboard üzerinde, farklı kapıları fiziksel olarak birbirine bağlayarak gerçekleştirilebilir. Elektrik akımı, kapılar arasında sinyal alışverişine izin verir ve bu da çıkışın hesaplanmasına yol açar.
Biyolojide durum daha karmaşıktır. Bir Boole kapısı, transkripsiyon veya translasyonun (veya her ikisinin) düzenlendiği bir transkripsiyon birimi (TU; yani, ökaryotik hücreler içindeki “promotör-kodlayıcı bölge-sonlandırıcı” dizisi) olarak gerçekleştirilebilir. Böylece, en az iki tür molekül biyolojik bir kablolama kurar: transkripsiyon faktörü proteinleri ve kodlamayan, antisens RNA’lar1.
Bir gen dijital devresi, iki veya üç kapı katmanı halinde düzenlenir, yani: 1) EVET (tampon) ve NOT kapılarından oluşan ve giriş kimyasallarını kablolama moleküllerine dönüştüren giriş katmanı; 2) karşılık gelen Boole formülünde yan tümceler olduğu kadar TU’dan oluşan iç katman. Devre SÇP formülüne göre tasarlanmışsa, iç katmandaki her cümle, dağıtılmış çıkış mimarisi olarak adlandırılan bir devrede devre çıkışını (örneğin, floresan) üretecektir. Toplam (POS) formülünün ürünü kullanılırsa, kablolama moleküllerini iç katmandan toplayan tek bir çarpma kapısı içerecek olan 3) son katman gereklidir.
Genel olarak, sentetik biyolojide, aynı devre için birçok farklı şema tasarlanabilir. Hem TU’ların hem de kablolama moleküllerinin sayısı ve türü bakımından farklılık gösterirler. Maya hücrelerinde uygulanacak en kolay çözümü seçmek için, her devre tasarımı, şu şekilde tanımlanan bir karmaşıklık skoru S ile ilişkilendirilir:
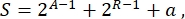
burada A, aktivatörlerin sayısını, R, baskılayıcıların sayısını ve A, antisens RNA moleküllerinin miktarını temsil eder. Aktivatörler veya baskılayıcılar devrede yoksa, S’ye katkıları sıfırdır. Bu nedenle, çok sayıda ortogonal transkripsiyon faktörü gerektirdiğinde laboratuarda bir devre şeması (yüksek S) gerçekleştirmek daha zordur. Bu, dijital devrelerin içindeki tüm kablolamayı gerçekleştirmek için yeni aktivatörlerin ve baskılayıcıların de novo olarak tasarlanacağı anlamına gelir. Prensip olarak, yeni DNA bağlayıcı proteinler, şablon olarak Çinko Parmak proteinleri3 ve TAL efektörleri4 kullanılarak birleştirilebilir. Ancak, bu seçenek çok zorlu ve zaman alıcı görünüyor; bu nedenle, karmaşık gen devrelerini sonlandırmak için çoğunlukla küçük RNA’lara ve çeviri düzenlemesine güvenilmelidir.
Başlangıçta, bu yöntem bakterilerde dijital devreler üretmek için geliştirilmiştir. Gerçekten de, ökaryotik hücrelerde, antisens RNA’lar yerine, mikroRNA’lardan (miRNA’lar) veya küçük müdahale eden RNA’lardan (siRNA’lar)5 bahsetmek daha uygundur. Bununla birlikte, RNAi yolu maya S. cerevisiae’de mevcut değildir. Bu nedenle, kişi tamamen transkripsiyonel ağları tercih etmelidir. Bir devrenin beş aktivatöre ve beş baskıcıya ihtiyacı olduğunu varsayalım; karmaşıklık puanı S = 32 olacaktır. Devre karmaşıklığı, 10 transkripsiyon faktörünün bir aktivasyon alanına (AD) kaynaşmış tek bir dCas96 (nükleaz eksikliği Cas9) ile değiştirilmesiyle azaltılabilir. 7’de gösterildiği gibi, dCas9-AD, TATA kutusu ile TSS (transkripsiyon başlangıç bölgesi) arasında bir promotör bağlarken mayada bir baskılayıcı olarak ve TATA kutusunun yukarı akışını iyi bağlarken bir aktivatör olarak çalışır. Böylece, 10 transkripsiyon faktörünü, tek bir dCas9-AD füzyon proteini ve 10 sgRNA (tek kılavuz RNA’lar) ile S = 11’lik toplam karmaşıklık skoru için değiştirebiliriz. On sgRNA’yı sentezlemek hızlı ve kolaydır, oysa daha önce yorumlandığı gibi, 10 proteinin montajı çok daha uzun ve daha karmaşık bir çalışma gerektirecektir.
Alternatif olarak, biri iki ortogonal dCas proteini (örneğin, dCas9 ve dCas12a) kullanabilir: biri bir AD ile kaynaşmak için, diğeri çıplak veya bir baskı alanı ile kombinasyon halinde. Karmaşıklık puanı yalnızca bir birim artacaktır (S = 12). Bu nedenle, CRISPR-dCas sistemleri, S. cerevisiae’de çok karmaşık gen dijital devrelerinin inşasının anahtarıdır.
Bu makale, mayadaki hem dCas9- hem de dCas12a bazlı baskılayıcıların ve aktivatörlerin verimliliğini derinden karakterize etmektedir. Sonuçlar, aktivitelerini optimize etmek için yüksek miktarda sgRNA talep etmediklerini göstermektedir, bu nedenle epizomal plazmidlerden tercihen kaçınılmaktadır. Dahası, dCas9 tabanlı aktivatörler, VP64 AD’nin kopyalarını toplayan bir iskele RNA’sı (scRNA) kullanıldığında çok daha etkilidir. Buna karşılık, dCas12a doğrudan güçlü VPR AD ile kaynaştırıldığında iyi çalışır. Ayrıca, sentetik olarak aktive edilmiş bir promotör, aktivatörün konfigürasyonuna bağlı olarak değişken sayıda hedef bölge gerektirir (örneğin, dCas12a-VPR kullanırken üç, dCas9-VP64 için altı ve dCas9 ve scRNA ile sadece bir tane). Bir baskılayıcı olarak, dCas12a, promotörden ziyade kodlama bölgesini bağlarken daha keskin görünür.
Bununla birlikte, bir dezavantaj olarak, CRISPR-dCas9 / dCas12a kimyasallarla doğrudan etkileşime girmez. Bu nedenle, giriş katmanında hiçbir işe yaramayabilirler. Bu nedenle, anti-CRISPR proteinleri (Acrs) içeren alternatif Boole kapısı tasarımları araştırılmıştır. Akr’ler (d)Cas proteinleri üzerinde etkili olur ve çalışmalarını engeller8. Bu nedenle, CRISPR-(d)Cas sistemlerinin aktivitesini modüle etmenin bir yoludur. Bu yazıda S. cerevisiae’de tip II Acrs ve (d)Cas9 ile tip V Acrs ve (d)Cas12a arasındaki etkileşimler kapsamlı bir şekilde analiz edilmiştir. Acrs, Cas proteinlerinden çok daha küçük olduğundan, östrojen β-östradiol’e yanıt veren bir NOT kapısı, insan östrojen reseptörü 9-HBD’nin (hER) hormon bağlama alanını AcrIIA4’e kaynaştırarak inşa edilmiştir. Ayrıca, dCas12a(-AD)’yi yapısal olarak ifade eden bir avuç EVET ve DEĞİL kapısı ve galaktoz ile indüksiyon üzerine AcrVA’lar gerçekleştirildi. Şu anda, bu kapılar sadece bir kavram kanıtı olarak hizmet vermektedir. Bununla birlikte, aynı zamanda, maya hücrelerindeki sentetik gen dijital devrelerinin hesaplamalı otomatik tasarımını gerçekleştirmek için algoritmanın derinlemesine yeniden düşünülmesine yönelik ilk adımı temsil ederler.
Protocol
Representative Results
Discussion
Protokol, “Tasarla-Yap-Test Et-Öğren” (DBTL) biyolojik mühendislik döngüsünü takiben ve hem kuru laboratuvar hem de ıslak laboratuvar deneyleriyle ilgili sentetik gen dijital devreleri için olası bir tam iş akışı gösterdi. Burada, CRISPR-Cas sistemine, özellikle dSpCas9, denAsCas12a, dLbCas12a ve buna karşılık gelen anti-CRISPR proteinlerine, S. cerevisiae küçük transkripsiyonel ağlarını tasarlayarak ve inşa ederek odaklandık. Bazıları dijital devrelerin temel bileşenleri olan Boole…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Sentetik Biyoloji laboratuvarı SPST, TJU’nun tüm öğrencilerine genel yardımları için, Zhi Li ve Xiangyang Zhang ile birlikte FACS deneylerindeki yardımları için teşekkür etmek istiyoruz.
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
References
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

