Polymeraskedjereaktion och dot-blot-hybridisering för Leptospira-detektion i vattenprover
Summary
I denna studie utformades en dot-blot-applikation för att detektera Leptospira från de tre huvudkladerna i vattenprover. Denna metod gör det möjligt att identifiera minimala DNA-mängder som specifikt angrips av en digoxigeninmärkt sond, som lätt detekteras av en anti-digoxigenin-antikropp. Detta tillvägagångssätt är ett värdefullt och tillfredsställande verktyg för screeningändamål.
Abstract
Dot-blot är en enkel, snabb, känslig och mångsidig teknik som gör det möjligt att identifiera minimala mängder DNA som specifikt är måltavla för sondhybridisering i närvaro av bärar-DNA. Den är baserad på överföring av en känd mängd DNA till ett inert fast stöd, såsom ett nylonmembran, med hjälp av dot-blot-apparaten och utan elektroforetisk separation. Nylonmembran har fördelen av hög nukleinsyrabindningskapacitet (400 μg/cm2), hög hållfasthet och är positivt eller neutralt laddade. Sonden som används är ett mycket specifikt ssDNA-fragment av 18 till 20 baser som är märkta med digoxigenin (DIG). Sonden kommer att konjugera med Leptospiras DNA. När sonden har hybridiserats med mål-DNA:t detekteras den av en anti-digoxigeninantikropp, vilket gör det lätt att upptäcka den genom dess utsläpp som avslöjas i en röntgenfilm. Prickarna med en emission kommer att motsvara de DNA-fragment som är av intresse. Denna metod använder icke-isotopisk märkning av sonden, som kan ha en mycket lång halveringstid. Nackdelen med denna standardimmunetikett är en lägre känslighet än isotopsonder. Icke desto mindre mildras det genom att koppla polymeraskedjereaktion (PCR) och dot-blot-analyser. Den här metoden gör det möjligt att berika målsekvensen och identifiera den. Dessutom kan det användas som en kvantitativ applikation jämfört med en seriell utspädning av en välkänd standard. En dot-blot-applikation för att detektera Leptospira från de tre huvudkladerna i vattenprover presenteras här. Denna metod kan tillämpas på stora mängder vatten när de har koncentrerats genom centrifugering för att ge bevis på närvaron av Leptospiral-DNA. Detta är ett värdefullt och tillfredsställande verktyg för allmänna screeningändamål och kan användas för andra icke-odlingsbara bakterier som kan finnas i vatten, vilket förbättrar förståelsen av ekosystemet.
Introduction
Leptospiros hos människor har huvudsakligen sitt ursprung i miljökällor 1,2. Förekomsten av Leptospira i sjöar, floder och vattendrag är en indikator på överföring av leptospiros bland vilda djur och tamdjur och produktionsdjur som så småningom kan komma i kontakt med dessa vattenförekomster 1,3,4. Dessutom har Leptospira identifierats i icke-naturliga källor, inklusive avloppsvatten, stillastående vatten och kranvatten 5,6.
Leptospira är en bakterie som distribueras över hela världen 7,8, och miljöns roll i dess bevarande och överföring har varit välkänd. Leptospira kan överleva i dricksvatten under varierande pH och mineraler9, och i naturliga vattenförekomster1. Den kan också överleva under långa perioder i destillerat vatten10, och under konstant pH (7,8) kan den överleva upp till 152 dagar11. Dessutom kan Leptospira interagera i bakteriella konsortier för att överleva svåra förhållanden12,13. Det kan ingå i biofilm i sötvatten med Azospirillum och Sphingomonas och kan till och med växa och uthärda temperaturer över 49 °C14,15. Den kan också föröka sig i vattenmättad jord och förbli livskraftig i upp till 379 dagar16, vilket bevarar dess förmåga att orsaka sjukdomen så länge som ett år17,18. Man vet dock inte mycket om ekologin i vattendrag och hur den är fördelad inom dem.
Sedan upptäckten har studiet av släktet Leptospira baserats på serologiska tester. Det var inte förrän under det innevarande århundradet som molekylära tekniker blev mer utbredda i studiet av denna spiroket. Dot-bloten har knappast använts för att identifiera den med hjälp av (1) en isotopsond baserad på 16S rRNA och på en inter-simple sequence repeat (ISSR)19,20, (2) som en nanoguldbaserad immunanalys för human leptospiros applicerad på urin21, eller (3) som en antikroppsbaserad analys för urinprover från nötkreatur22. Tekniken föll ur bruk eftersom den ursprungligen var baserad på isotopsonder. Det är dock en välkänd teknik som, tillsammans med PCR, ger förbättrade resultat, och den anses vara säker på grund av användningen av icke-isotopiska sonder. PCR spelar en avgörande roll i anrikningen av Leptospira DNA genom att amplifiera ett specifikt DNA-fragment som kan hittas i spårmängder i ett prov. Under varje PCR-cykel fördubblas mängden av det riktade DNA-fragmentet i reaktionen. I slutet av reaktionen har amplikonet multiplicerats med en faktor på mer än en miljon23. Produkten amplifierad med PCR, som ofta inte syns i agaroselektrofores, blir synlig genom specifik hybridisering med en DIG-märkt sond i dot-blot 24,25,26.
Dot-blot-tekniken är enkel, robust och lämplig för många prover, vilket gör den tillgänglig för laboratorier med begränsade resurser. Det har använts i en mängd olika bakteriestudier, inklusive (1) orala bakterier27, (2) andra provtyper som mat och avföring28, och (3) identifiering av icke-odlingsbara bakterier29, ofta i överensstämmelse med andra molekylära tekniker. Bland fördelarna med dot-blot-tekniken är: (1) Membranet har en hög bindningskapacitet, som kan binda över 200 μg/cm2 nukleinsyror och upp till 400 μg/cm2; (2) Dot-blot-resultat kan tolkas visuellt utan att det krävs specialutrustning, och (3) de kan bekvämt förvaras i åratal i rumstemperatur (RT).
Släktet Leptospira har klassificerats i patogena, intermediära och saprofytiska klader30,31. Skillnaden mellan dessa klader kan uppnås baserat på specifika gener som lipL41, lipL32 och 16S rRNA. LipL32 finns i de patogena kladerna och uppvisar hög känslighet i olika serologiska och molekylära verktyg, medan det saknas hos saprofytarter21. Hushållningsgenen lipL41 är känd för sitt stabila uttryck och används i molekylära tekniker32, medan 16S rRNA-genen används för deras klassificering.
Denna metod kan tillämpas på stora volymer vatten när de har koncentrerats genom centrifugering. Det gör det möjligt att bedöma olika punkter och djup i en vattenförekomst för att upptäcka förekomsten av leptospiral-DNA och den klad som den tillhör. Detta verktyg är värdefullt för både ekologiska och allmänna screeningändamål och kan också användas för att upptäcka andra icke-odlingsbara bakterier som kan finnas i vatten.
Dessutom är PCR- och dot-blot-analyser tekniskt och ekonomiskt överkomliga för ett brett spektrum av laboratorier, även de som saknar sofistikerad eller dyr utrustning. Denna studie syftar till att tillämpa den digoxigeninbaserade dot-bloten för att identifiera de tre Leptospira-kladerna i vattenprover insamlade från naturliga vattenförekomster.
Bakteriella stammar
Tolv Leptospira serovarer (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassoven och Wolffi) inkluderades i denna studie. Dessa serovarer är en del av samlingen vid institutionen för mikrobiologi och immunologi, fakulteten för veterinärmedicin och zooteknik, National Autonomous University of Mexico, och de används för närvarande i mikroagglutinationstestet (MAT).
Alla Leptospira serovarer odlades i EMJH och deras DNA extraherades med hjälp av ett kommersiellt DNA-extraktionskit (se Materialtabell). En genomisk DNA-blandning av de tolv serovarerna användes som en positiv kontroll för den patogena kladen Leptospira . Som en positiv kontroll av Leptospira intermediär klad inkluderades genomiskt DNA från Leptospira fainei serovar Hurstbridge stam BUT6, och som en positiv kontroll för Leptospira saprofytkladen inkluderades även genomiskt DNA från Leptospira biflexa serovar Patoc stam Patoc I.
Negativa kontroller bestod av en tom plasmid, DNA från icke-besläktade bakterier (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumanii och Escherichia coli) och PCR-klassat vatten, som fungerade som icke-mallkontroll.
Vattenprover
Tolv provprover samlades in med hjälp av en stratifierad-slumpmässig provtagningsmetod från Cuemanco Biological and Aquaculture Research Center (CIBAC) (19° 16′ 54″ N 99° 6′ 11″ W). Dessa prover togs på tre djup: ytliga, 10 och 30 cm (Figur 1A, B). Rutinerna för att samla in vatten påverkade inte några utrotningshotade eller skyddade arter. Varje prov samlades in i ett sterilt 15 ml mikrocentrifugrör. För att samla in provet sänktes varje rör försiktigt ner i vattnet, fylldes på det valda djupet och förseglades sedan. Proverna hölls vid 22 °C och transporterades omedelbart till laboratoriet för bearbetning.
Varje prov koncentrerades genom centrifugering i sterila 1,5 ml mikrocentrifugrör vid 8000 x g i 20 minuter vid rumstemperatur. Detta steg upprepades tills alla prover koncentrerades till ett rör, som sedan användes för DNA-extraktion (Figur 1C).
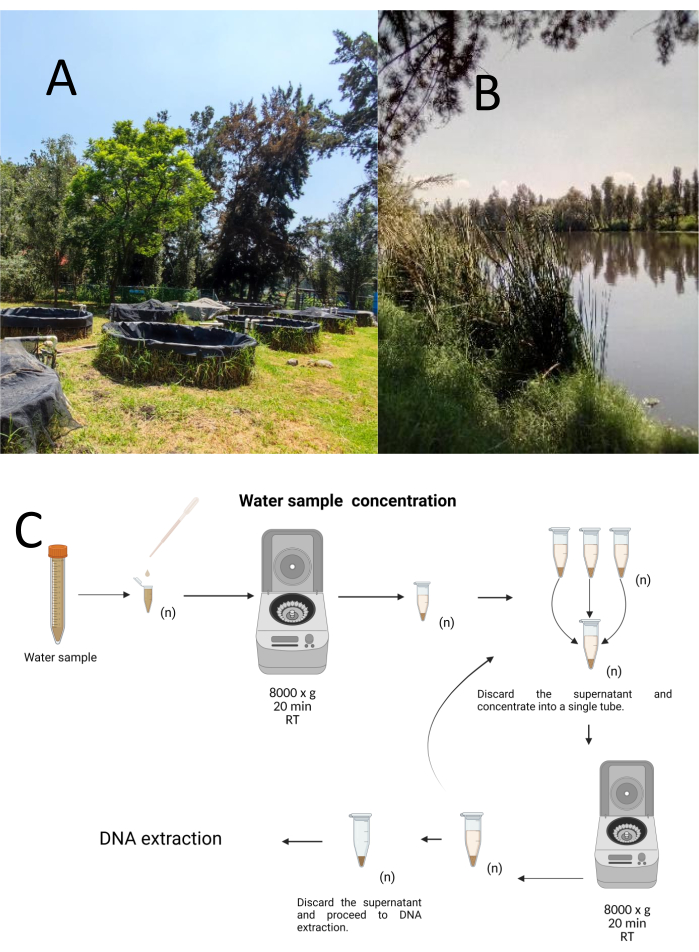
Figur 1: Koncentration av vattenprover genom centrifugering. A) Vattenprovtagningsdammar och B) Naturliga vattendrag. C) Centrifugeringsbaserad bearbetning av vattenprover i upprepade steg så många gånger som behövs (n). Klicka här för att se en större version av denna figur.
DNA-extraktion
Totalt DNA isolerades med hjälp av ett kommersiellt genomiskt DNA-kit enligt tillverkarens instruktioner (se materialtabell). DNA-extraktioner eluerades i 20 μL elueringsbuffert och DNA-koncentrationen bestämdes av en UV-spektrofotometer vid 260-280 nm och förvarades vid 4 °C tills användning.
PCR-amplifiering
PCR-målen var de 16S rRNA-, lipL41- och lipL32-generna, som identifierar DNA från släktet Leptospira och gör det möjligt att skilja mellan de tre kladerna: patogen, saprofytisk och intermediär. Både primers och sonddesign baserades på tidigare arbeten av Ahmed et al., Azali et al., Bourhy et al., Weiss et al. och Branger et al.33,34,35,36,37. Sekvensen för varje sond, primer och amplifierat fragment beskrivs i tabell 1, och deras anpassning till referenssekvenser finns i Supplementary File 1, Supplementary File 2, Supplementary File 3, Supplementary File 4 och Supplementary File 5. PCR-reagenserna och termocykliska förhållanden beskrivs i protokollavsnittet.
Amplifieringsprodukter visualiserades genom elektroforetisk separation på en 1 % agarosgel i TAE (40 mM Tris-bas, 20 mM ättiksyra och 1 mM EDTA; pH 8,3), vid 60 V i 45 minuter med etidium-bromiddetektion, som visas i kompletterande figur 1. Genomiskt DNA erhållet från varje serovar användes med koncentrationer från 6 x 106 till 1 x 104 genomekvivalenta kopior (GEq) i varje PCR-reaktion, baserat på genomstorleken för L. interrogans (4, 691, 184 bp)38 för patogen Leptospira, genomstorleken för L. biflexa (3, 956, 088 bp)39 för saprofytisk Leptospira, och genomstorleken för L. fainei serovar Hurstbridge stam BUT6 (4, 267, 324 bp) med accessionsnummer AKWZ00000000.2.
Sondernas känslighet bedömdes med DNA från varje patogen serovar, L. biflexa serovar Patoc strain Patoc I och L. fainei serovar Hurstbridge strain BUT6 i varje experiment. För att bedöma specificiteten hos PCR- och dot-blot-hybridiseringsanalysen inkluderades DNA från icke-besläktade bakterier.
Tabell 1: PCR-primrar och sonder för att amplifiera produkter för identifiering av patogena, saprofyt- och intermediära klader av Leptospira. Klicka här för att ladda ner denna tabell.
Analys av dot-blot-hybridisering
Tekniken kallas dot-blot eftersom hålen som DNA-provet placeras i har en prickform, och när de sugs in för att fixeras på plats genom vakuumsugning får de denna form. Denna teknik utvecklades av Kafatos et al.40. Tekniken gör det möjligt att göra en semikvantifiering av Leptospira i varje PCR-positivt prov. Protokollet består av en denaturering med NaOH 0,4 M vid rumstemperatur, prover med Leptospira DNA från 30 ng till 0,05 ng, motsvarande 6 x 106 till 1 x 104 leptospirer, torkas på ett nylonmembran med en 96-brunnars dot-blot-apparat. Efter immobilisering binds DNA:t till membranet genom exponering för 120 mJ UV-ljus. Varje DNA-sond konjugeras med digoxigenin-11 dUTP genom ett terminalt överföringskatalyssteg i 3′-änden (Digoxigenin är en växtsteroid erhållen från Digitalis purpurea, som används som en reporter41). Efter den stränga hybridiseringen av den märkta DNA-sonden (50 pmol) vid den specifika temperaturen på mål-DNA, visualiseras DNA-hybriderna genom kemiluminiscensreaktionen med den anti-digoxigenin alkaliska fosfatasantikroppen kovalent konjugerad med dess substrat CSPD. Luminiscensen fångas genom exponering för en röntgenfilm (figur 2).
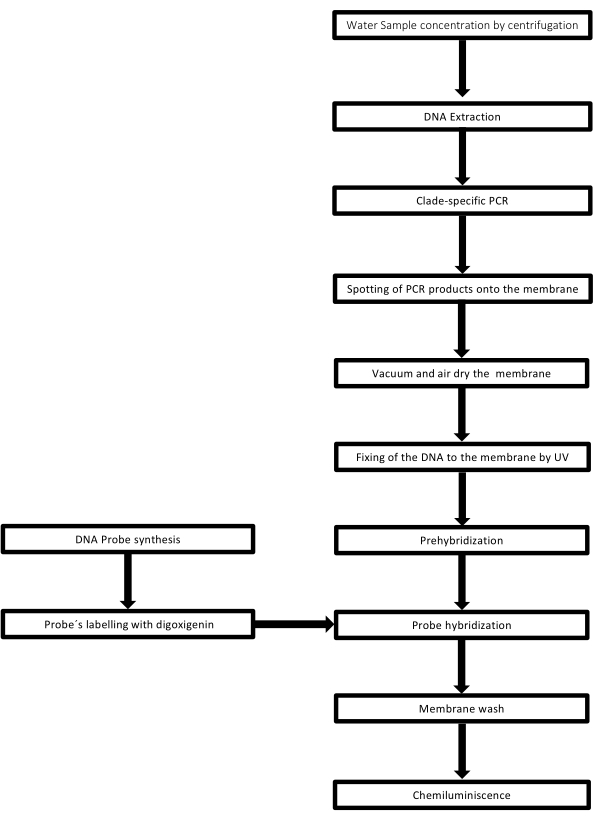
Figur 2: Steg i proceduren för PCR-dot-blot-analysen. Klicka här för att se en större version av denna figur.
Protocol
Representative Results
Discussion
De kritiska stegen i dot-blot-tekniken inkluderar (1) DNA-immobilisering, (2) blockering av de fria bindningsställena på membranet med icke-homologt DNA, (3) komplementariteten mellan sonden och målfragmentet under glödgningsförhållanden, (4) avlägsnande av den ohybridiserade sonden och (5) detektion av reportermolekylen41.
PCR-Dot-bloten har vissa begränsningar, till exempel att tekniken inte ger information om storleken på det hybridiserade fragmentet<sup cla…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi står i tacksamhetsskuld till Leptospira-samlingen vid Institutionen för mikrobiologi och immunologi, fakulteten för veterinärmedicin och zooteknik, National Autonomous University of Mexico. Vi är tacksamma för den generösa donationen av referensstammarna Leptospira ; Leptospira fainei serovar Hurstbridge stam BUT6 och Leptospira biflexa serovar Patoc stam Patoc I till Dr. Alejandro de la Peña Moctezuma. Vi tackar Dr. José Antonio Ocampo Cervantes, CIBAC-samordnaren, och personalen för deras logistiska stöd. EDT var under Terminal Project-programmet för studenter på grundnivå vid Metropolitan Autonomous University-Campus Cuajimalpa. Vi är medvetna om att det finns Biorender.com programvara för att skapa figurerna 1 och 3 till 9.
Materials
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
References
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5′-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).

