RGBradford:スマートフォンカメラによるタンパク質定量(英語)
Summary
この論文では、ブラッドフォードアッセイとスマートフォンを分析デバイスとして使用したタンパク質定量のためのプロトコルを提供します。サンプル中のタンパク質レベルは、スマートフォンで撮影したマイクロプレートの写真から抽出したカラーデータを使用して定量化できます。
Abstract
タンパク質の定量は、ライフサイエンス研究において不可欠な手順です。他のいくつかの方法の中でも、ブラッドフォードアッセイは最も使用されている方法の1つです。ブラッドフォードアッセイは広く普及しているため、その性能を向上させるための元の方法のいくつかの変更を含め、限界と利点が徹底的に報告されています。元の方法の変更点の1つは、分析機器としてスマートフォンのカメラを使用することです。この論文では、ブラッドフォードアッセイの条件に存在する3種類のクマシーブリリアントブルー色素を利用して、マイクロプレートの1枚の写真から抽出したカラーデータを使用して、サンプル中のタンパク質を正確に定量する方法について説明します。マイクロプレートでアッセイを行った後、スマートフォンのカメラで写真を撮影し、その画像から無料のオープンソースの画像解析ソフトウェアアプリケーションを使用してRGBカラーデータを抽出します。次に、タンパク質濃度が不明なサンプルの青と緑の強度の比率(RGBスケール)を使用して、標準曲線に基づいてタンパク質含有量を計算します。RGBカラーデータを用いて計算した値と従来の吸光度データを用いて計算した値との間に有意差は認められなかった。
Introduction
下流の用途(ELISA、酵素動態、ウェスタンブロッティング、タンパク質精製、質量分析など)にかかわらず、ライフサイエンスラボでの正確な分析にはタンパク質の定量が不可欠です。二次読み出し(タンパク質の質量あたりの分析種の相対レベルを計算する)としての使用に加えて、サンプル中のタンパク質レベルは、それ自体が目的の出力になることもあります。例えば、食物資源1や尿2のタンパク質レベルに関心を持つことができます。サンプル3のタンパク質濃度の測定には、UV吸光度の直接測定4、タンパク質-銅キレート化5,6、タンパク質-色素結合比色アッセイ7、タンパク質-色素結合蛍光アッセイ8など、多くの方法があります。タンパク質定量の関連性は、最も引用された文献9,10のトップ3にタンパク質測定法5,7を説明する2つの論文が存在することによって証明されています。多くの著者が、一次文献以外の文献を引用したり、何も引用しなかったりして、実際の引用を無視しているという事実にもかかわらず、Lowryタンパク質アッセイとブラッドフォードタンパク質アッセイを記述した元の論文は、それぞれ200,000>引用されています10。
ブラッドフォードアッセイの人気は、その手頃な価格、シンプルさ、スピード、感度に起因しています。このアッセイは、酸性条件下でのタンパク質と色素Coomassie Brilliant Blue Gとの相互作用に基づいています。アッセイの条件下(すなわち、低pH)では、色素は3つの形態で存在します:470 nmでλmaxを有する赤色のカチオン型。650 nmにλmaxを有する緑色の中性体。λmaxが590 nm11,12の青色アニオン型です(図1)。カチオン型は、タンパク質の非存在下で優勢である。タンパク質が色素と相互作用すると、青色の陰イオン性形態が安定化し、溶液の色が茶色がかった色から青色に顕著な変化が生じます。通常、青色の色素の濃度の変化は分光光度法で定量化され、その590〜595nmでの吸光度はアッセイ中のタンパク質の量に比例します。
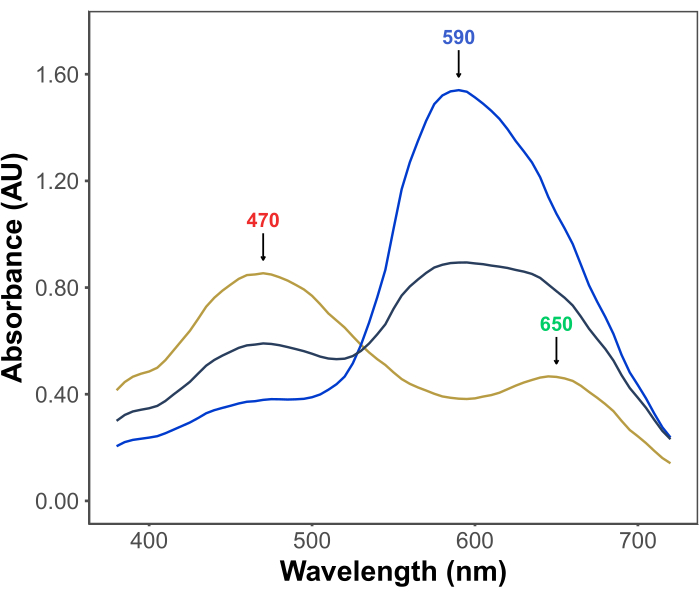
図1:ブラッドフォードアッセイの条件下でのクマシーブリリアントブルーG吸収スペクトル。 3 つの主要なピークには、赤色(470 nm)、緑色(650 nm)、青色(590 nm)の色素の λmax を示す矢印が付いています。スペクトルは、タンパク質の非存在下(黄色の線)および3 μg(灰色の線)および10 μg(青の線)のウシ血清アルブミンの存在下で記録されました。 この図の拡大版をご覧になるには、ここをクリックしてください。
ブラッドフォードアッセイの広範な使用は、いくつかの制限(例えば、異なるタンパク質に対するさまざまな応答11、脂質13と界面活性剤7による干渉)の特定と、その性能を改善するための修飾の開発(例えば、界面活性剤の添加14,15、アルカリ化14,16、吸光度の比率の使用17)の開発につながった).アッセイ自体の変更に加えて、分析シグナルを捕捉するためのスマートフォンやカメラなどの代替デバイスの使用も説明されています18,19,20。実際、スマートフォンを携帯型化学分析装置として活用する方法の開発は、活発な研究分野です。スマートフォンを使用する動機は、これらのデバイスの手頃な価格、携帯性、使いやすさ、および広範な可用性に起因しています。
この論文では、スマートフォンを分析デバイスとして使用するRGBradfordアッセイ20を使用したタンパク質定量のためのプロトコルを提供します。元のRGBradford公報20とは対照的に、ここでは、色抽出プロセスを合理化する手順が導入されている。これは、無料で入手できるソフトウェアアプリケーションを利用して、マイクロプレート画像の各ウェルから色情報を自動的に抽出し、時間と労力を大幅に節約することを含みます。これは、グラフィックエディタソフトウェアアプリケーション20を用いて、各ウェルから色データを1つずつ手動で取得する以前の方法に代わるものである。最終的には、スマートフォンで撮影したマイクロプレートの写真から抽出したカラーデータを使用して、サンプル中のタンパク質レベルを定量化できます。
Protocol
Representative Results
Discussion
この論文では、スマートフォンのカメラを使用してブラッドフォードタンパク質アッセイからのデータを記録し、カラーデータを抽出し、生物学的サンプル中のタンパク質レベルを正確に定量する方法であるRGBradfordについて説明します。元のRGBradfordメソッドとの違いの1つは、ここではImageJプラグイン22 でカラーデータを自動的に取得する手順が用いられ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
この研究は、ブラジルの国家科学技術開発評議会(CNPq)[助成金番号428048/2018-8および402556/2022-4]とブラジリア大学(ブラジル)から資金提供を受けました。著者は、この研究で使用したスマートフォンへのアクセスを提供してくれたDuarte Nuno Carvalho博士とEvelyn Santos博士(i3s、ポルトガル、ポルト)に感謝します。
Materials
| 96-well flat-bottom polystyrene microtiter plates | Jet Biofil, Guangzhou, China | TCP011096 | Any flat-bottom microplate compativle with optical reading will suffice. |
| Bovine serum albumin | Sigma-Aldrich, St. Louis, MO | A2153 | |
| Coomassie Brilliant Blue G | Sigma-Aldrich, St. Louis, MO | B0770 | |
| Ethyl alcohol | |||
| iPhone 11 | Apple | MWM02BR/A | Can be substituted with other smartphone equiped with a camera |
| iPhone 14 Pro | Apple | N/A | |
| Phosphoric acid | Sigma-Aldrich, St. Louis, MO | 695017 | |
| Redmi Note 9 Pro | XIAOMI | N/A | |
| S22 Ultra | Samsung | N/A | |
| SpectraMax 384 Plus. Microplate reader. | Molecular Devices, San Jose, CA | PLUS 384 | Any microplate reader capable of reading at 450 nm and 590 nm will work. This is optional. The method was actually created to dismiss the need of a microplate reader. |
References
- Zaguri, M., Kandel, S., Rinehart, S. A., Torsekar, V. R., Hawlena, D. Protein quantification in ecological studies: A literature review and empirical comparisons of standard methodologies. Methods in Ecology and Evolution. 12 (7), 1240-1251 (2021).
- Koga, T., et al. Mild electrical stimulation and heat shock ameliorates progressive proteinuria and renal inflammation in mouse model of Alport syndrome. PLoS One. 7 (8), e43852 (2012).
- Peterson, G. L. Determination of total protein. Methods in Enzymology. 91, 95-119 (1983).
- Goldfarb, A. R., Saidel, L. J., Mosovich, E. The ultraviolet absorption spectra of proteins. The Journal of Biological Chemistry. 193 (1), 397-404 (1951).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. The Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Datki, Z., et al. Application of BisANS fluorescent dye for developing a novel protein assay. PLoS One. 14 (4), e0215863 (2019).
- Van Noorden, R., Maher, B., Nuzzo, R. The top 100 papers. Nature. 514 (7524), 550-553 (2014).
- . Scopus Available from: https://www.scopus.com/ (2022)
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Analytical Biochemistry. 151 (2), 369-374 (1985).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie Blue G. Analytical Biochemistry. 209 (2), 258-266 (1993).
- Pande, S. V., Murthy, M. S. R. A modified micro-Bradford procedure for elimination of interference from sodium dodecyl sulfate, other detergents, and lipids. Analytical Biochemistry. 220 (2), 424-426 (1994).
- Gogstad, G. O., Krutnes, M. -. B. Measurement of protein in cell suspensions using the Commassie brilliant blue dye-binding assay. Analytical Biochemistry. 126 (2), 355-359 (1982).
- Friedenauer, S., Berlet, H. H. Sensitivity and variability of the Bradford protein assay in the presence of detergents. Analytical Biochemistry. 178 (2), 263-268 (1989).
- Stoscheck, C. M. Increased uniformity in the response of the Coomassie blue G protein assay to different proteins. Analytical Biochemistry. 184 (1), 111-116 (1990).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: Theoretical and experimental studies. Analytical Biochemistry. 236 (2), 302-308 (1996).
- Gee, C. T., Kehoe, E., Pomerantz, W. C. K., Penn, R. L. Quantifying protein concentrations using smartphone colorimetry: A new method for an established test. Journal of Chemical Education. 94 (7), 941-945 (2017).
- de Camargo, C., Vicentini, M., Gobbi, A., Martinez, D., Lima, R. Smartphone for point-of-care quantification of protein by Bradford assay. Journal of the Brazilian Chemical Society. 28 (4), 689-693 (2016).
- Moreira, D. C. RGBradford: Accurate measurement of protein concentration using a smartphone camera and the blue to green intensity ratio. Analytical Biochemistry. 655, 114839 (2022).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. Journal of Visualized Experiments. (38), 1918 (2010).
- Angelani, C. R., et al. A metabolic control analysis approach to introduce the study of systems in biochemistry: the glycolytic pathway in the red blood cell: Metabolic control analysis and the glycolytic pathway. Biochemistry and Molecular Biology Education. 46 (5), 502-515 (2018).

