Fenotypisk profilering av humane stamcellederiverte midbrain dopaminerge nevroner
Summary
Denne protokollen beskriver celledyrking av humane midbrain dopaminerge nevroner, etterfulgt av immunologisk farging og generering av neuronale fenotypiske profiler fra ervervede mikroskopiske bilder med høyt innhold som tillater identifisering av fenotypiske variasjoner på grunn av genetiske eller kjemiske modulasjoner.
Abstract
Parkinsons sykdom (PD) er knyttet til en rekke cellebiologiske prosesser som forårsaker midbrain dopaminerge (mDA) nevrontap. Mange nåværende in vitro PD-cellulære modeller mangler kompleksitet og tar ikke hensyn til flere fenotyper. Fenotypisk profilering i humane induserte pluripotente stamceller (iPSC)-avledede mDA-nevroner kan adressere disse manglene ved samtidig å måle en rekke nevrale fenotyper i en PD-relevant celletype parallelt. Her beskriver vi en protokoll for å oppnå og analysere fenotypiske profiler fra kommersielt tilgjengelige humane mDA-nevroner. Et nevronspesifikt fluorescerende fargepanel brukes til å visualisere kjernefysiske, α-synuclein, tyrosinhydroksylase (TH) og mikrotubuli-assosiert protein 2 (MAP2) relaterte fenotyper. Den beskrevne fenotypiske profileringsprotokollen er skalerbar da den bruker 384-brønnplater, automatisk væskehåndtering og mikroskopi med høy gjennomstrømning. Nytten av protokollen eksemplifiseres ved bruk av friske donor mDA-nevroner og mDA-nevroner som bærer den PD-koblede G2019S-mutasjonen i Leucin-rik gjentatt kinase 2 (LRRK2) genet. Begge cellelinjene ble behandlet med LRRK2-kinasehemmeren PFE-360 og fenotypiske forandringer ble målt. I tillegg demonstrerer vi hvordan flerdimensjonale fenotypiske profiler kan analyseres ved hjelp av clustering eller maskinlæringsdrevne overvåkede klassifiseringsmetoder. Den beskrevne protokollen vil spesielt interessere forskere som arbeider med modellering av nevronsykdom eller studerer kjemiske sammensatte effekter i humane nevroner.
Introduction
En rekke cellebiologiske prosesser forstyrres i Parkinsons sykdom (PD). For eksempel har mitokondriell dysfunksjon, oksidativt stress, proteinnedbrytningsdefekter, forstyrrelse av vesikulær trafikk og endolysosomal funksjon vært assosiert med midbrain dopaminerge (mDA) nevrontap, er ofte observert i PD1. Derfor ser PD ut til å involvere flere sykdomsmekanismer som kan samhandle med og forverre hverandre. En nyttig måte å undersøke dette mekanistiske samspillet på er å lage et omfattende fenotypisk fingeravtrykk eller profil av midbrain dopaminerge (mDA) nevroner.
Fenotypisk profilering er en tilnærming som innebærer å lage en profil av en prøve basert på en samling av målbare egenskaper, og for det andre innebærer det å gjøre spådommer om et utvalg basert på denne profilen 2,3. Målet med profilering er å fange opp et mangfoldig utvalg av funksjoner, hvorav noen kanskje ikke tidligere har vært forbundet med en sykdom eller behandling3. Som et resultat kan profilering avsløre uventede biologiske prosesser. Fenotypisk profilering er vanligvis avhengig av fluorescerende fargede celler, og standardiserte analyser, for eksempel Cell Painting, er utviklet for å lage fenotypiske profiler4. Nylig har fenotypisk profilering for eksempel blitt brukt til karakterisering av små molekyler eller nøyaktig prediksjon av PD-subtyper utelukkende basert på pasientavledede fibroblaster 5,6. Til tross for disse fremskrittene har fenotypisk profilering sjelden blitt brukt på postmitotiske differensierte celler, slik som humane induserte pluripotente stamceller (iPSC)-avledede mDA-nevroner som uttrykker PD-koblede mutasjoner som LRRK2 G2019S. Betydelige utfordringer med iPSC-avledede modeller inkluderer tilstedeværelsen av subtile eller variable patologiske trekk på tvers av differensieringspartier eller genotyper, og det faktum at isolerte PD-fenotyper ikke fanger opp sykdommens fulle kompleksitet. Videre, mens iPSC nevronmodeller er fysiologisk relevante, blir de sjelden brukt i PD-oppdagelsesprosesser på grunn av bekymringer om teknisk kompleksitet 7,8.
Vi har tidligere utviklet en robust metodikk for å måle flere PD-relaterte patofysiologiske fenotyper i humane mDA-nevroner som både er følsomme for genetiske og kjemiske forbindelsesinduserte fenotypiske endringer9. Denne artikkelen beskriver i detalj en ytterligere optimalisert versjon av denne metoden for å lage fenotypiske profiler fra mDA-nevroner (figur 1). Denne protokollen har flere fordeler i forhold til de tidligere beskrevne fenotypiske profileringsmetodene, for eksempel bruk av høykvalitets mDA-nevroner og teknisk reproduserbarhet. For første gang muliggjør denne protokollen fenotypisk profilering i fysiologisk relevante postmitotiske mDA-nevroner etter kjemiske forstyrrelser på en svært skalerbar måte. Fullt differensierte og kryopreserverte mDA-nevroner er kommersielt tilgjengelige, noe som reduserer batch-til-batch-differensieringsvariabiliteten betydelig. For det andre kan teknisk variabilitet reduseres ytterligere ved å bruke et veldefinert eksperimentelt design (dvs. kulturvarighet eller unngå kantbrønner), automatisert væskehåndtering og automatisert mikroskopi. I tillegg er de første trinnene i fenotypisk profilanalyse ved hjelp av ikke-overvåket klynging eller veiledede klassifiseringstilnærminger skissert her, noe som indikerer hvordan fenotypiske profileringsdata kan analyseres. Denne protokollen vil være til nytte for forskere som er interessert i fenotypiske endringer av mDA-nevroner indusert av genetiske eller kjemiske forstyrrelser, spesielt når et svært skalerbart studieoppsett kreves, for eksempel under screeningskampanjer eller når effekten av et mindre antall forbindelser skal studeres, for eksempel for å bestemme toksiske effekter. Oppsummert forventes det at anvendelsen av fenotypisk profilering av humane nevroner er en verdifull teknikk for å studere komplekse sykdomsrelaterte fenotyper og karakterisere de cellulære effektene av legemiddelkandidater.
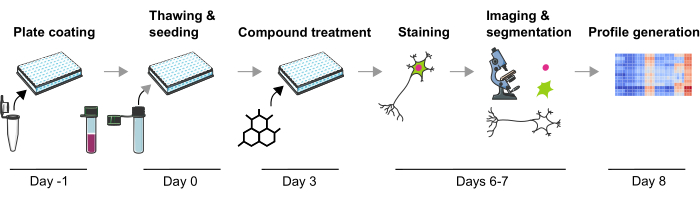
Figur 1: Skjematisk fremstilling av eksperimentell protokoll for å generere bildebaserte fenotypiske profiler fra humane iPSC-deriverte mDA-nevroner. Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
Fenotypisk profilering er en teknikk for å måle et stort antall fenotyper i celler ved å bruke fluorescerende farginger, mikroskopi og bildeanalyse3. Fenotypiske profiler kan oppnås og sammenlignes på tvers av cellelinjer eller andre eksperimentelle forhold for å forstå komplekse endringer i cellulær biologi som kan gå ubemerket hen når du bruker en enkelt avlesning. Her beskriver vi anvendelsen av fenotypisk profilering på humane iPSC-avledede mDA-nevroner, en celletype som ofte brukes…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Forfatterne vil gjerne takke alle kolleger på Ksilink for deres verdifulle hjelp og diskusjoner som førte til utformingen av den presenterte protokollen.
Materials
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse – Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit – Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
References
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson’s disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson’s disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson’s Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson’s Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson’s disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson’s disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson’s disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson’s disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).

