Perfil fenotípico de neuronas dopaminérgicas del mesencéfalo derivadas de células madre humanas
Summary
Este protocolo describe el cultivo celular de neuronas dopaminérgicas del mesencéfalo humano, seguido de la tinción inmunológica y la generación de perfiles fenotípicos neuronales a partir de imágenes microscópicas adquiridas de alto contenido que permiten la identificación de variaciones fenotípicas debidas a modulaciones genéticas o químicas.
Abstract
La enfermedad de Parkinson (EP) está relacionada con una serie de procesos biológicos celulares que causan la pérdida de neuronas dopaminérgicas del mesencéfalo (mDA). Muchos modelos celulares actuales de EP in vitro carecen de complejidad y no tienen en cuenta múltiples fenotipos. El perfil fenotípico en neuronas mDA derivadas de células madre pluripotentes inducidas humanas (iPSC) puede abordar estas deficiencias midiendo simultáneamente una serie de fenotipos neuronales en un tipo de célula relevante para la EP en paralelo. Aquí, describimos un protocolo para obtener y analizar perfiles fenotípicos de neuronas mDA humanas disponibles comercialmente. Se utiliza un panel de tinción fluorescente específico de neurona para visualizar los fenotipos relacionados con la proteína nuclear, la α-sinucleína, la tirosina hidroxilasa (TH) y la proteína 2 asociada a microtúbulos (MAP2). El protocolo de perfil fenotípico descrito es escalable, ya que utiliza placas de 384 pocillos, manipulación automática de líquidos y microscopía de alto rendimiento. La utilidad del protocolo se ejemplifica utilizando neuronas mDA de donantes sanos y neuronas mDA portadoras de la mutación G2019S ligada a PD en el gen de la quinasa repetida 2 rica en leucina (LRRK2). Ambas líneas celulares fueron tratadas con el inhibidor de la quinasa LRRK2 PFE-360 y se midieron los cambios fenotípicos. Además, demostramos cómo se pueden analizar los perfiles fenotípicos multidimensionales utilizando métodos de clasificación supervisada basados en clústeres o aprendizaje automático. El protocolo descrito interesará especialmente a los investigadores que trabajan en el modelado de enfermedades neuronales o en el estudio de los efectos de los compuestos químicos en las neuronas humanas.
Introduction
En la enfermedad de Parkinson (EP) se alteran diversos procesos biológicos celulares. Por ejemplo, la disfunción mitocondrial, el estrés oxidativo, los defectos de degradación de proteínas, la interrupción del tráfico vesicular y la función endolisosomal se han asociado con la pérdida de neuronas dopaminérgicas del mesencéfalo (mDA), se observan comúnmente en la EP1. Por lo tanto, la EP parece implicar múltiples mecanismos de enfermedad que pueden interactuar y empeorarse entre sí. Una forma útil de investigar esta interacción mecanicista es la creación de una huella dactilar fenotípica completa o un perfil de las neuronas dopaminérgicas del mesencéfalo (mDA).
El perfil fenotípico es un enfoque que implica la creación de un perfil de una muestra basado en una colección de características medibles y, en segundo lugar, implica hacer predicciones sobre una muestra basada en este perfil 2,3. El objetivo de la elaboración de perfiles es capturar una amplia gama de características, algunas de las cuales pueden no haberse asociado previamente con una enfermedad o tratamiento3. Como resultado, la elaboración de perfiles puede revelar procesos biológicos inesperados. El perfil fenotípico generalmente se basa en células teñidas con fluorescencia, y se han desarrollado ensayos estandarizados, como Cell Painting, para crear perfiles fenotípicos4. Recientemente, el perfil fenotípico se ha aplicado, por ejemplo, para la caracterización de moléculas pequeñas o la predicción precisa de subtipos de EP basados únicamente en fibroblastos derivados del paciente 5,6. A pesar de estos avances, el perfil fenotípico rara vez se ha aplicado a células diferenciadas postmitóticas, como las neuronas mDA derivadas de células madre pluripotentes inducidas humanas (iPSC) que expresan mutaciones ligadas a la enfermedad de Parkinson, como LRRK2 G2019S. Los desafíos significativos de los modelos derivados de iPSC incluyen la presencia de características patológicas sutiles o variables en lotes de diferenciación o genotipos, y el hecho de que los fenotipos aislados de EP no capturan toda la complejidad de la enfermedad. Además, aunque los modelos neuronales de iPSC son fisiológicamente relevantes, rara vez se utilizan en los procesos de descubrimiento de fármacos en la EP debido a las preocupaciones sobre la complejidad técnica 7,8.
Previamente desarrollamos una metodología robusta para medir múltiples fenotipos fisiopatológicos relacionados con la EP en neuronas mDA humanas que son sensiblesa los cambios fenotípicos inducidos por compuestos genéticos y químicos. Este artículo describe en detalle una versión optimizada de esta metodología para crear perfiles fenotípicos a partir de neuronas mDA (Figura 1). Este protocolo tiene varias ventajas sobre los enfoques de perfiles fenotípicos descritos anteriormente, como el uso de neuronas mDA de alta calidad y la reproducibilidad técnica. Por primera vez, este protocolo permite el perfil fenotípico en neuronas mDA postmitóticas fisiológicamente relevantes después de perturbaciones químicas de una manera altamente escalable. Las neuronas mDA totalmente diferenciadas y criopreservadas están disponibles comercialmente, lo que disminuye significativamente la variabilidad de la diferenciación de un lote a otro. En segundo lugar, la variabilidad técnica puede reducirse aún más mediante el uso de un diseño experimental bien definido (es decir, la duración del cultivo o evitar los pozos de borde), la manipulación automatizada de líquidos y la microscopía automatizada. Además, aquí se describen los pasos iniciales del análisis del perfil fenotípico mediante enfoques de agrupamiento no supervisado o clasificación supervisada, lo que indica cómo se pueden analizar los datos del perfil fenotípico. Este protocolo será de utilidad para los investigadores interesados en los cambios fenotípicos de las neuronas mDA inducidos por perturbaciones genéticas o químicas, específicamente cuando se requiere una configuración de estudio altamente escalable, por ejemplo, durante las campañas de cribado o cuando se van a estudiar los efectos de un número menor de compuestos, por ejemplo, para determinar los efectos tóxicos. En resumen, se anticipa que la aplicación del perfil fenotípico de las neuronas humanas es una técnica valiosa para estudiar fenotipos complejos relacionados con enfermedades y caracterizar los efectos celulares de los candidatos a fármacos.
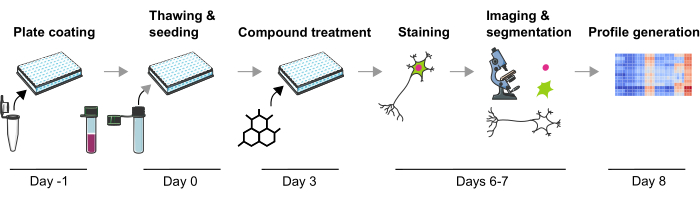
Figura 1: Representación esquemática del protocolo experimental para generar perfiles fenotípicos basados en imágenes a partir de neuronas mDA derivadas de iPSC humanas. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
El perfil fenotípico es una técnica para medir un gran número de fenotipos en las células mediante la aplicación de tinciones fluorescentes, microscopía y análisis de imágenes3. Los perfiles fenotípicos se pueden obtener y comparar a través de líneas celulares u otras condiciones experimentales para comprender cambios complejos en la biología celular que podrían pasar desapercibidos cuando se usa una sola lectura. En este trabajo describimos la aplicación del perfil fenotípico a las…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Los autores desean agradecer a todos los colegas de Ksilink por su valiosa ayuda y discusiones que condujeron al diseño del protocolo presentado.
Materials
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse – Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit – Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
References
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson’s disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson’s disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson’s Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson’s Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson’s disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson’s disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson’s disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson’s disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).

