Fenotypisk profilering av humana stamcellshärledda dopaminerga neuroner i mellanhjärnan
Summary
Detta protokoll beskriver cellodling av humana dopaminerga neuroner i mellanhjärnan, följt av immunologisk färgning och generering av neuronala fenotypiska profiler från förvärvade mikroskopiska bilder med högt innehåll som möjliggör identifiering av fenotypiska variationer på grund av genetiska eller kemiska modulationer.
Abstract
Parkinsons sjukdom (PD) är kopplad till en rad cellbiologiska processer som orsakar förlust av neuroner i mellanhjärnan (mDA). Många nuvarande in vitro PD-cellulära modeller saknar komplexitet och tar inte hänsyn till flera fenotyper. Fenotypisk profilering i humant inducerade pluripotenta stamceller (iPSC)-härledda mDA-neuroner kan åtgärda dessa brister genom att samtidigt mäta en rad neuronala fenotyper i en PD-relevant celltyp parallellt. Här beskriver vi ett protokoll för att erhålla och analysera fenotypiska profiler från kommersiellt tillgängliga humana mDA-neuroner. En neuronspecifik fluorescerande färgningspanel används för att visualisera de nukleära, α-synuklein, tyrosinhydroxylas (TH) och mikrotubuliassocierat protein 2 (MAP2) relaterade fenotyperna. Det beskrivna fenotypiska profileringsprotokollet är skalbart eftersom det använder 384-brunnars plattor, automatisk vätskehantering och mikroskopi med hög genomströmning. Nyttan av protokollet exemplifieras med hjälp av friska mDA-neuroner från donatorer och mDA-neuroner som bär på den PD-länkade G2019S-mutationen i genen Leucine-rich repeat kinase 2 (LRRK2). Båda cellinjerna behandlades med LRRK2-kinashämmaren PFE-360 och fenotypiska förändringar mättes. Dessutom visar vi hur flerdimensionella fenotypiska profiler kan analyseras med hjälp av klustring eller maskininlärningsdrivna övervakade klassificeringsmetoder. Det beskrivna protokollet kommer särskilt att intressera forskare som arbetar med modellering av neuronala sjukdomar eller studerar kemiska sammansatta effekter i mänskliga neuroner.
Introduction
En mängd olika cellbiologiska processer störs vid Parkinsons sjukdom (PD). Till exempel har mitokondriell dysfunktion, oxidativ stress, proteinnedbrytningsdefekter, störning av vesikulär trafik och endolysosomal funktion associerats med förlust av neuroner i mellanhjärnan (mDA), som vanligtvis observeras vid PD1. Därför verkar PD involvera flera sjukdomsmekanismer som kan interagera med och förvärra varandra. Ett användbart sätt att undersöka detta mekanistiska samspel är att skapa ett omfattande fenotypiskt fingeravtryck eller profil av mitthjärnans dopaminerga (mDA) neuroner.
Fenotypisk profilering är ett tillvägagångssätt som innebär att man skapar en profil av ett prov baserat på en samling mätbara egenskaper, och för det andra innebär det att man gör förutsägelser om ett prov baserat på denna profil 2,3. Målet med profilering är att fånga ett brett spektrum av egenskaper, av vilka några kanske inte tidigare har förknippats med en sjukdom eller behandling3. Som ett resultat kan profilering avslöja oväntade biologiska processer. Fenotypisk profilering förlitar sig vanligtvis på fluorescerande färgade celler, och standardiserade analyser, såsom cellmålning, har utvecklats för att skapa fenotypiska profiler4. På senare tid har fenotypisk profilering till exempel tillämpats för karakterisering av små molekyler eller noggrann förutsägelse av PD-subtyper enbart baserat på fibroblaster från patienter 5,6. Trots dessa framsteg har fenotypisk profilering sällan tillämpats på postmitotiska differentierade celler, såsom humant inducerade pluripotenta stamceller (iPSC)-härledda mDA-neuroner som uttrycker PD-kopplade mutationer såsom LRRK2 G2019S. Betydande utmaningar med iPSC-härledda modeller inkluderar förekomsten av subtila eller variabla patologiska egenskaper över differentieringssatser eller genotyper, och det faktum att isolerade PD-fenotyper inte fångar sjukdomens fulla komplexitet. Dessutom, även om iPSC-neuronala modeller är fysiologiskt relevanta, används de sällan i PD-läkemedelsupptäcktsprocesser på grund av oro för teknisk komplexitet 7,8.
Vi har tidigare utvecklat en robust metodik för att mäta flera PD-relaterade patofysiologiska fenotyper i humana mDA-neuroner som både är känsliga för genetiska och kemiska föreningsinducerade fenotypiska förändringar9. Den här artikeln beskriver i detalj en ytterligare optimerad version av denna metod för att skapa fenotypiska profiler från mDA-neuroner (figur 1). Detta protokoll har flera fördelar jämfört med de tidigare beskrivna fenotypiska profileringsmetoderna, såsom användningen av högkvalitativa mDA-neuroner och teknisk reproducerbarhet. För första gången möjliggör detta protokoll fenotypisk profilering i fysiologiskt relevanta postmitotiska mDA-neuroner efter kemiska störningar på ett mycket skalbart sätt. Fullt differentierade och kryokonserverade mDA-neuroner är kommersiellt tillgängliga, vilket avsevärt minskar differentieringsvariabiliteten mellan batcher. För det andra kan den tekniska variabiliteten minskas ytterligare genom att använda en väldefinierad experimentell design (dvs. odlingstid eller undvikande av kantbrunnar), automatiserad vätskehantering och automatiserad mikroskopi. Dessutom beskrivs de inledande stegen i fenotypisk profilanalys med hjälp av oövervakad klustring eller övervakade klassificeringsmetoder här, vilket indikerar hur fenotypiska profileringsdata kan analyseras. Detta protokoll kommer att vara användbart för forskare som är intresserade av fenotypiska förändringar av mDA-neuroner inducerade av genetiska eller kemiska störningar, särskilt när en mycket skalbar studieuppställning krävs, till exempel under screeningkampanjer eller när effekterna av ett mindre antal föreningar ska studeras, till exempel för att bestämma toxiska effekter. Sammanfattningsvis förväntas tillämpningen av fenotypisk profilering av humana nervceller vara en värdefull teknik för att studera komplexa sjukdomsrelaterade fenotyper och karakterisera de cellulära effekterna av läkemedelskandidater.
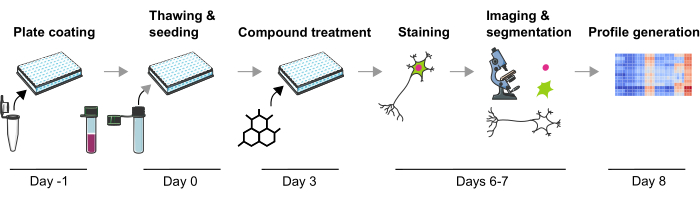
Figur 1: Schematisk skildring av det experimentella protokollet för att generera bildbaserade fenotypiska profiler från humana iPSC-härledda mDA-neuroner. Klicka här för att se en större version av denna figur.
Protocol
Representative Results
Discussion
Fenotypisk profilering är en teknik för att mäta ett stort antal fenotyper i celler genom att tillämpa fluorescerande färgningar, mikroskopi och bildanalys3. Fenotypiska profiler kan erhållas och jämföras över cellinjer eller andra experimentella förhållanden för att förstå komplexa förändringar i cellbiologi som kan gå obemärkt förbi när man använder en enda avläsning. Här beskriver vi tillämpningen av fenotypisk profilering på humana iPSC-härledda mDA-neuroner, en cellt…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Författarna vill tacka alla kollegor på Ksilink för deras värdefulla hjälp och diskussioner som ledde fram till utformningen av det presenterade protokollet.
Materials
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse – Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit – Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
References
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson’s disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson’s disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson’s Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson’s Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson’s disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson’s disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson’s disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson’s disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).

