सिमुलेशन अध्ययन में जटिल लिपिड मिश्रण का उपयोग करते हुए यथार्थवादी झिल्ली मॉडलिंग
Summary
संरचना और संरचना में झिल्ली लिपिड विविधता सेलुलर प्रक्रियाओं में एक महत्वपूर्ण योगदानकर्ता है और बीमारी का एक मार्कर हो सकता है। आणविक गतिशीलता सिमुलेशन हमें परमाणु संकल्प पर बायोमोलेक्यूल्स के साथ झिल्ली और उनकी बातचीत का अध्ययन करने की अनुमति देते हैं। यहां, हम जटिल झिल्ली प्रणालियों के निर्माण, चलाने और विश्लेषण करने के लिए एक प्रोटोकॉल प्रदान करते हैं।
Abstract
लिपिड कोशिका झिल्ली के संरचनात्मक निर्माण खंड हैं; लिपिड प्रजातियां सेल ऑर्गेनेल और जीवों में भिन्न होती हैं। इस किस्म के परिणामस्वरूप झिल्ली में विभिन्न यांत्रिक और संरचनात्मक गुण होते हैं जो सीधे इस इंटरफ़ेस पर होने वाले अणुओं और प्रक्रियाओं को प्रभावित करते हैं। लिपिड संरचना गतिशील है और सेल सिग्नलिंग प्रक्रियाओं को संशोधित करने के लिए काम कर सकती है। बायोमोलेक्यूल्स के बीच बातचीत की भविष्यवाणी करने और प्रयोगात्मक अवलोकनों को आणविक अंतर्दृष्टि प्रदान करने के लिए कम्प्यूटेशनल दृष्टिकोण का तेजी से उपयोग किया जाता है। आणविक गतिशीलता (एमडी) सांख्यिकीय यांत्रिकी पर आधारित एक तकनीक है जो उन पर कार्य करने वाले बलों के आधार पर परमाणुओं के आंदोलन की भविष्यवाणी करती है। एमडी सिमुलेशन का उपयोग बायोमोलेक्यूल्स की बातचीत को चिह्नित करने के लिए किया जा सकता है। यहां, हम संक्षेप में तकनीक का परिचय देते हैं, शुरुआती लोगों के लिए व्यावहारिक कदमों की रूपरेखा तैयार करते हैं जो लिपिड बाइलेयर का अनुकरण करने में रुचि रखते हैं, शुरुआती अनुकूल सॉफ्टवेयर के साथ प्रोटोकॉल का प्रदर्शन करते हैं, और विकल्पों, चुनौतियों और प्रक्रिया के महत्वपूर्ण विचारों पर चर्चा करते हैं। विशेष रूप से, हम सिमुलेशन में उपयुक्त हाइड्रोफोबिक और यांत्रिक वातावरण को पकड़ने के लिए रुचि के सेल झिल्ली को मॉडल करने के लिए जटिल लिपिड मिश्रण का उपयोग करने की प्रासंगिकता पर जोर देते हैं। हम कुछ उदाहरणों पर भी चर्चा करते हैं जहां झिल्ली संरचना और गुण अन्य बायोमोलेक्यूल्स के साथ बाइलेयर की बातचीत को संशोधित करते हैं।
Introduction
लिपिड झिल्ली के प्रमुख घटक हैं, जो कोशिकाओं के लिए सीमाएं प्रदान करते हैं और इंट्रासेल्युलर कंपार्टमेंटलाइजेशन 1,2,3 को सक्षम करते हैं। लिपिड एम्फीफिलिक होते हैं, जिसमें एक ध्रुवीय सिर समूह और दो हाइड्रोफोबिक फैटी एसिड पूंछ होते हैं; ये पानी के साथ हाइड्रोफोबिक श्रृंखलाओं के संपर्क को कम करने के लिए एक बाईलेयर में स्वयं इकट्ठा होते हैं 3,4. हाइड्रोफिलिक हेड ग्रुप और हाइड्रोफोबिक पूंछ के विभिन्न संयोजनों के परिणामस्वरूप जैविक झिल्ली में लिपिड के विभिन्न वर्ग होते हैं, जैसे ग्लिसरोफॉस्फोलिपिड्स, स्फिंगोलिपिड्स और स्टेरोल्स (चित्रा 1)1,5,6। ग्लिसरोफॉस्फोलिपिड्स यूकेरियोटिक कोशिका झिल्ली के प्राथमिक निर्माण खंड हैं जो ग्लिसरोफॉस्फेट, लंबी श्रृंखला फैटी एसिड और कम आणविक भारके सिर समूहों से बने होते हैं। लिपिड नामकरण सिर समूहों में अंतर पर आधारित है; उदाहरणों में फॉस्फेटिडिल-कोलीन (पीसी), फॉस्फेटिडिल-इथेनॉलमाइन (पीई), फॉस्फेटिडिल-सेरीन (पीएस), फॉस्फेटिडिल-ग्लिसरॉल (पीजी), फॉस्फेटिडिल-इनोसिटोल (पीआई), या असंशोधित फॉस्फेटिडिक एसिड (पीए) 5,6 शामिल हैं। हाइड्रोफोबिक पूंछ के लिए, रीढ़ की संरचना के साथ संतृप्ति की लंबाई और डिग्री भिन्न होती है। संभावित संयोजन कई हैं, जिसके परिणामस्वरूप स्तनधारी कोशिकाओं में हजारों लिपिड प्रजातियांहैं। झिल्ली लिपिड संरचना में परिवर्तन विभिन्न यांत्रिक और संरचनात्मक झिल्ली गुणों को जन्म देता है जो अभिन्न झिल्ली प्रोटीन और परिधीय प्रोटीन 2,6 दोनों की गतिविधि को प्रभावित करते हैं।
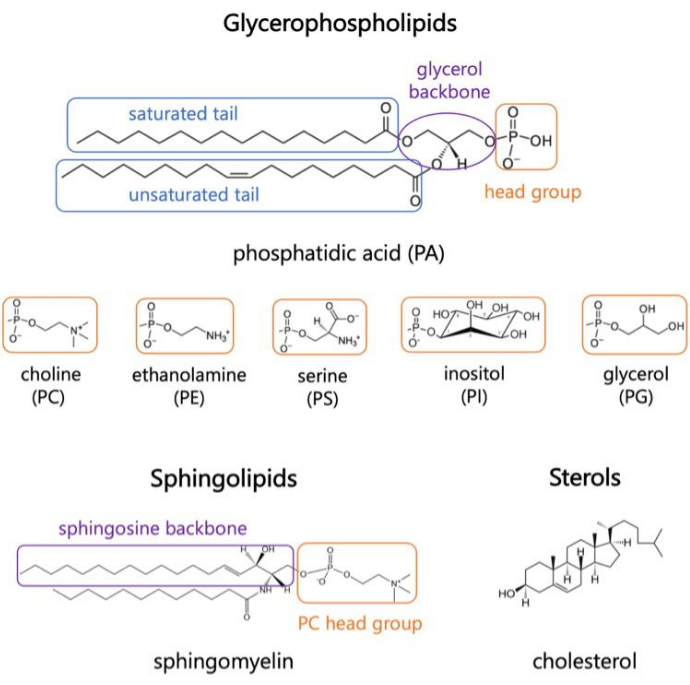
चित्र 1. प्रतिनिधि लिपिड संरचनाएं। फैटी एसिड पूंछ नीले बक्से में दिखाए जाते हैं, नारंगी में सामान्य लिपिड हेड समूह, और बैंगनी में नमूना बैकबोन। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
लिपिड सेलुलर प्रक्रियाओं में सक्रिय खिलाड़ी हैं, सिग्नलिंग कैस्केड में प्रोटीन सक्रियण, और स्वस्थ सेल होमियोस्टैसिस 8,9। परिवर्तित लिपिड गतिशीलता संक्रमण का परिणाम है या रोग10,11,12,13,14,15 के रोगजनन के मार्कर हो सकते हैं। कोशिका के लिए बाधाओं के रूप में, झिल्ली लिपिड का अध्ययन और छोटे अणुओं के पारगम्यता में उनकी भूमिका दवा वितरण प्रणाली और झिल्ली विघटन तंत्र16,17 के लिए प्रासंगिक है। रासायनिक विविधता और ऑर्गेनेल, ऊतकों और जीवों में लिपिड प्रजातियों के विभिन्न अनुपात जटिलझिल्ली गतिशीलता को जन्म देते हैं। इसलिए लिपिड बाइलेयर के मॉडलिंग अध्ययन में इन विशेषताओं को बनाए रखना महत्वपूर्ण है, खासकर जब एक अध्ययन का लक्ष्य झिल्ली के साथ अन्य बायोमोलेक्यूल्स की बातचीत की जांच करना है। एक मॉडल में विचार करने के लिए लिपिड प्रजातियां जीव और रुचि के सेलुलर डिब्बे पर निर्भर करती हैं। उदाहरण के लिए, पीजी लिपिड प्रकाश संश्लेषक बटेरिया18 में इलेक्ट्रॉन हस्तांतरण के लिए महत्वपूर्ण हैं, जबकि फॉस्फोराइलेटेड इनोसिटोल लिपिड (पीआईपी) प्लाज्मा झिल्ली (पीएम) गतिशीलता और स्तनधारी कोशिकाओं में सिग्नलिंग कैस्केड 19,20 में प्रमुख खिलाड़ी हैं। कोशिका के अंदर, पीएम, एंडोप्लाज्मिक रेटिकुलम (ईआर), गोल्गी और माइटोकॉन्ड्रियल झिल्ली में अद्वितीय लिपिड बहुतायत होते हैं जो उनके कार्य को प्रभावित करते हैं। उदाहरण के लिए, ईआर लिपिड बायोजेनेसिस के लिए केंद्र है और कोलेस्ट्रॉल को पीएम और गोल्गी तक पहुंचाता है; इसमें पीसी और पीई की प्रचुरता के साथ एक उच्च लिपिड विविधता होती है, लेकिन कम स्टेरोल सामग्री, जो झिल्ली तरलता21,22,23,24 को बढ़ावा देती है। इसके विपरीत, पीएम जीव25 के आधार पर सैकड़ों और यहां तक कि हजारों लिपिड प्रजातियों को शामिल करता है, इसमें स्फिंगोलिपिड्स और कोलेस्ट्रॉल के उच्च स्तर होते हैं जो इसे सेल24 में अन्य झिल्ली की तुलना में एक विशिष्ट कठोरता देते हैं। पत्रक विषमता को पीएम जैसी झिल्लियों के लिए माना जाना चाहिए, जिसमें स्फिंगोमाइलिन, पीसी और कोलेस्ट्रॉल से भरपूर एक बाहरी पत्रक होता है, और पीई, पीआई और पीएस में समृद्ध एक आंतरिक पत्रक होता है जो सिग्नलिंग कैस्केड24 के लिए महत्वपूर्ण होता है। अंत में, लिपिड विविधता भी माइक्रो-डोमेन के गठन को प्रेरित करती है जो पैकिंग और आंतरिक क्रम में भिन्न होती है, जिसे लिपिड राफ्ट24,26 के रूप में जाना जाता है; ये पार्श्व विषमता प्रदर्शित करते हैं, सेलुलर सिग्नलिंग26 में महत्वपूर्ण भूमिका निभाने के लिए परिकल्पित हैं, और उनकी क्षणिक प्रकृति के कारण अध्ययन करना कठिन है।
फ्लोरोस्कोपी, स्पेक्ट्रोस्कोपी, और मॉडल झिल्ली प्रणालियों जैसे विशाल यूनिलामेलर पुटिकाओं (जीयूवी) जैसी प्रयोगात्मक तकनीकों का उपयोग झिल्ली के साथ बायोमोलेक्यूल्स की बातचीत की जांच करने के लिए किया गया है। हालांकि, इसमें शामिल घटकों की जटिल और गतिशील प्रकृति को अकेले प्रयोगात्मक तरीकों से पकड़ना मुश्किल है। उदाहरण के लिए, प्रोटीन के ट्रांसमेम्ब्रेन डोमेन की इमेजिंग, ऐसे अध्ययनों में उपयोग की जाने वाली झिल्ली की जटिलता और ब्याज की प्रक्रिया के दौरान मध्यवर्ती या क्षणिक अवस्थाओं की पहचान27,28,29 पर सीमाएं हैं। 1980 के दशक में लिपिड मोनोलेयर औरबाइलेयर के आणविक सिमुलेशन के आगमन के बाद से, लिपिड-प्रोटीन सिस्टम और उनकी बातचीत को अब आणविक स्तर पर निर्धारित किया जा सकता है। आणविक गतिशीलता (एमडी) सिमुलेशन एक सामान्य कम्प्यूटेशनल तकनीक है जो कणों के आंदोलन की भविष्यवाणी उनके इंटरमॉलिक्युलर बलों के आधार पर करती है। एक योजक इंटरैक्शन क्षमता सिस्टम30 के कणों के बीच बंधुआ और गैर-बंधुआ बातचीत का वर्णन करती है। इन इंटरैक्शन को मॉडल करने के लिए उपयोग किए जाने वाले मापदंडों के सेट को सिमुलेशन फोर्सफील्ड (एफएफ) कहा जाता है। इन मापदंडों को प्रारंभिक गणनाओं, अर्ध-अनुभवजन्य और क्वांटम यांत्रिक गणनाओं से प्राप्त किया जाता है, और एक्स-रे और इलेक्ट्रॉन विवर्तन प्रयोगों, एनएमआर, इन्फ्रारेड, रमन और न्यूट्रॉन स्पेक्ट्रोस्कोपी से डेटा को पुन: पेश करने के लिए अनुकूलित किया जाताहै।
एमडी सिमुलेशन का उपयोग संकल्प32,33,34 के विभिन्न स्तरों पर सिस्टम का अध्ययन करने के लिए किया जा सकता है। सिस्टम जो विशिष्ट बायोमोलेक्यूलर इंटरैक्शन, हाइड्रोजन बॉन्ड और अन्य उच्च-रिज़ॉल्यूशन विवरणों को चिह्नित करने का लक्ष्य रखते हैं, उनका अध्ययन ऑल-एटम (एए) सिमुलेशन के साथ किया जाता है। इसके विपरीत, मोटे-दाने वाले (सीजी) सिमुलेशन कम्प्यूटेशनल लागत को कम करने और बड़े पैमाने पर गतिशीलता33 की जांच करने के लिए बड़े कार्यात्मक समूहों में गांठ परमाणुओं को जोड़ते हैं। इन दोनों के बीच में स्थित हैं संयुक्त-परमाणु (यूए) सिमुलेशन, जहां हाइड्रोजन परमाणुओं को उनके संबंधित भारी परमाणुओं के साथ जोड़ा जाता है ताकि गणना33,35 में तेजी लाई जा सके। एमडी सिमुलेशन लिपिड झिल्ली की गतिशीलता और अन्य अणुओं के साथ उनकी बातचीत की खोज के लिए एक शक्तिशाली उपकरण हैं और झिल्ली इंटरफ़ेस पर रुचि की प्रक्रियाओं के लिए आणविक स्तर के तंत्र प्रदान करने के लिए काम कर सकते हैं। इसके अतिरिक्त, एमडी सिमुलेशन प्रयोगात्मक लक्ष्यों को कम करने और सूक्ष्म इंटरैक्शन के आधार पर किसी दिए गए सिस्टम के मैक्रोमोलेक्यूलर गुणों की भविष्यवाणी करने के लिए काम कर सकते हैं।
संक्षेप में, प्रारंभिक निर्देशांक, वेग और निरंतर तापमान और दबाव जैसी स्थितियों के एक सेट को देखते हुए, प्रत्येक कण की स्थिति और वेग की गणना इंटरैक्शन पोटेंशियल और न्यूटन के गति के नियम के संख्यात्मक एकीकरण के माध्यम से की जाती है। यह पुनरावृत्ति रूप से दोहराया जाता है, जिससे सिमुलेशन प्रक्षेपवक्र30 उत्पन्न होता है। ये गणना एमडी इंजन के साथ की जाती है; कई ओपन-सोर्स पैकेजों के बीच, GROMACS36 सबसे अधिक इस्तेमाल किए जाने वाले इंजनों में से एक है और जिसे हम यहां वर्णित करते हैं। इसमें विश्लेषण और सिस्टम के प्रारंभिक निर्देशांक के निर्माण के लिए उपकरण भी शामिल हैं जिन्हें सिम्युलेटेडकिया जा सकता है। अन्य एमडी इंजनों में एनएएमडी38 शामिल हैं; चार्मम39, और एम्बर40, जिसे उपयोगकर्ता किसी दिए गए सिस्टम के कम्प्यूटेशनल प्रदर्शन के आधार पर अपने विवेक पर चुन सकता है। सिमुलेशन के दौरान प्रक्षेपपथ की कल्पना करना और साथ ही परिणामों के विश्लेषण और व्याख्या के लिए महत्वपूर्ण है। विभिन्न प्रकार के उपकरण उपलब्ध हैं; यहां हम दृश्य आणविक गतिशीलता (वीएमडी) पर चर्चा करते हैं जो सुविधाओं की एक विस्तृत श्रृंखला प्रदान करता है, जिसमें विस्तृत ड्राइंग और रंग विधियों के साथ तीन आयामी (3-डी) विज़ुअलाइज़ेशन, वॉल्यूमेट्रिक डेटा विज़ुअलाइज़ेशन, एमडी सिमुलेशन सिस्टम के निर्माण, तैयारी और विश्लेषण और सिस्टम आकार पर कोई सीमा नहीं होने के साथ प्रक्षेपवक्र-फिल्म बनाना शामिल है, अगर मेमोरी41,42,43 उपलब्ध है।
सिस्टम घटकों के बीच अनुमानित गतिशीलता की सटीकता सीधे प्रक्षेपवक्र के प्रसार के लिए चुने गए एफएफ से प्रभावित होती है। अनुभवजन्य एफएफ परमेट्राइजेशन प्रयासों को कुछ शोध समूहों द्वारा आगे बढ़ाया जाता है। एमडी के लिए सबसे स्थापित और आम एफएफ में चार्मम39, एम्बर 40, मार्टिनी 44, ओपीएलएस45 और एसआईआरएएच 46 शामिल हैं। ऑल-एटम एडिटिव CHARMM36 (सी 36) बल क्षेत्र47 का व्यापक रूप से झिल्ली प्रणालियों के एए एमडी के लिए उपयोग किया जाता है क्योंकि यह प्रयोगात्मक संरचनात्मक डेटा को सटीक रूप से पुन: पेश करता है। यह मूल रूप से चार्मम समुदाय द्वारा विकसित किया गया था, और यह ग्रोमाक्स और एनएएमडी जैसे कई एमडी इंजनों के साथ संगत है। सामान्य एफएफ में सुधार के बावजूद, पैरामीटर सेटों में सुधार करने के लिए निरंतर प्रयास किया जाता है ताकि उन भविष्यवाणियों की अनुमति मिल सके जो प्रयोगात्मक अवलोकनों को बारीकी से पुन: पेश करते हैं, जो अध्ययनकी विशेष प्रणालियों में रुचियों से प्रेरित हैं।
लिपिड झिल्ली का अनुकरण करते समय एक चुनौती सिमुलेशन प्रक्षेपवक्र की लंबाई निर्धारित कर रही है। यह काफी हद तक विश्लेषण किए जाने वाले मैट्रिक्स और उस प्रक्रिया पर निर्भर करता है जिसे कोई चिह्नित करना चाहता है। आमतौर पर, जटिल लिपिड मिश्रण को संतुलन तक पहुंचने के लिए अधिक समय की आवश्यकता होती है, क्योंकि अधिक प्रजातियों को झिल्ली तल पर फैलने और एक स्थिर पार्श्व संगठन तक पहुंचने के लिए पर्याप्त समय होना चाहिए। एक सिमुलेशन को संतुलन में कहा जाता है जब ब्याज की संपत्ति एक पठार पर पहुंच जाती है और एक स्थिर मूल्य के बारे में उतार-चढ़ाव करती है। रुचि के गुणों और अंतःक्रियाओं पर उचित सांख्यिकीय विश्लेषण करने के लिए कम से कम 100-200 एनएस समतुल्य प्रक्षेपवक्र प्राप्त करना आम बात है। लिपिड मिश्रण और शोध प्रश्न की जटिलता के आधार पर 200-500 एनएस के बीच झिल्ली-केवल सिमुलेशन चलाना आम है। प्रोटीन-लिपिड इंटरैक्शन को आमतौर पर 500-2000 एनएस के बीच लंबे सिमुलेशन समय की आवश्यकता होती है। झिल्ली प्रणालियों के साथ नमूनाकरण और अवलोकन योग्य गतिशीलता में तेजी लाने के लिए कुछ दृष्टिकोण हैं: (i) अत्यधिक मोबाइल झिल्ली मिमेटिक (एचएमएमएम) मॉडल, जो नमूना करण में तेजी लाने के लिए कार्बनिक विलायक के साथ झिल्लीमें लिपिड के अंत कार्बन को प्रतिस्थापित करता है; और (ii) हाइड्रोजन द्रव्यमान पुनर्विभाजन (एचएमआर), जो हाइड्रोजन परमाणुओं के साथ एक प्रणाली के भीतर भारी परमाणुओं के द्रव्यमान के एक अंश को जोड़ता है ताकि एक बड़े सिमुलेशन टाइमस्टेप51 के उपयोग की अनुमति मिल सके।
निम्नलिखित प्रोटोकॉल एए एमडी का उपयोग करके यथार्थवादी झिल्ली मॉडल बनाने, चलाने और विश्लेषण करने के लिए एक शुरुआती-अनुकूल दृष्टिकोण पर चर्चा करता है। एमडी सिमुलेशन की प्रकृति को देखते हुए, परिणामों की प्रजनन क्षमता और उचित सांख्यिकीय विश्लेषण के लिए कई प्रक्षेपपथ चलाए जाने चाहिए। यह वर्तमान प्रथा है कि रुचि की प्रति प्रणाली में कम से कम तीन प्रतिकृतियां चलाई जाती हैं। एक बार जब जीव और रुचि की प्रक्रिया के लिए लिपिड प्रजातियों का चयन किया जाता है, तो झिल्ली-केवल प्रणाली के सिमुलेशन प्रक्षेपवक्र के निर्माण, चलाने और विश्लेषण करने के लिए बुनियादी चरणों को चित्र 2 में रेखांकित और सारांशित किया जाता है।
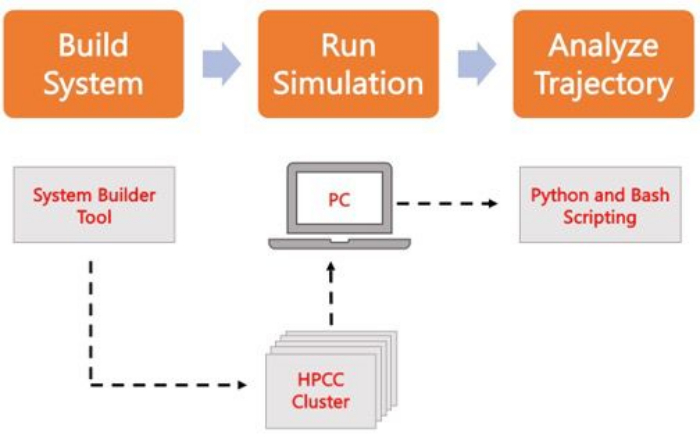
चित्र 2. एमडी सिमुलेशन चलाने के लिए योजनाबद्ध। नारंगी बक्से प्रोटोकॉल में वर्णित तीन मुख्य चरणों के अनुरूप हैं। नीचे सिमुलेशन प्रक्रिया का वर्कफ़्लो है। सिस्टम सेटअप के दौरान, सॉल्वेटेड मेम्ब्रेन सिस्टम के प्रारंभिक निर्देशांक वाले सिस्टम को चार्म-जीयूआई मेम्ब्रेन बिल्डर जैसे सिस्टम इनपुट जनरेटर के साथ बनाया जाता है। इनपुट फ़ाइलों को उच्च-प्रदर्शन कंप्यूटिंग क्लस्टर में स्थानांतरित करने के बाद, सिमुलेशन प्रक्षेपवक्र को एमडी इंजन का उपयोग करके प्रचारित किया जाता है, जैसे कि GROMACS। प्रक्षेपवक्र विश्लेषण कंप्यूटर क्लस्टर या विज़ुअलाइज़ेशन के साथ एक स्थानीय कार्य स्टेशन पर किया जा सकता है। विश्लेषण तब किया जाता है, या तो अंतर्निहित विश्लेषण कोड जैसे GROMACS और VMD के साथ पैकेज का उपयोग करके, या बैश स्क्रिप्ट या विभिन्न पायथन पुस्तकालयों का उपयोग करके। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
Protocol
Representative Results
Discussion
प्रायोगिक तकनीक क्रायो-इलेक्ट्रॉन माइक्रोस्कोपी (क्रायो-ईएम) 58, फ्लोरेसेंस तकनीक और परमाणु बल माइक्रोस्कोपी (एएफएम) 59 का उपयोग करके उच्च रिज़ॉल्यूशन पर बायोमोलेक्यूल्स की कल्पना कर…
Disclosures
The authors have nothing to disclose.
Acknowledgements
लेखक ों ने इस पांडुलिपि के लेखन के दौरान अपने सिमुलेशन प्रक्षेपवक्र और चर्चाओं के लिए जिनहुई ली और रिकार्डो एक्स रामिरेज़ को धन्यवाद दिया। ओ.सी. को बफ़ेलो प्रेसिडेंशियल फैलोशिप विश्वविद्यालय और नेशनल इंस्टीट्यूट ऑफ हेल्थ की पहल फॉर मैक्सिममाइज़िंग स्टूडेंट डेवलपमेंट ट्रेनिंग ग्रांट 1T32GM144920-01 द्वारा मार्गरीटा एल डुबोकोविच (पीआई) को सम्मानित किया गया था।
Materials
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
References
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M., Edward, H. .. . E. g. e. l. m. a. n. . Comprehensive Biophysics. , 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis, , Martin Raff, , Keith Roberts, , Peter Walter, . Molecular Biology of the Cell. , (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, &. #. 2. 2. 0. ;., Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Douglas G, H. a. y. e. s., Daniel, K. Y., Solaiman, D., Richard, . Biobased Surfactants (Second Edition) eds. , 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41 (2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264 (2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. 생화학. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5 (2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897 (2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167 (2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441 (2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S., Scott Shell, M. . Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. , 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational ‘microscopy’ of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901 (2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B., Otto Geiger, . Biogenesis of Fatty Acids, Lipids and Membranes. , 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068 (2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130 (2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. . Using VMD. , (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. . CCR Facility Description. , (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841 (2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38 (2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- . . Hepatitis C. , (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957 (2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. 생화학. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. o. b. e. r. t. . T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621 (2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).

